Document not found (404)
+This URL is invalid, sorry. Please use the navigation bar or search to continue.
+ +This URL is invalid, sorry. Please use the navigation bar or search to continue.
+ +++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Predict the 3D structure of a protein from its amino acid sequence using a simplified version of DeepMind’s AlphaFold2 originally released and benchmarked for AlphaFold Colab.
+Returns: Predicted structure (PDB) and alignment error (json).
Before using gget alphafold for the first time:
conda install -qy conda==4.13.0 && conda install -qy -c conda-forge openmm=7.5.1openmm=7.7.0 for Python >= 3.10)conda update -qy conda to update conda to the latest version afterwards.gget setup alphafold / gget.setup("alphafold") once (also see gget setup). Running gget setup alphafold / gget.setup("alphafold") will download and install the latest version of AlphaFold2 hosted on the AlphaFold GitHub Repo. You can rerun this command any time to update the software after a new AlphaFold release.Positional argument
+sequence
+Amino acid sequence (str), or list of sequences (gget alphafold will automatically use the multimer model if multiple sequences are passed), or path to FASTA file.
Optional arguments
+-mr --multimer_recycles
+The multimer model will continue recycling until the predictions stop changing, up to the limit set here. Default: 3.
+For higher accuracy, at the potential cost of longer inference times, set this to 20.
-o --out
+Path to folder to save prediction results in (str). Default: "./[date_time]_gget_alphafold_prediction".
Flags
+-mfm --multimer_for_monomer
+Use multimer model for a monomer.
-r --relax
+AMBER relax the best model.
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
plot
+Python only. plot=True provides an interactive, 3D graphical overview of the predicted structure and alignment quality using py3Dmol and matplotlib (default: True).
show_sidechains
+Python only. show_sidechains=True includes side chains in the plot (default: True).
# Generate new prediction from amino acid sequence
+gget alphafold MAAHKGAEHHHKAAEHHEQAAKHHHAAAEHHEKGEHEQAAHHADTAYAHHKHAEEHAAQAAKHDAEHHAPKPH
+
+# Find similar sequences deposited on the PDB for comparative analysis
+gget blast --database pdbaa MAAHKGAEHHHKAAEHHEQAAKHHHAAAEHHEKGEHEQAAHHADTAYAHHKHAEEHAAQAAKHDAEHHAPKPH
+
+# Fetch the PDB files of similar structures returned by gget blast for comparison, to get a measure for model quality
+gget pdb 3UQ3 -o 3UQ3.pdb
+gget pdb 2K42 -o 2K42.pdb
+# Python
+gget.alphafold("MAAHKGAEHHHKAAEHHEQAAKHHHAAAEHHEKGEHEQAAHHADTAYAHHKHAEEHAAQAAKHDAEHHAPKPH")
+
+# Find similar sequences deposited on the PDB for comparative analysis
+gget.blast("MAAHKGAEHHHKAAEHHEQAAKHHHAAAEHHEKGEHEQAAHHADTAYAHHKHAEEHAAQAAKHDAEHHAPKPH", database="pdbaa")
+
+# Fetch the PDB files of similar structures returned by gget blast for comparison, to get a measure for model quality
+gget.pdb("3UQ3", save=True)
+gget.pdb("2K42", save=True)
+→ gget alphafold returns the predicted structure (PDB) and predicted alignment error (.json) in a new folder ("./[date_time]_gget_alphafold_prediction"). The use case above exemplifies how to use gget blast and gget pdb for a comparative analysis of the new prediction. PDB files can be viewed interactively in 3D online, or using programs like PyMOL or Blender. To compare two PDB files, you can use this website. The Python interface also returns interactive plots, which can be generated from the PDB and JSON as described in the gget alphafold FAQ Q4.
If you use gget alphafold in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Jumper, J., Evans, R., Pritzel, A. et al. Highly accurate protein structure prediction with AlphaFold. Nature 596, 583–589 (2021). https://doi.org/10.1038/s41586-021-03819-2
+And, if applicable:
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Find the most correlated genes to a gene of interest or find the gene's tissue expression atlas using ARCHS4.
+Return format: JSON (command-line) or data frame/CSV (Python).
Positional argument
+gene
+Short name (gene symbol) of gene of interest, e.g. STAT4.
+Alternatively: use flag --ensembl to input an Ensembl gene IDs, e.g. ENSG00000138378.
Optional arguments
+-w --which
+'correlation' (default) or 'tissue'.
+'correlation' returns a gene correlation table that contains the 100 most correlated genes to the gene of interest. The Pearson correlation is calculated over all samples and tissues in ARCHS4.
+'tissue' returns a tissue expression atlas calculated from human or mouse samples (as defined by 'species') in ARCHS4.
-s --species
+'human' (default) or 'mouse'.
+Defines whether to use human or mouse samples from ARCHS4.
+(Only for tissue expression atlas.)
-o --out
+Path to the file the results will be saved in, e.g. path/to/directory/results.csv (or .json). Default: Standard out.
+Python: save=True will save the output in the current working directory.
Flags
+-e --ensembl
+Add this flag if gene is given as an Ensembl gene ID.
-csv --csv
+Command-line only. Returns results in CSV format.
+Python: Use json=True to return output in JSON format.
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
gget archs4 ACE2
+# Python
+gget.archs4("ACE2")
+→ Returns the 100 most correlated genes to ACE2:
+| gene_symbol | pearson_correlation |
|---|---|
| SLC5A1 | 0.579634 |
| CYP2C18 | 0.576577 |
| . . . | . . . |
gget archs4 -w tissue ACE2
+# Python
+gget.archs4("ACE2", which="tissue")
+→ Returns the tissue expression of ACE2 (by default, human data is used):
+| id | min | q1 | median | q3 | max |
|---|---|---|---|---|---|
| System.Urogenital/Reproductive System.Kidney.RENAL CORTEX | 0.113644 | 8.274060 | 9.695840 | 10.51670 | 11.21970 |
| System.Digestive System.Intestine.INTESTINAL EPITHELIAL CELL | 0.113644 | 5.905560 | 9.570450 | 13.26470 | 13.83590 |
| . . . | . . . | . . . | . . . | . . . | . . . |
+Check out this tutorial by Dave Tang who wrote an R script to create this figure from the gget archs4 JSON output:
If you use gget archs4 in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Lachmann A, Torre D, Keenan AB, Jagodnik KM, Lee HJ, Wang L, Silverstein MC, Ma’ayan A. Massive mining of publicly available RNA-seq data from human and mouse. Nature Communications 9. Article number: 1366 (2018), doi:10.1038/s41467-018-03751-6
+Bray NL, Pimentel H, Melsted P and Pachter L, Near optimal probabilistic RNA-seq quantification, Nature Biotechnology 34, p 525--527 (2016). https://doi.org/10.1038/nbt.3519
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Fetch orthology and gene expression data from Bgee using Ensembl IDs.
+Return format: JSON/CSV (command-line) or data frame (Python).
++If you are specifically interested in human gene expression data, consider using gget opentargets or gget archs4 instead. +gget bgee has less data, but supports more species.
+
This module was written by Sam Wagenaar.
+Positional argument
+ens_id
+Ensembl gene ID, e.g. ENSG00000169194 or ENSSSCG00000014725.
Required arguments
+-t --type
+Type of data to fetch. Options: orthologs, expression.
Optional arguments
+-o --out
+Path to the JSON file the results will be saved in, e.g. path/to/directory/results.json. Default: Standard out.
Flags
+-csv --csv
+Command-line only. Returns the output in CSV format, instead of JSON format.
+Python: Use json=True to return output in JSON format.
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
Get orthologs for a gene
+gget bgee ENSSSCG00000014725 -t orthologs
+import gget
+gget.bgee("ENSSSCG00000014725", type="orthologs")
+→ Returns orthologs for the gene with Ensembl ID ENSSSCG00000014725.
+| gene_id | gene_name | species_id | genus | species |
|---|---|---|---|---|
| 734881 | hbb1 | 8355 | Xenopus | laevis |
| ENSFCAG00000038029 | LOC101098159 | 9685 | Felis | catus |
| ENSBTAG00000047356 | LOC107131172 | 9913 | Bos | taurus |
| ENSOARG00000019163 | LOC101105437 | 9940 | Ovis | aries |
| ENSXETG00000025667 | hbg1 | 8364 | Xenopus | tropicalis |
| ... | ... | ... | ... | ... |
Get gene expression data for a gene
+gget bgee ENSSSCG00000014725 -t expression
+import gget
+gget.bgee("ENSSSCG00000014725", type="expression")
+→ Returns gene expression data for the gene with Ensembl ID ENSSSCG00000014725.
+| anat_entity_id | anat_entity_name | score | score_confidence | expression_state |
|---|---|---|---|---|
| UBERON:0000178 | blood | 99.98 | high | expressed |
| UBERON:0002106 | spleen | 99.96 | high | expressed |
| UBERON:0002190 | subcutaneous adipose tissue | 99.70 | high | expressed |
| UBERON:0005316 | endocardial endothelium | 99.61 | high | expressed |
| UBERON:0002107 | liver | 99.27 | high | expressed |
| ... | ... | ... | ... | ... |
If you use gget bgee in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Frederic B Bastian, Julien Roux, Anne Niknejad, Aurélie Comte, Sara S Fonseca Costa, Tarcisio Mendes de Farias, Sébastien Moretti, Gilles Parmentier, Valentine Rech de Laval, Marta Rosikiewicz, Julien Wollbrett, Amina Echchiki, Angélique Escoriza, Walid H Gharib, Mar Gonzales-Porta, Yohan Jarosz, Balazs Laurenczy, Philippe Moret, Emilie Person, Patrick Roelli, Komal Sanjeev, Mathieu Seppey, Marc Robinson-Rechavi (2021). The Bgee suite: integrated curated expression atlas and comparative transcriptomics in animals. Nucleic Acids Research, Volume 49, Issue D1, 8 January 2021, Pages D831–D847, https://doi.org/10.1093/nar/gkaa793
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
BLAST a nucleotide or amino acid sequence to any BLAST database.
+Return format: JSON (command-line) or data frame/CSV (Python).
Positional argument
+sequence
+Nucleotide or amino acid sequence, or path to FASTA or .txt file.
Optional arguments
+-p --program
+'blastn', 'blastp', 'blastx', 'tblastn', or 'tblastx'.
+Default: 'blastn' for nucleotide sequences; 'blastp' for amino acid sequences.
-db --database
+'nt', 'nr', 'refseq_rna', 'refseq_protein', 'swissprot', 'pdbaa', or 'pdbnt'.
+Default: 'nt' for nucleotide sequences; 'nr' for amino acid sequences.
+More info on BLAST databases
-l --limit
+Limits number of hits to return. Default: 50.
-e --expect
+Defines the expect value cutoff. Default: 10.0.
-o --out
+Path to the file the results will be saved in, e.g. path/to/directory/results.csv (or .json). Default: Standard out.
+Python: save=True will save the output in the current working directory.
Flags
+-lcf --low_comp_filt
+Turns on low complexity filter.
-mbo --megablast_off
+Turns off MegaBLAST algorithm. Default: MegaBLAST on (blastn only).
-csv --csv
+Command-line only. Returns results in CSV format.
+Python: Use json=True to return output in JSON format.
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
wrap_text
+Python only. wrap_text=True displays data frame with wrapped text for easy reading (default: False).
gget blast MKWMFKEDHSLEHRCVESAKIRAKYPDRVPVIVEKVSGSQIVDIDKRKYLVPSDITVAQFMWIIRKRIQLPSEKAIFLFVDKTVPQSR
+# Python
+gget.blast("MKWMFKEDHSLEHRCVESAKIRAKYPDRVPVIVEKVSGSQIVDIDKRKYLVPSDITVAQFMWIIRKRIQLPSEKAIFLFVDKTVPQSR")
+→ Returns the BLAST result of the sequence of interest. gget blast automatically detects this sequence as an amino acid sequence and therefore sets the BLAST program to blastp with database nr.
| Description | Scientific Name | Common Name | Taxid | Max Score | Total Score | Query Cover | ... |
|---|---|---|---|---|---|---|---|
| PREDICTED: gamma-aminobutyric acid receptor-as... | Colobus angolensis palliatus | NaN | 336983 | 180 | 180 | 100% | ... |
| . . . | . . . | . . . | . . . | . . . | . . . | . . . | ... |
+BLAST from .fa or .txt file:
gget blast fasta.fa
+# Python
+gget.blast("fasta.fa")
+→ Returns the BLAST results of the first sequence contained in the fasta.fa file.
+If you use gget blast in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ. Basic local alignment search tool. J Mol Biol. 1990 Oct 5;215(3):403-10. doi: 10.1016/S0022-2836(05)80360-2. PMID: 2231712.
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Find the genomic location of a nucleotide or amino acid sequence using BLAT.
+Return format: JSON (command-line) or data frame/CSV (Python).
Positional argument
+sequence
+Nucleotide or amino acid sequence, or path to FASTA or .txt file.
Optional arguments
+-st --seqtype
+'DNA', 'protein', 'translated%20RNA', or 'translated%20DNA'.
+Default: 'DNA' for nucleotide sequences; 'protein' for amino acid sequences.
-a --assembly
+'human' (hg38) (default), 'mouse' (mm39), 'zebrafinch' (taeGut2),
+or any of the species assemblies available here (use short assembly name).
-o --out
+Path to the file the results will be saved in, e.g. path/to/directory/results.csv (or .json). Default: Standard out.
+Python: save=True will save the output in the current working directory.
Flags
+-csv --csv
+Command-line only. Returns results in CSV format.
+Python: Use json=True to return output in JSON format.
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
gget blat -a taeGut2 MKWMFKEDHSLEHRCVESAKIRAKYPDRVPVIVEKVSGSQIVDIDKRKYLVPSDITVAQFMWIIRKRIQLPSEKAIFLFVDKTVPQSR
+# Python
+gget.blat("MKWMFKEDHSLEHRCVESAKIRAKYPDRVPVIVEKVSGSQIVDIDKRKYLVPSDITVAQFMWIIRKRIQLPSEKAIFLFVDKTVPQSR", assembly="taeGut2")
+→ Returns BLAT results for assembly taeGut2 (zebra finch). In the above example, gget blat automatically detects this sequence as an amino acid sequence and therefore sets the BLAT seqtype to protein.
| genome | query_size | aligned_start | aligned_end | matches | mismatches | %_aligned | ... |
|---|---|---|---|---|---|---|---|
| taeGut2 | 88 | 12 | 88 | 77 | 0 | 87.5 | ... |
If you use gget blat in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Kent WJ. BLAT--the BLAST-like alignment tool. Genome Res. 2002 Apr;12(4):656-64. doi: 10.1101/gr.229202. PMID: 11932250; PMCID: PMC187518.
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Plot cancer genomics heatmaps using data from cBioPortal using Ensembl IDs or gene names.
+This module was written by Sam Wagenaar.
+Positional argument
+subcommand
+Either search or plot
search subcommand (Python: gget.cbio_search)Find cBioPortal study IDs by keyword.
+Return format: JSON (command-line) or string list (Python).
+Note: This does not return studies with mixed cancer types.
Positional argument
+keywords
+Space-separated list of keywords to search for, e.g. breast lung.
+Python: Pass keywords as a list of strings.
plot subcommand (Python: gget.cbio_plot)Plot cancer genomics heatmaps using data from cBioPortal. +Return format: PNG (command-line and Python)
+Required arguments
+-s --study_ids
+Space-separated list of cBioPortal study IDs, e.g. msk_impact_2017 egc_msk_2023.
-g --genes
+Space-separated list of gene names or Ensembl IDs, e.g. NOTCH3 ENSG00000108375.
Optional arguments
+-st --stratification
+Column to stratify the data by. Default: tissue.
+Options:
-vt --variation_type
+Type of variation to plot. Default: mutation_occurrences.
+Options:
stratification must be 'sample' for this option)stratification must be 'sample' for this option)-f --filter
+Filter the data by a specific value in a specific column, e.g. study_id:msk_impact_2017
+Python: filter=(column, value)
-dd --data_dir
+Directory to store data files. Default: ./gget_cbio_cache.
-fd --figure_dir
+Directory to output figures. Default: ./gget_cbio_figures.
-fn --filename
+Filename for the output figure, relative to figure_dir. Default: auto-generated
+Python: figure_filename
-t --title
+Title for the output figure. Default: auto-generated
+Python: figure_title
-dpi --dpi
+DPI of the output figure. Default: 100.
Flags
+-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
-nc --no_confirm
+Command-line only. Skip download confirmation prompts.
+Python: Use confirm_download=True to enable download confirmation prompts.
-sh --show
+Show the plot in a window (automatic in Jupyter notebooks).
Find all cBioPortal studies with cancer types matching specific keywords:
+gget cbio search esophag ovary ovarian
+# Python
+import gget
+gget.cbio_search(['esophag', 'ovary', 'ovarian'])
+→ Returns a list of studies with cancer types matching the keywords esophag, ovary, or ovarian.
['egc_tmucih_2015', 'egc_msk_2017', ..., 'msk_spectrum_tme_2022']
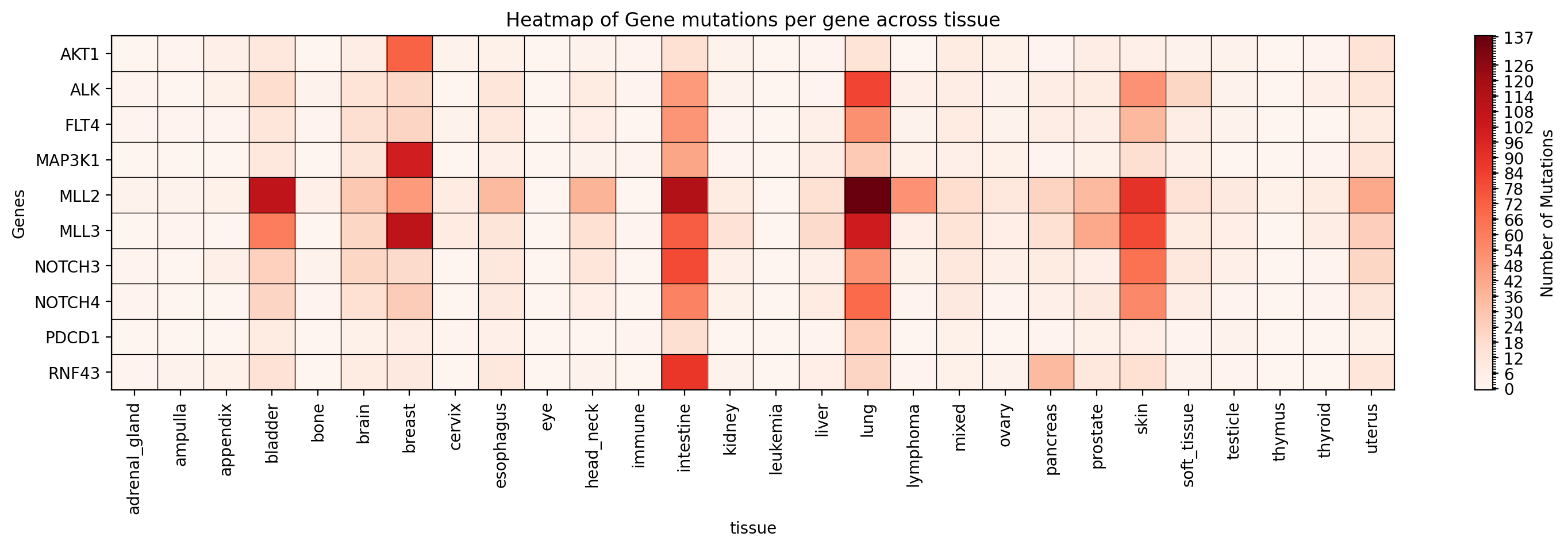
+Plot a heatmap of mutation occurrences for specific genes in a specific study:
+gget cbio plot \
+ -s msk_impact_2017 \
+ -g AKT1 ALK FLT4 MAP3K1 MLL2 MLL3 NOTCH3 NOTCH4 PDCD1 RNF43 \
+ -st tissue \
+ -vt mutation_occurrences \
+ -dpi 200 \
+ -y
+# Python
+import gget
+gget.cbio_plot(
+ ['msk_impact_2017'],
+ ['AKT1', 'ALK', 'FLT4', 'MAP3K1', 'MLL2', 'MLL3', 'NOTCH3', 'NOTCH4', 'PDCD1', 'RNF43'],
+ stratification='tissue',
+ variation_type='mutation_occurrences',
+ dpi=200
+)
+→ Saves a heatmap of mutation occurrences for the specified genes in the specified study to ./gget_cbio_figures/Heatmap_tissue.png.
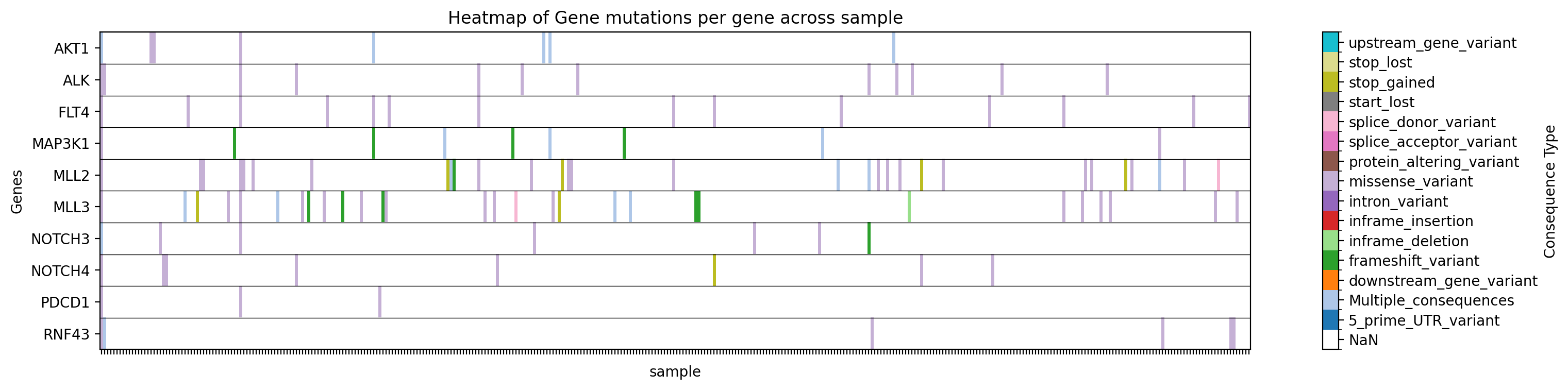
Plot a heatmap of mutation types for specific genes in a specific study:
+gget cbio plot \
+ -s msk_impact_2017 \
+ -g AKT1 ALK FLT4 MAP3K1 MLL2 MLL3 NOTCH3 NOTCH4 PDCD1 RNF43 \
+ -st sample \
+ -vt Consequence \
+ -dpi 200 \
+ -y
+# Python
+import gget
+gget.cbio_plot(
+ ['msk_impact_2017'],
+ ['AKT1', 'ALK', 'FLT4', 'MAP3K1', 'MLL2', 'MLL3', 'NOTCH3', 'NOTCH4', 'PDCD1', 'RNF43'],
+ stratification='sample',
+ variation_type='Consequence',
+ dpi=200,
+)
+→ Saves a heatmap of mutation types for the specified genes in the specified study to ./gget_cbio_figures/Heatmap_sample.png.
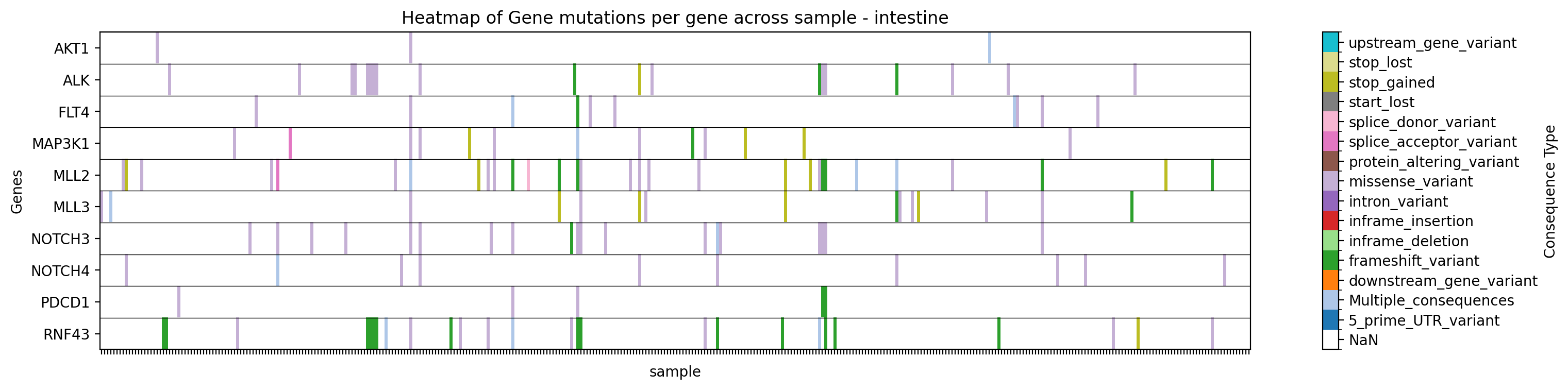
Plot a heatmap of mutation types for specific genes in a specific study, filtering by tissue:
+gget cbio plot \
+ -s msk_impact_2017 \
+ -g AKT1 ALK FLT4 MAP3K1 MLL2 MLL3 NOTCH3 NOTCH4 PDCD1 RNF43 \
+ -st sample \
+ -vt Consequence \
+ -f tissue:intestine \
+ -dpi 200 \
+ -y
+# Python
+import gget
+gget.cbio_plot(
+ ['msk_impact_2017'],
+ ['AKT1', 'ALK', 'FLT4', 'MAP3K1', 'MLL2', 'MLL3', 'NOTCH3', 'NOTCH4', 'PDCD1', 'RNF43'],
+ stratification='sample',
+ variation_type='Consequence',
+ filter=('tissue', 'intestine'),
+ dpi=200,
+)
+→ Saves a heatmap of mutation types for the specified genes in the specified study, filtered by tissue, to ./gget_cbio_figures/Heatmap_sample_intestine.png.
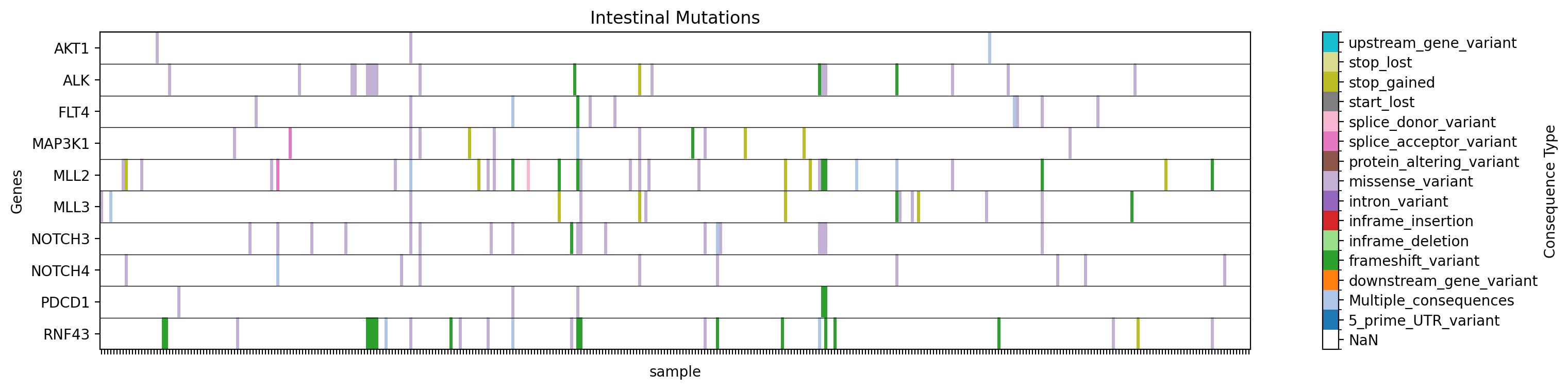
Plot a heatmap with a custom title and filename:
+gget cbio plot \
+ -s msk_impact_2017 \
+ -g AKT1 ALK FLT4 MAP3K1 MLL2 MLL3 NOTCH3 NOTCH4 PDCD1 RNF43 \
+ -st sample \
+ -vt Consequence \
+ -f tissue:intestine \
+ -dpi 200 \
+ -y \
+ -t "Intestinal Mutations" \
+ -fn intestinal_mutations.png
+# Python
+import gget
+gget.cbio_plot(
+ ['msk_impact_2017'],
+ ['AKT1', 'ALK', 'FLT4', 'MAP3K1', 'MLL2', 'MLL3', 'NOTCH3', 'NOTCH4', 'PDCD1', 'RNF43'],
+ stratification='sample',
+ variation_type='Consequence',
+ filter=('tissue', 'intestine'),
+ dpi=200,
+ figure_title='Intestinal Mutations',
+ figure_filename='intestinal_mutations.png'
+)
+→ Saves a heatmap of mutation types for the specified genes in the specified study, filtered by tissue, with the title "Intestinal Mutations" to ./gget_cbio_figures/intestinal_mutations.png.
If you use gget cbio in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Cerami E, Gao J, Dogrusoz U, Gross BE, Sumer SO, Aksoy BA, Jacobsen A, Byrne CJ, Heuer ML, Larsson E, Antipin Y, Reva B, Goldberg AP, Sander C, Schultz N. The cBio cancer genomics portal: an open platform for exploring multidimensional cancer genomics data. Cancer Discov. 2012 May;2(5):401-4. doi: 10.1158/2159-8290.CD-12-0095. Erratum in: Cancer Discov. 2012 Oct;2(10):960. PMID: 22588877; PMCID: PMC3956037.
+Gao J, Aksoy BA, Dogrusoz U, Dresdner G, Gross B, Sumer SO, Sun Y, Jacobsen A, Sinha R, Larsson E, Cerami E, Sander C, Schultz N. Integrative analysis of complex cancer genomics and clinical profiles using the cBioPortal. Sci Signal. 2013 Apr 2;6(269):pl1. doi: 10.1126/scisignal.2004088. PMID: 23550210; PMCID: PMC4160307.
+de Bruijn I, Kundra R, Mastrogiacomo B, Tran TN, Sikina L, Mazor T, Li X, Ochoa A, Zhao G, Lai B, Abeshouse A, Baiceanu D, Ciftci E, Dogrusoz U, Dufilie A, Erkoc Z, Garcia Lara E, Fu Z, Gross B, Haynes C, Heath A, Higgins D, Jagannathan P, Kalletla K, Kumari P, Lindsay J, Lisman A, Leenknegt B, Lukasse P, Madela D, Madupuri R, van Nierop P, Plantalech O, Quach J, Resnick AC, Rodenburg SYA, Satravada BA, Schaeffer F, Sheridan R, Singh J, Sirohi R, Sumer SO, van Hagen S, Wang A, Wilson M, Zhang H, Zhu K, Rusk N, Brown S, Lavery JA, Panageas KS, Rudolph JE, LeNoue-Newton ML, Warner JL, Guo X, Hunter-Zinck H, Yu TV, Pilai S, Nichols C, Gardos SM, Philip J; AACR Project GENIE BPC Core Team, AACR Project GENIE Consortium; Kehl KL, Riely GJ, Schrag D, Lee J, Fiandalo MV, Sweeney SM, Pugh TJ, Sander C, Cerami E, Gao J, Schultz N. Analysis and Visualization of Longitudinal Genomic and Clinical Data from the AACR Project GENIE Biopharma Collaborative in cBioPortal. Cancer Res. 2023 Dec 1;83(23):3861-3867. doi: 10.1158/0008-5472.CAN-23-0816. PMID: 37668528; PMCID: PMC10690089.
+Please also cite the source of the data if you are using a publicly available dataset.
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Query data from CZ CELLxGENE Discover using the CZ CELLxGENE Discover Census. CZ CELLxGENE Discover provides ready-to-use single-cell RNA sequencing count matrices for certain tissues/diseases/genes/etc.
+Returns: An AnnData object containing the count matrix and metadata of single-cell RNA sequencing data from the defined tissues/genes/etc.
+Before using gget cellxgene for the first time, run gget setup cellxgene / gget.setup("cellxgene") once (also see gget setup).
Optional arguments
+-s --species
+Choice of 'homo_sapiens' or 'mus_musculus'. Default: 'homo_sapiens'.
-g --gene
+Str or list of gene name(s) or Ensembl ID(s). Default: None.
+NOTE: Use -e / --ensembl (Python: ensembl=True) when providing Ensembl ID(s) instead of gene name(s).
+See https://cellxgene.cziscience.com/gene-expression for examples of available genes.
-cv --census_version
+Str defining version of Census, e.g. "2023-05-15", or "latest" or "stable". Default: "stable".
-cn --column_names
+List of metadata columns to return (stored in AnnData.obs).
+Default: ['dataset_id', 'assay', 'suspension_type', 'sex', 'tissue_general', 'tissue', 'cell_type']
+For more options, see: https://api.cellxgene.cziscience.com/curation/ui/#/ -> Schemas -> dataset
-o --out
+Path to file to save generated AnnData .h5ad file (or .csv with -mo / --meta_only).
+Required when using from command line!
Flags
+-e --ensembl
+Use when genes are provided as Ensembl IDs instead of gene names.
-mo --meta_only
+Only returns metadata data frame (corresponds to AnnData.obs).
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
Optional arguments corresponding to CZ CELLxGENE Discover metadata attributes
+--tissue
+Str or list of tissue(s), e.g. ['lung', 'blood']. Default: None.
+See https://cellxgene.cziscience.com/gene-expression for examples of available tissues.
--cell_type
+Str or list of cell type (s), e.g. ['mucus secreting cell', 'neuroendocrine cell']. Default: None.
+See https://cellxgene.cziscience.com/gene-expression and select a tissue to see examples of available cell types.
--development_stage
+Str or list of development stage(s). Default: None.
--disease
+Str or list of disease(s). Default: None.
--sex
+Str or list of sex(es), e.g. 'female'. Default: None.
--dataset_id
+Str or list of CELLxGENE dataset ID(s). Default: None.
--tissue_general_ontology_term_id
+Str or list of high-level tissue UBERON ID(s). Default: None.
+Tissue labels and their corresponding UBERON IDs are listed here.
--tissue_general
+Str or list of high-level tissue label(s). Default: None.
+Tissue labels and their corresponding UBERON IDs are listed here.
--tissue_ontology_term_id
+Str or list of tissue ontology term ID(s) as defined in the CELLxGENE dataset schema. Default: None.
--assay_ontology_term_id
+Str or list of assay ontology term ID(s) as defined in the CELLxGENE dataset schema. Default: None.
--assay
+Str or list of assay(s) as defined in the CELLxGENE dataset schema. Default: None.
--cell_type_ontology_term_id
+Str or list of cell type ontology term ID(s) as defined in the CELLxGENE dataset schema. Default: None.
--development_stage_ontology_term_id
+Str or list of development stage ontology term ID(s) as defined in the CELLxGENE dataset schema. Default: None.
--disease_ontology_term_id
+Str or list of disease ontology term ID(s) as defined in the CELLxGENE dataset schema. Default: None.
--donor_id
+Str or list of donor ID(s) as defined in the CELLxGENE dataset schema. Default: None.
--self_reported_ethnicity_ontology_term_id
+Str or list of self-reported ethnicity ontology ID(s) as defined in the CELLxGENE dataset schema. Default: None.
--self_reported_ethnicity
+Str or list of self-reported ethnicity as defined in the CELLxGENE dataset schema. Default: None.
--sex_ontology_term_id
+Str or list of sex ontology ID(s) as defined in the CELLxGENE dataset schema. Default: None.
--suspension_type
+Str or list of suspension type(s) as defined in the CELLxGENE dataset schema. Default: None.
gget cellxgene --gene ACE2 ABCA1 SLC5A1 --tissue lung --cell_type 'mucus secreting cell' 'neuroendocrine cell' -o example_adata.h5ad
+# Python
+adata = gget.cellxgene(
+ gene = ["ACE2", "ABCA1", "SLC5A1"],
+ tissue = "lung",
+ cell_type = ["mucus secreting cell", "neuroendocrine cell"]
+)
+adata
+→ Returns an AnnData object containing the scRNAseq ACE2, ABCA1, and SLC5A1 count matrix of 3322 human lung mucus secreting and neuroendocrine cells from CZ CELLxGENE Discover and their corresponding metadata.
+Fetch metadata (corresponds to AnnData.obs) only:
+gget cellxgene --meta_only --gene ENSMUSG00000015405 --ensembl --tissue lung --species mus_musculus -o example_meta.csv
+# Python
+df = gget.cellxgene(
+ meta_only = True,
+ gene = "ENSMUSG00000015405",
+ ensembl = True,
+ tissue = "lung",
+ species = "mus_musculus"
+)
+df
+→ Returns only the metadata from ENSMUSG00000015405 (ACE2) expression datasets corresponding to mouse lung cells.
+Also see: https://chanzuckerberg.github.io/cellxgene-census/notebooks/api_demo/census_gget_demo.html
+If you use gget cellxgene in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Chanzuckerberg Initiative. (n.d.). CZ CELLxGENE Discover. Retrieved [insert date here], from https://cellxgene.cziscience.com/
+If you use gget in a publication, please cite:
+Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
If using gget alphafold, please also cite:
And, if applicable:
+If using gget archs4, please also cite:
Lachmann A, Torre D, Keenan AB, Jagodnik KM, Lee HJ, Wang L, Silverstein MC, Ma’ayan A. Massive mining of publicly available RNA-seq data from human and mouse. Nature Communications 9. Article number: 1366 (2018), doi:10.1038/s41467-018-03751-6
+Bray NL, Pimentel H, Melsted P and Pachter L, Near optimal probabilistic RNA-seq quantification, Nature Biotechnology 34, p 525--527 (2016). https://doi.org/10.1038/nbt.3519
+If using gget bgee, please also cite:
If using gget blast, please also cite:
If using gget blat, please also cite:
If using gget cbio, please also cite:
Cerami E, Gao J, Dogrusoz U, Gross BE, Sumer SO, Aksoy BA, Jacobsen A, Byrne CJ, Heuer ML, Larsson E, Antipin Y, Reva B, Goldberg AP, Sander C, Schultz N. The cBio cancer genomics portal: an open platform for exploring multidimensional cancer genomics data. Cancer Discov. 2012 May;2(5):401-4. doi: 10.1158/2159-8290.CD-12-0095. Erratum in: Cancer Discov. 2012 Oct;2(10):960. PMID: 22588877; PMCID: PMC3956037.
+Gao J, Aksoy BA, Dogrusoz U, Dresdner G, Gross B, Sumer SO, Sun Y, Jacobsen A, Sinha R, Larsson E, Cerami E, Sander C, Schultz N. Integrative analysis of complex cancer genomics and clinical profiles using the cBioPortal. Sci Signal. 2013 Apr 2;6(269):pl1. doi: 10.1126/scisignal.2004088. PMID: 23550210; PMCID: PMC4160307.
+de Bruijn I, Kundra R, Mastrogiacomo B, Tran TN, Sikina L, Mazor T, Li X, Ochoa A, Zhao G, Lai B, Abeshouse A, Baiceanu D, Ciftci E, Dogrusoz U, Dufilie A, Erkoc Z, Garcia Lara E, Fu Z, Gross B, Haynes C, Heath A, Higgins D, Jagannathan P, Kalletla K, Kumari P, Lindsay J, Lisman A, Leenknegt B, Lukasse P, Madela D, Madupuri R, van Nierop P, Plantalech O, Quach J, Resnick AC, Rodenburg SYA, Satravada BA, Schaeffer F, Sheridan R, Singh J, Sirohi R, Sumer SO, van Hagen S, Wang A, Wilson M, Zhang H, Zhu K, Rusk N, Brown S, Lavery JA, Panageas KS, Rudolph JE, LeNoue-Newton ML, Warner JL, Guo X, Hunter-Zinck H, Yu TV, Pilai S, Nichols C, Gardos SM, Philip J; AACR Project GENIE BPC Core Team, AACR Project GENIE Consortium; Kehl KL, Riely GJ, Schrag D, Lee J, Fiandalo MV, Sweeney SM, Pugh TJ, Sander C, Cerami E, Gao J, Schultz N. Analysis and Visualization of Longitudinal Genomic and Clinical Data from the AACR Project GENIE Biopharma Collaborative in cBioPortal. Cancer Res. 2023 Dec 1;83(23):3861-3867. doi: 10.1158/0008-5472.CAN-23-0816. PMID: 37668528; PMCID: PMC10690089.
+Please also cite the source of the data if you are using a publicly available dataset.
+If using gget cellxgene, please also cite:
If using gget cosmic, please also cite:
If using gget diamond, please also cite:
If using gget elm, please also cite:
Laura Luebbert, Chi Hoang, Manjeet Kumar, Lior Pachter, Fast and scalable querying of eukaryotic linear motifs with gget elm, Bioinformatics, 2024, btae095, https://doi.org/10.1093/bioinformatics/btae095
+Manjeet Kumar, Sushama Michael, Jesús Alvarado-Valverde, Bálint Mészáros, Hugo Sámano‐Sánchez, András Zeke, Laszlo Dobson, Tamas Lazar, Mihkel Örd, Anurag Nagpal, Nazanin Farahi, Melanie Käser, Ramya Kraleti, Norman E Davey, Rita Pancsa, Lucía B Chemes, Toby J Gibson, The Eukaryotic Linear Motif resource: 2022 release, Nucleic Acids Research, Volume 50, Issue D1, 7 January 2022, Pages D497–D508, https://doi.org/10.1093/nar/gkab975
+If using gget enrichr, please also cite:
Chen EY, Tan CM, Kou Y, Duan Q, Wang Z, Meirelles GV, Clark NR, Ma'ayan A. Enrichr: interactive and collaborative HTML5 gene list enrichment analysis tool. BMC Bioinformatics. 2013; 128(14). https://doi.org/10.1186/1471-2105-14-128
+Kuleshov MV, Jones MR, Rouillard AD, Fernandez NF, Duan Q, Wang Z, Koplev S, Jenkins SL, Jagodnik KM, Lachmann A, McDermott MG, Monteiro CD, Gundersen GW, Ma'ayan A. Enrichr: a comprehensive gene set enrichment analysis web server 2016 update. Nucleic Acids Research. 2016; gkw377. doi: 10.1093/nar/gkw377
+Xie Z, Bailey A, Kuleshov MV, Clarke DJB., Evangelista JE, Jenkins SL, Lachmann A, Wojciechowicz ML, Kropiwnicki E, Jagodnik KM, Jeon M, & Ma’ayan A. Gene set knowledge discovery with Enrichr. Current Protocols, 1, e90. 2021. doi: 10.1002/cpz1.90.
+If working with non-human/mouse datasets, please also cite:
+If using gget info, please also cite:
Martin FJ, Amode MR, Aneja A, Austine-Orimoloye O, Azov AG, Barnes I, Becker A, Bennett R, Berry A, Bhai J, Bhurji SK, Bignell A, Boddu S, Branco Lins PR, Brooks L, Ramaraju SB, Charkhchi M, Cockburn A, Da Rin Fiorretto L, Davidson C, Dodiya K, Donaldson S, El Houdaigui B, El Naboulsi T, Fatima R, Giron CG, Genez T, Ghattaoraya GS, Martinez JG, Guijarro C, Hardy M, Hollis Z, Hourlier T, Hunt T, Kay M, Kaykala V, Le T, Lemos D, Marques-Coelho D, Marugán JC, Merino GA, Mirabueno LP, Mushtaq A, Hossain SN, Ogeh DN, Sakthivel MP, Parker A, Perry M, Piližota I, Prosovetskaia I, Pérez-Silva JG, Salam AIA, Saraiva-Agostinho N, Schuilenburg H, Sheppard D, Sinha S, Sipos B, Stark W, Steed E, Sukumaran R, Sumathipala D, Suner MM, Surapaneni L, Sutinen K, Szpak M, Tricomi FF, Urbina-Gómez D, Veidenberg A, Walsh TA, Walts B, Wass E, Willhoft N, Allen J, Alvarez-Jarreta J, Chakiachvili M, Flint B, Giorgetti S, Haggerty L, Ilsley GR, Loveland JE, Moore B, Mudge JM, Tate J, Thybert D, Trevanion SJ, Winterbottom A, Frankish A, Hunt SE, Ruffier M, Cunningham F, Dyer S, Finn RD, Howe KL, Harrison PW, Yates AD, Flicek P. Ensembl 2023. Nucleic Acids Res. 2023 Jan 6;51(D1):D933-D941. doi: 10.1093/nar/gkac958. PMID: 36318249; PMCID: PMC9825606.
+Sayers EW, Beck J, Bolton EE, Brister JR, Chan J, Comeau DC, Connor R, DiCuccio M, Farrell CM, Feldgarden M, Fine AM, Funk K, Hatcher E, Hoeppner M, Kane M, Kannan S, Katz KS, Kelly C, Klimke W, Kim S, Kimchi A, Landrum M, Lathrop S, Lu Z, Malheiro A, Marchler-Bauer A, Murphy TD, Phan L, Prasad AB, Pujar S, Sawyer A, Schmieder E, Schneider VA, Schoch CL, Sharma S, Thibaud-Nissen F, Trawick BW, Venkatapathi T, Wang J, Pruitt KD, Sherry ST. Database resources of the National Center for Biotechnology Information. Nucleic Acids Res. 2024 Jan 5;52(D1):D33-D43. doi: 10.1093/nar/gkad1044. PMID: 37994677; PMCID: PMC10767890.
+The UniProt Consortium , UniProt: the Universal Protein Knowledgebase in 2023, Nucleic Acids Research, Volume 51, Issue D1, 6 January 2023, Pages D523–D531, https://doi.org/10.1093/nar/gkac1052
+If using gget muscle, please also cite:
If using gget opentargets, please also cite:
If using gget pdb, please also cite:
If using gget ref or gget search, please also cite:
If using gget seq, please also cite:
Martin FJ, Amode MR, Aneja A, Austine-Orimoloye O, Azov AG, Barnes I, Becker A, Bennett R, Berry A, Bhai J, Bhurji SK, Bignell A, Boddu S, Branco Lins PR, Brooks L, Ramaraju SB, Charkhchi M, Cockburn A, Da Rin Fiorretto L, Davidson C, Dodiya K, Donaldson S, El Houdaigui B, El Naboulsi T, Fatima R, Giron CG, Genez T, Ghattaoraya GS, Martinez JG, Guijarro C, Hardy M, Hollis Z, Hourlier T, Hunt T, Kay M, Kaykala V, Le T, Lemos D, Marques-Coelho D, Marugán JC, Merino GA, Mirabueno LP, Mushtaq A, Hossain SN, Ogeh DN, Sakthivel MP, Parker A, Perry M, Piližota I, Prosovetskaia I, Pérez-Silva JG, Salam AIA, Saraiva-Agostinho N, Schuilenburg H, Sheppard D, Sinha S, Sipos B, Stark W, Steed E, Sukumaran R, Sumathipala D, Suner MM, Surapaneni L, Sutinen K, Szpak M, Tricomi FF, Urbina-Gómez D, Veidenberg A, Walsh TA, Walts B, Wass E, Willhoft N, Allen J, Alvarez-Jarreta J, Chakiachvili M, Flint B, Giorgetti S, Haggerty L, Ilsley GR, Loveland JE, Moore B, Mudge JM, Tate J, Thybert D, Trevanion SJ, Winterbottom A, Frankish A, Hunt SE, Ruffier M, Cunningham F, Dyer S, Finn RD, Howe KL, Harrison PW, Yates AD, Flicek P. Ensembl 2023. Nucleic Acids Res. 2023 Jan 6;51(D1):D933-D941. doi: 10.1093/nar/gkac958. PMID: 36318249; PMCID: PMC9825606.
+The UniProt Consortium , UniProt: the Universal Protein Knowledgebase in 2023, Nucleic Acids Research, Volume 51, Issue D1, 6 January 2023, Pages D523–D531, https://doi.org/10.1093/nar/gkac1052
+gget is only as accurate as the databases/servers/APIs it queries from. The accuracy or reliability of the data is not guaranteed or warranted in any way and the providers disclaim liability of any kind whatsoever, including, without limitation, liability for quality, performance, merchantability and fitness for a particular purpose arising out of the use, or inability to use the data.
We as members, contributors, and leaders pledge to make participation in our +community a harassment-free experience for everyone, regardless of age, body +size, visible or invisible disability, ethnicity, sex characteristics, gender +identity and expression, level of experience, education, socio-economic status, +nationality, personal appearance, race, religion, or sexual identity +and orientation.
+We pledge to act and interact in ways that contribute to an open, welcoming, +diverse, inclusive, and healthy community.
+Examples of behavior that contributes to a positive environment for our +community include:
+Examples of unacceptable behavior include:
+Community leaders are responsible for clarifying and enforcing our standards of +acceptable behavior and will take appropriate and fair corrective action in +response to any behavior that they deem inappropriate, threatening, offensive, +or harmful.
+Community leaders have the right and responsibility to remove, edit, or reject +comments, commits, code, wiki edits, issues, and other contributions that are +not aligned to this Code of Conduct, and will communicate reasons for moderation +decisions when appropriate.
+This Code of Conduct applies within all community spaces, and also applies when +an individual is officially representing the community in public spaces. +Examples of representing our community include using an official e-mail address, +posting via an official social media account, or acting as an appointed +representative at an online or offline event.
+Instances of abusive, harassing, or otherwise unacceptable behavior may be +reported to the community leaders responsible for enforcement at +lpachter@caltech.edu. +All complaints will be reviewed and investigated promptly and fairly.
+All community leaders are obligated to respect the privacy and security of the +reporter of any incident.
+Community leaders will follow these Community Impact Guidelines in determining +the consequences for any action they deem in violation of this Code of Conduct:
+Community Impact: Use of inappropriate language or other behavior deemed +unprofessional or unwelcome in the community.
+Consequence: A private, written warning from community leaders, providing +clarity around the nature of the violation and an explanation of why the +behavior was inappropriate. A public apology may be requested.
+Community Impact: A violation through a single incident or series +of actions.
+Consequence: A warning with consequences for continued behavior. No +interaction with the people involved, including unsolicited interaction with +those enforcing the Code of Conduct, for a specified period of time. This +includes avoiding interactions in community spaces as well as external channels +like social media. Violating these terms may lead to a temporary or +permanent ban.
+Community Impact: A serious violation of community standards, including +sustained inappropriate behavior.
+Consequence: A temporary ban from any sort of interaction or public +communication with the community for a specified period of time. No public or +private interaction with the people involved, including unsolicited interaction +with those enforcing the Code of Conduct, is allowed during this period. +Violating these terms may lead to a permanent ban.
+Community Impact: Demonstrating a pattern of violation of community +standards, including sustained inappropriate behavior, harassment of an +individual, or aggression toward or disparagement of classes of individuals.
+Consequence: A permanent ban from any sort of public interaction within +the community.
+This Code of Conduct is adapted from the Contributor Covenant, +version 2.0, available at https://www.contributor-covenant.org/version/2/0/code_of_conduct.html.
+Community Impact Guidelines were inspired by Mozilla's code of conduct +enforcement ladder.
+For answers to common questions about this code of conduct, see the FAQ at +https://www.contributor-covenant.org/faq. Translations are available at +https://www.contributor-covenant.org/translations.
+ +Thank you for investing your time in contributing to our project! Any contribution you make will be reflected on the gget repo. ✨
+Read our Code of Conduct to keep our community approachable and respectable.
+In this guide you will get an overview of the contribution workflow from opening an issue or creating a pull request (PR) to reviewing and merging a PR.
+If you spot a problem with gget or you have an idea for a new feature, check if an issue already exists. If a related issue doesn't exist, you can open a new issue using the relevant issue form.
+Scan through our existing issues to find one that interests you. You can narrow down the search using labels as filters. If you find an issue to work on, you are welcome to open a PR with a fix.
Using GitHub Desktop:
+Using the command line:
+Commit the changes once you are happy with them.
+pip install -r dev-requirements.txtcoverage run -m pytest -ra -v tests && coverage report --omit=main.py,tests*If you have any questions, feel free to start a discussion or create an issue as described above.
+When you're finished with the changes, create a pull request, also known as a PR.
+‼️ Please make all PRs against the dev branch of the gget repository.
Once you submit your PR, a gget team member will review your proposal. We may ask questions or request additional information.
+Congratulations! 🎉 The gget team thanks you. ✨
+Once your PR is merged, your contributions will be publicly visible on the gget repo.
+ +++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Search for genes, mutations, and other factors associated with cancer using the COSMIC (Catalogue Of Somatic Mutations In Cancer) database.
+Return format: JSON (command-line) or data frame/CSV (Python) when download_cosmic=False. When download_cosmic=True, downloads the requested database into the specified folder.
This module was written in part by @AubakirovArman (information querying) and @josephrich98 (database download).
+NOTE: License fees apply for the commercial use of COSMIC. You can read more about licensing COSMIC data here.
+Positional argument (for querying information)
+searchterm
+Search term, which can be a mutation, or gene name (or Ensembl ID), or sample, etc.
+Examples for the searchterm and entitity arguments:
| searchterm | entitity | |
|---|---|---|
| EGFR | mutations | -> Find mutations in the EGFR gene that are associated with cancer |
| v600e | mutations | -> Find genes for which a v600e mutation is associated with cancer |
| COSV57014428 | mutations | -> Find mutations associated with this COSMIC mutations ID |
| EGFR | genes | -> Get the number of samples, coding/simple mutations, and fusions observed in COSMIC for EGFR |
| prostate | cancer | -> Get number of tested samples and mutations for prostate cancer |
| prostate | tumour_site | -> Get number of tested samples, genes, mutations, fusions, etc. with 'prostate' as primary tissue site |
| ICGC | studies | -> Get project code and descriptions for all studies from the ICGC (International Cancer Genome Consortium) |
| EGFR | pubmed | -> Find PubMed publications on EGFR and cancer |
| ICGC | samples | -> Get metadata on all samples from the ICGC (International Cancer Genome Consortium) |
| COSS2907494 | samples | -> Get metadata on this COSMIC sample ID (cancer type, tissue, # analyzed genes, # mutations, etc.) |
NOTE: (Python only) Set to None when downloading COSMIC databases with download_cosmic=True.
Optional arguments (for querying information)
+-e --entity
+'mutations' (default), 'genes', 'cancer', 'tumour site', 'studies', 'pubmed', or 'samples'.
+Defines the type of the results to return.
-l --limit
+Limits number of hits to return. Default: 100.
Flags (for downloading COSMIC databases)
+-d --download_cosmic
+Switches into database download mode.
-gm --gget_mutate
+TURNS OFF creation of a modified version of the database for use with gget mutate.
+Python: gget_mutate is True by default. Set gget_mutate=False to disable.
Optional arguments (for downloading COSMIC databases)
+-mc --mutation_class
+'cancer' (default), 'cell_line', 'census', 'resistance', 'genome_screen', 'targeted_screen', or 'cancer_example'
+Type of COSMIC database to download.
-cv --cosmic_version
+Version of the COSMIC database. Default: None -> Defaults to latest version.
-gv --grch_version
+Version of the human GRCh reference genome the COSMIC database was based on (37 or 38). Default: 37
--keep_genome_info
+Whether to keep genome information in the modified database for use with gget mutate. Default: False
--remove_duplicates
+Whether to remove duplicate rows from the modified database for use with gget mutate. Default: False
Optional arguments (general)
+-o --out
+Path to the file (or folder when downloading databases with the download_cosmic flag) the results will be saved in, e.g. 'path/to/results.json'.
+Default: None
+-> When download_cosmic=False: Results will be returned to standard out
+-> When download_cosmic=True: Database will be downloaded into current working directory
Flags (general)
+-csv --csv
+Command-line only. Returns results in CSV format.
+Python: Use json=True to return output in JSON format.
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
gget cosmic EGFR
+# Python
+gget.cosmic("EGFR")
+→ Returns mutations in the EGFR gene that are associated with cancer in the format:
+| Gene | Syntax | Alternate IDs | Canonical |
|---|---|---|---|
| EGFR | c.*2446A>G | EGFR c.*2446A>G, EGFR p.?, ... | y |
| EGFR | c.(2185_2283)ins(18) | EGFR c.(2185_2283)ins(18), EGFR p.?, ... | y |
| . . . | . . . | . . . | . . . |
gget cosmic --download_cosmic
+# Python
+gget.cosmic(searchterm=None, download_cosmic=True)
+→ Downloads the COSMIC cancer database of the latest COSMIC release into the current working directory.
+If you use gget cosmic in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Tate JG, Bamford S, Jubb HC, Sondka Z, Beare DM, Bindal N, Boutselakis H, Cole CG, Creatore C, Dawson E, Fish P, Harsha B, Hathaway C, Jupe SC, Kok CY, Noble K, Ponting L, Ramshaw CC, Rye CE, Speedy HE, Stefancsik R, Thompson SL, Wang S, Ward S, Campbell PJ, Forbes SA. COSMIC: the Catalogue Of Somatic Mutations In Cancer. Nucleic Acids Res. 2019 Jan 8;47(D1):D941-D947. doi: 10.1093/nar/gky1015. PMID: 30371878; PMCID: PMC6323903.
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Align multiple protein or translated DNA sequences using DIAMOND (DIAMOND is similar to BLAST, but this is a local computation).
+Return format: JSON (command-line) or data frame/CSV (Python).
Positional argument
+query
+Sequences (str or list) or path to FASTA file containing sequences to be aligned against the reference.
Required arguments
+-ref --reference
+Reference sequences (str or list) or path to FASTA file containing reference sequences.
Optional arguments
+-db --diamond_db
+Path to save DIAMOND database created from reference (str).
+Default: None -> Temporary db file will be deleted after alignment or saved in out if out is provided.
-s --sensitivity
+Sensitivity of alignment (str). Default: "very-sensitive".
+One of the following: fast, mid-sensitive, sensitive, more-sensitive, very-sensitive, or ultra-sensitive.
-t --threads
+Number of threads used (int). Default: 1.
-db --diamond_binary
+Path to DIAMOND binary (str). Default: None -> Uses DIAMOND binary installed with gget.
-o --out
+Path to the folder to save results in (str), e.g. "path/to/directory". Default: Standard out; temporary files are deleted.
Flags
+-csv --csv
+Command-line only. Returns results in CSV format.
+Python: Use json=True to return output in JSON format.
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
# !!! Make sure to list the positional argument first here so it is not added as a reference sequence
+gget diamond GGETISAWESQME ELVISISALIVE LQVEFRANKLIN PACHTERLABRQCKS -ref GGETISAWESQMEELVISISALIVELQVEFRANKLIN PACHTERLABRQCKS
+# Python
+gget.diamond(["GGETISAWESQME", "ELVISISALIVE", "LQVEFRANKLIN", "PACHTERLABRQCKS"], reference=["GGETISAWESQMEELVISISALIVELQVEFRANKLIN", "PACHTERLABRQCKS"])
+→ Returns results in JSON (command-line) or data frame/CSV (Python) format:
+| query_accession | subject_accession | identity_percentage | query_seq_length | subject_seq_length | length | mismatches | gap_openings | query_start | query_end | subject_start | subject_end | e-value | bit_score |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Seq0 | Seq0 | 100 | 13 | 37 | 13 | 0 | 0 | 1 | 13 | 1 | 13 | 2.82e-09 | 30.8 |
| Seq2 | Seq0 | 100 | 12 | 37 | 12 | 0 | 0 | 1 | 12 | 26 | 37 | 4.35e-08 | 27.7 |
| Seq3 | Seq1 | 100 | 15 | 15 | 15 | 0 | 0 | 1 | 15 | 1 | 15 | 2.01e-11 | 36.2 |
If you use gget diamond in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Buchfink, B., Xie, C. & Huson, D. Fast and sensitive protein alignment using DIAMOND. Nat Methods 12, 59–60 (2015). https://doi.org/10.1038/nmeth.3176
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Locally predict Eukaryotic Linear Motifs from an amino acid sequence or UniProt Acc using data from the ELM database.
+Return format: JSON (command-line) or data frame/CSV (Python). This module returns two data frames (or JSON formatted files) (see examples).
ELM data can be downloaded & distributed for non-commercial use according to the ELM Software License Agreement.
+Before using gget elm for the first time, run gget setup elm (bash) / gget.setup("elm") (Python) once (also see gget setup).
Positional argument
+sequence
+Amino acid sequence or Uniprot Acc (str).
+When providing a Uniprot Acc, use flag --uniprot (Python: uniprot=True).
Optional arguments
+-s --sensitivity
+Sensitivity of DIAMOND alignment (str). Default: "very-sensitive".
+One of the following: fast, mid-sensitive, sensitive, more-sensitive, very-sensitive, or ultra-sensitive.
-t --threads
+Number of threads used in DIAMOND alignment (int). Default: 1.
-bin --diamond_binary
+Path to DIAMOND binary (str). Default: None -> Uses DIAMOND binary installed with gget.
-o --out
+Path to the folder to save results in (str), e.g. "path/to/directory". Default: Standard out; temporary files are deleted.
Flags
+-u --uniprot
+Set to True if sequence is a Uniprot Acc instead of an amino acid sequence.
-e --expand
+Expand the information returned in the regex data frame to include the protein names, organisms, and references that the motif was orignally validated on.
-csv --csv
+Command-line only. Returns results in CSV format.
+Python: Use json=True to return output in JSON format.
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
Find ELMs in an amino acid sequence:
+gget setup elm # Downloads/updates local ELM database
+gget elm -o gget_elm_results LIAQSIGQASFV
+# Python
+gget.setup(“elm”) # Downloads/updates local ELM database
+ortholog_df, regex_df = gget.elm("LIAQSIGQASFV")
+Find ELMs giving a UniProt Acc as input:
+gget setup elm # Downloads/updates local ELM database
+gget elm -o gget_elm_results --uniprot Q02410 -e
+# Python
+gget.setup(“elm”) # Downloads/updates local ELM database
+ortholog_df, regex_df = gget.elm("Q02410", uniprot=True, expand=True)
+→ Returns two data frames (or JSON formatted dictionaries for command line) containing extensive information about linear motifs associated with orthologous proteins and motifs found in the input sequence directly based on their regex expressions:
+ortholog_df:
+| Ortholog_UniProt_Acc | ProteinName | class_accession | ELMIdentifier | FunctionalSiteName | Description | Organism | … |
|---|---|---|---|---|---|---|---|
| Q02410 | APBA1_HUMAN | ELME000357 | LIG_CaMK_CASK_1 | CASK CaMK domain binding ligand motif | Motif that mediates binding to the calmodulin-dependent protein kinase (CaMK) domain of the peripheral plasma membrane protein CASK/Lin2. | Homo sapiens | … |
| Q02410 | APBA1_HUMAN | ELME000091 | LIG_PDZ_Class_2 | PDZ domain ligands | The C-terminal class 2 PDZ-binding motif is classically represented by a pattern such as | Homo sapiens | … |
regex_df:
+| Instance_accession | ELMIdentifier | FunctionalSiteName | ELMType | Description | Instances (Matched Sequence) | Organism | … |
|---|---|---|---|---|---|---|---|
| ELME000321 | CLV_C14_Caspase3-7 | Caspase cleavage motif | CLV | Caspase-3 and Caspase-7 cleavage site. | ERSDG | Mus musculus | … |
| ELME000102 | CLV_NRD_NRD_1 | NRD cleavage site | CLV | N-Arg dibasic convertase (NRD/Nardilysin) cleavage site. | RRA | Rattus norvegicus | … |
| ELME000100 | CLV_PCSK_PC1ET2_1 | PCSK cleavage site | CLV | NEC1/NEC2 cleavage site. | KRD | Mus musculus | … |
| ELME000146 | CLV_PCSK_SKI1_1 | PCSK cleavage site | CLV | Subtilisin/kexin isozyme-1 (SKI1) cleavage site. | RLLTA | Homo sapiens | … |
| ELME000231 | DEG_APCC_DBOX_1 | APCC-binding Destruction motifs | DEG | An RxxL-based motif that binds to the Cdh1 and Cdc20 components of APC/C thereby targeting the protein for destruction in a cell cycle dependent manner | SRVKLNIVR | Saccharomyces cerevisiae S288c | … |
| … | … | … | … | … | … | … | … |
gget elm demogget elm results based on disordered protein regionsIf you use gget elm in a publication, please cite the following articles:
Laura Luebbert, Chi Hoang, Manjeet Kumar, Lior Pachter, Fast and scalable querying of eukaryotic linear motifs with gget elm, Bioinformatics, 2024, btae095, https://doi.org/10.1093/bioinformatics/btae095
+Manjeet Kumar, Sushama Michael, Jesús Alvarado-Valverde, Bálint Mészáros, Hugo Sámano‐Sánchez, András Zeke, Laszlo Dobson, Tamas Lazar, Mihkel Örd, Anurag Nagpal, Nazanin Farahi, Melanie Käser, Ramya Kraleti, Norman E Davey, Rita Pancsa, Lucía B Chemes, Toby J Gibson, The Eukaryotic Linear Motif resource: 2022 release, Nucleic Acids Research, Volume 50, Issue D1, 7 January 2022, Pages D497–D508, https://doi.org/10.1093/nar/gkab975
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Perform an enrichment analysis on a list of genes using Enrichr or modEnrichr.
+Return format: JSON (command-line) or data frame/CSV (Python).
Positional argument
+genes
+Short names (gene symbols) of genes to perform enrichment analysis on, e.g. PHF14 RBM3 MSL1 PHF21A.
+Alternatively: use flag --ensembl to input a list of Ensembl gene IDs, e.g. ENSG00000106443 ENSG00000102317 ENSG00000188895.
Other required arguments
+-db --database
+Database database to use as reference for the enrichment analysis.
+Supports any database listed here under 'Gene-set Library' or one of the following shortcuts:
+'pathway' (KEGG_2021_Human)
+'transcription' (ChEA_2016)
+'ontology' (GO_Biological_Process_2021)
+'diseases_drugs' (GWAS_Catalog_2019)
+'celltypes' (PanglaoDB_Augmented_2021)
+'kinase_interactions' (KEA_2015)
NOTE: database shortcuts are not supported for species other than 'human' or 'mouse'. Click on the species databases listed below under species to view a list of databases available for each species.
Optional arguments
+-s --species
+Species to use as reference for the enrichment analysis. (Default: human)
+Options:
| Species | Database list |
|---|---|
human | Enrichr |
mouse | Equivalent to human |
fly | FlyEnrichr |
yeast | YeastEnrichr |
worm | WormEnrichr |
fish | FishEnrichr |
-bkg_l --background_list
+Short names (gene symbols) of background genes to perform enrichment analysis on, e.g. NSUN3 POLRMT NLRX1.
+Alternatively: use flag --ensembl_background to input a list of Ensembl gene IDs.
+See this Tweetorial to learn why you should use a background gene list when performing an enrichment analysis.
-o --out
+Path to the file the results will be saved in, e.g. path/to/directory/results.csv (or .json). (Default: Standard out.)
+Python: save=True will save the output in the current working directory.
-ko --kegg_out
+Path to the png file the marked KEGG pathway images will be saved in, e.g. path/to/directory/pathway.png. (Default: None)
-kr --kegg_rank
+Rank of the KEGG pathway to be plotted. (Default: 1)
figsize
+Python only. (width, height) of plot in inches. (Default: (10,10))
ax
+Python only. Pass a matplotlib axes object for plot customization. (Default: None)
Flags
+-e --ensembl
+Add this flag if genes are given as Ensembl gene IDs.
-e_b --ensembl_background
+Add this flag if background_list are given as Ensembl gene IDs.
-bkg --background
+If True, use set of > 20,000 default background genes listed here.
-csv --csv
+Command-line only. Returns results in CSV format.
+Python: Use json=True to return output in JSON format.
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
plot
+Python only. plot=True provides a graphical overview of the first 15 results (default: False).
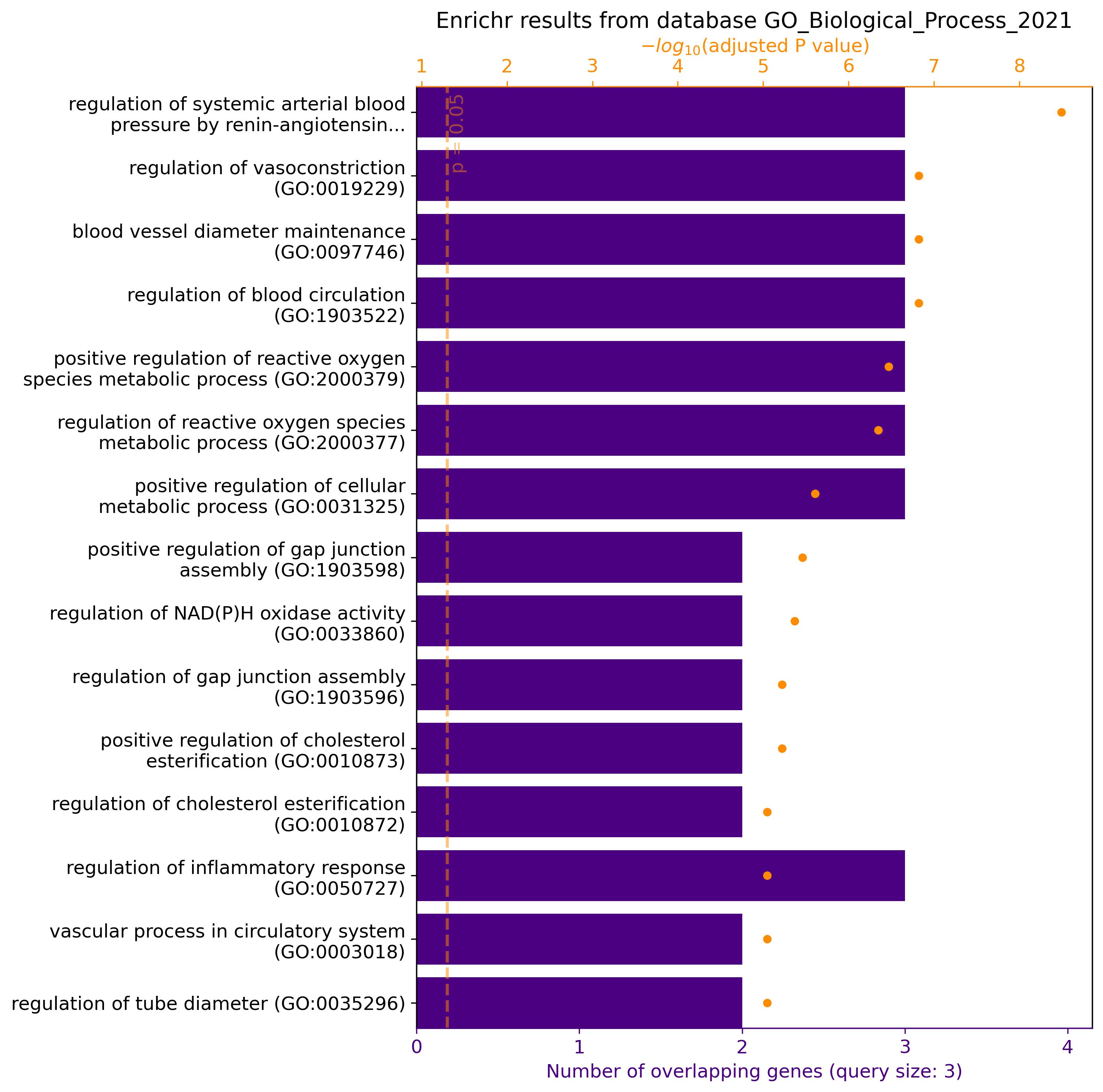
gget enrichr -db ontology ACE2 AGT AGTR1
+# Python
+gget.enrichr(["ACE2", "AGT", "AGTR1"], database="ontology", plot=True)
+→ Returns pathways/functions involving genes ACE2, AGT, and AGTR1 from the GO Biological Process 2021 database. In Python, plot=True returns a graphical overview of the results:
Use gget enrichr with a background gene list:
+See this Tweetorial to learn why you should use a background gene list when performing an enrichment analysis.
# Here, we are passing the input genes first (positional argument 'genes'), so they are not added to the background gene list behind the '-bkgr_l' argument
+gget enrichr \
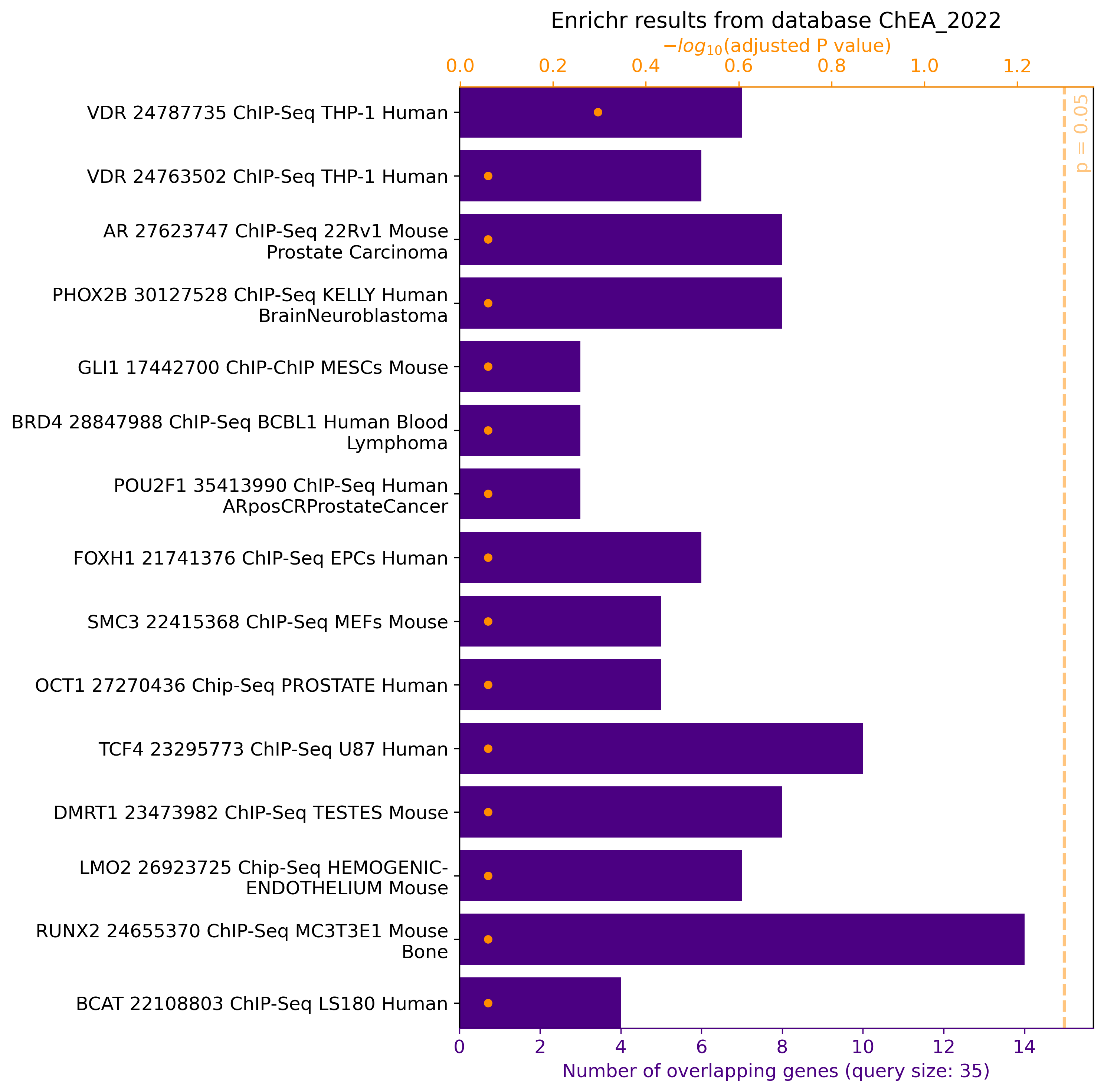
+ PHF14 RBM3 MSL1 PHF21A ARL10 INSR JADE2 P2RX7 LINC00662 CCDC101 PPM1B KANSL1L CRYZL1 ANAPC16 TMCC1 CDH8 RBM11 CNPY2 HSPA1L CUL2 PLBD2 LARP7 TECPR2 ZNF302 CUX1 MOB2 CYTH2 SEC22C EIF4E3 ROBO2 ADAMTS9-AS2 CXXC1 LINC01314 ATF7 ATP5F1 \
+ -db ChEA_2022 \
+ -bkg_l NSUN3 POLRMT NLRX1 SFXN5 ZC3H12C SLC25A39 ARSG DEFB29 PCMTD2 ACAA1A LRRC1 2810432D09RIK SEPHS2 SAC3D1 TMLHE LOC623451 TSR2 PLEKHA7 GYS2 ARHGEF12 HIBCH LYRM2 ZBTB44 ENTPD5 RAB11FIP2 LIPT1 INTU ANXA13 KLF12 SAT2 GAL3ST2 VAMP8 FKBPL AQP11 TRAP1 PMPCB TM7SF3 RBM39 BRI3 KDR ZFP748 NAP1L1 DHRS1 LRRC56 WDR20A STXBP2 KLF1 UFC1 CCDC16 9230114K14RIK RWDD3 2610528K11RIK ACO1 CABLES1 LOC100047214 YARS2 LYPLA1 KALRN GYK ZFP787 ZFP655 RABEPK ZFP650 4732466D17RIK EXOSC4 WDR42A GPHN 2610528J11RIK 1110003E01RIK MDH1 1200014M14RIK AW209491 MUT 1700123L14RIK 2610036D13RIK PHF14 RBM3 MSL1 PHF21A ARL10 INSR JADE2 P2RX7 LINC00662 CCDC101 PPM1B KANSL1L CRYZL1 ANAPC16 TMCC1 CDH8 RBM11 CNPY2 HSPA1L CUL2 PLBD2 LARP7 TECPR2 ZNF302 CUX1 MOB2 CYTH2 SEC22C EIF4E3 ROBO2 ADAMTS9-AS2 CXXC1 LINC01314 ATF7 ATP5F1COX15 TMEM30A NSMCE4A TM2D2 RHBDD3 ATXN2 NFS1 3110001I20RIK BC038156 C330002I19RIK ZFYVE20 POLI TOMM70A LOC100047782 2410012H22RIK RILP A230062G08RIK PTTG1IP RAB1 AFAP1L1 LYRM5 2310026E23RIK SLC7A6OS MAT2B 4932438A13RIK LRRC8A SMO NUPL2
+# Python
+gget.enrichr(
+ genes = [
+ "PHF14", "RBM3", "MSL1", "PHF21A", "ARL10", "INSR", "JADE2", "P2RX7",
+ "LINC00662", "CCDC101", "PPM1B", "KANSL1L", "CRYZL1", "ANAPC16", "TMCC1",
+ "CDH8", "RBM11", "CNPY2", "HSPA1L", "CUL2", "PLBD2", "LARP7", "TECPR2",
+ "ZNF302", "CUX1", "MOB2", "CYTH2", "SEC22C", "EIF4E3", "ROBO2",
+ "ADAMTS9-AS2", "CXXC1", "LINC01314", "ATF7", "ATP5F1"
+ ],
+ database = "ChEA_2022",
+ background_list = [
+ "NSUN3","POLRMT","NLRX1","SFXN5","ZC3H12C","SLC25A39","ARSG",
+ "DEFB29","PCMTD2","ACAA1A","LRRC1","2810432D09RIK","SEPHS2",
+ "SAC3D1","TMLHE","LOC623451","TSR2","PLEKHA7","GYS2","ARHGEF12",
+ "HIBCH","LYRM2","ZBTB44","ENTPD5","RAB11FIP2","LIPT1",
+ "INTU","ANXA13","KLF12","SAT2","GAL3ST2","VAMP8","FKBPL",
+ "AQP11","TRAP1","PMPCB","TM7SF3","RBM39","BRI3","KDR","ZFP748",
+ "NAP1L1","DHRS1","LRRC56","WDR20A","STXBP2","KLF1","UFC1",
+ "CCDC16","9230114K14RIK","RWDD3","2610528K11RIK","ACO1",
+ "CABLES1", "LOC100047214","YARS2","LYPLA1","KALRN","GYK",
+ "ZFP787","ZFP655","RABEPK","ZFP650","4732466D17RIK","EXOSC4",
+ "WDR42A","GPHN","2610528J11RIK","1110003E01RIK","MDH1","1200014M14RIK",
+ "AW209491","MUT","1700123L14RIK","2610036D13RIK",
+ "PHF14", "RBM3", "MSL1", "PHF21A", "ARL10", "INSR", "JADE2",
+ "P2RX7", "LINC00662", "CCDC101", "PPM1B", "KANSL1L", "CRYZL1",
+ "ANAPC16", "TMCC1","CDH8", "RBM11", "CNPY2", "HSPA1L", "CUL2",
+ "PLBD2", "LARP7", "TECPR2", "ZNF302", "CUX1", "MOB2", "CYTH2",
+ "SEC22C", "EIF4E3", "ROBO2", "ADAMTS9-AS2", "CXXC1", "LINC01314", "ATF7",
+ "ATP5F1""COX15","TMEM30A","NSMCE4A","TM2D2","RHBDD3","ATXN2","NFS1",
+ "3110001I20RIK","BC038156","C330002I19RIK","ZFYVE20","POLI","TOMM70A",
+ "LOC100047782","2410012H22RIK","RILP","A230062G08RIK",
+ "PTTG1IP","RAB1","AFAP1L1", "LYRM5","2310026E23RIK",
+ "SLC7A6OS","MAT2B","4932438A13RIK","LRRC8A","SMO","NUPL2"
+ ],
+ plot=True
+)
+→ Returns hits of the input gene list given the background gene list from the transcription factor/target library ChEA 2022. In Python, plot=True returns a graphical overview of the results:
Generate a KEGG pathway image with the genes from the enrichment analysis highlighted:
+This feature is available thanks to a PR by Noriaki Sato.
gget enrichr -db pathway --kegg_out kegg.png --kegg_rank 1 ZBP1 IRF3 RIPK1
+# Python
+gget.enrichr(["ZBP1", "IRF3", "RIPK1"], database="pathway", kegg_out="kegg.png", kegg_rank=1)
+→ In addition to the standard gget enrichr output, the kegg_out argument saves an image with the genes from the enrichment analysis highlighted in the KEGG pathway:
The following example was submitted by Dylan Lawless via PR:
+Use gget enrichr in R and create a similar plot using ggplot.
+NOTE the switch of axes compared to the Python plot.
system("pip install gget")
+install.packages("reticulate")
+library(reticulate)
+gget <- import("gget")
+
+# Perform enrichment analysis on a list of genes
+df <- gget$enrichr(list("ACE2", "AGT", "AGTR1"), database = "ontology")
+
+# Count number of overlapping genes
+df$overlapping_genes_count <- lapply(df$overlapping_genes, length) |> as.numeric()
+
+# Only keep the top 15 results
+df <- df[1:15, ]
+
+# Plot
+library(ggplot2)
+
+df |>
+ ggplot() +
+ geom_bar(aes(
+ x = -log10(adj_p_val),
+ y = reorder(path_name, -adj_p_val)
+ ),
+ stat = "identity",
+ fill = "lightgrey",
+ width = 0.5,
+ color = "black") +
+ geom_text(
+ aes(
+ y = path_name,
+ x = (-log10(adj_p_val)),
+ label = overlapping_genes_count
+ ),
+ nudge_x = 0.75,
+ show.legend = NA,
+ color = "red"
+ ) +
+ geom_text(
+ aes(
+ y = Inf,
+ x = Inf,
+ hjust = 1,
+ vjust = 1,
+ label = "# of overlapping genes"
+ ),
+ show.legend = NA,
+ color = "red"
+ ) +
+ geom_vline(linetype = "dotted", linewidth = 1, xintercept = -log10(0.05)) +
+ ylab("Pathway name") +
+ xlab("-log10(adjusted P value)")
+Using gget enrichr with background genes
If you use gget enrichr in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Chen EY, Tan CM, Kou Y, Duan Q, Wang Z, Meirelles GV, Clark NR, Ma'ayan A. Enrichr: interactive and collaborative HTML5 gene list enrichment analysis tool. BMC Bioinformatics. 2013; 128(14). https://doi.org/10.1186/1471-2105-14-128
+Kuleshov MV, Jones MR, Rouillard AD, Fernandez NF, Duan Q, Wang Z, Koplev S, Jenkins SL, Jagodnik KM, Lachmann A, McDermott MG, Monteiro CD, Gundersen GW, Ma'ayan A. Enrichr: a comprehensive gene set enrichment analysis web server 2016 update. Nucleic Acids Research. 2016; gkw377. doi: 10.1093/nar/gkw377
+Xie Z, Bailey A, Kuleshov MV, Clarke DJB., Evangelista JE, Jenkins SL, Lachmann A, Wojciechowicz ML, Kropiwnicki E, Jagodnik KM, Jeon M, & Ma’ayan A. Gene set knowledge discovery with Enrichr. Current Protocols, 1, e90. 2021. doi: 10.1002/cpz1.90.
+If working with non-human/mouse datasets, please also cite:
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Generates natural language text based on a given prompt using the OpenAI API's 'openai.ChatCompletion.create' endpoint. +This module, including its source code, documentation and unit tests, were partly written by OpenAI's Chat-GTP3.
+NOTE:
+OpenAI API calls are only 'free' for the first three months after generating your OpenAI Account (OpenAI provides a $5 credit that expires).
+You can define a hard monthly billing limit (e.g. $1) here.
+See their pricing and FAQ here.
+Get your OpenAI API key here.
Returns: A string containing the generated text.
+Before using gget gpt for the first time, run gget setup gpt / gget.setup("gpt") once (also see gget setup).
Positional argument
+prompt
+The input prompt to generate text from (str).
api_key
+Your OpenAI API key (str) (get your API key).
Optional arguments
+-m --model
+The name of the GPT model to use for generating the text (str). Default is "gpt-3.5-turbo".
+See https://platform.openai.com/docs/models/gpt-4 for more information on the available models.
-temp --temperature
+Value between 0 and 2 that controls the level of randomness and creativity in the generated text (float).
+Higher values result in more creative and varied text. Default is 1.
-tp --top_p
+Controls the diversity of the generated text as an alternative to sampling with temperature (float).
+Higher values result in more diverse and unexpected text. Default is 1.
+Note: OpenAI recommends altering this or temperature but not both.
-s --stop
+A sequence of tokens to mark the end of the generated text (str). Default is None.
-mt --max_tokens
+Controls the maximum length of the generated text, in tokens (int). Default is 200.
-pp --presence_penalty
+Number between -2.0 and 2.0. Higher values result increase the model's likelihood to talk about new topics (float). Default is 0.
-fp --frequency_penalty
+Number between -2.0 and 2.0. Higher values decrease the model's likelihood to repeat the same line verbatim (float). Default is 0.
-lb --logit_bias
+A dictionary that specifies a bias towards certain tokens in the generated text (dict). Default is None.
-o --out
+If provided, saves the generated text to a file with the specified path (str). Default: Standard out.
gget gpt "How are you today GPT?" your_api_token
+# Python
+print(gget.gpt("How are you today GPT?", "your_api_token"))
+ +
+
+
+ ++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Fetch extensive gene and transcript metadata from Ensembl, UniProt, and NCBI using Ensembl IDs.
+Return format: JSON (command-line) or data frame/CSV (Python).
Positional argument
+ens_ids
+One or more Ensembl IDs (WormBase and Flybase IDs are also supported).
Optional arguments
+-o --out
+Path to the file the results will be saved in, e.g. path/to/directory/results.csv (or .json). Default: Standard out.
+Python: save=True will save the output in the current working directory.
Flags
+-n --ncbi
+TURN OFF results from NCBI.
+Python: ncbi=False prevents data retrieval from NCBI (default: True).
-u --uniprot
+TURN OFF results from UniProt.
+Python: uniprot=False prevents data retrieval from UniProt (default: True).
-pdb --pdb
+INCLUDE PDB IDs in output (might increase runtime).
+Python: pdb=True includes PDB IDs in the results (default: False).
-csv --csv
+Command-line only. Returns results in CSV format.
+Python: Use json=True to return output in JSON format.
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
wrap_text
+Python only. wrap_text=True displays data frame with wrapped text for easy reading (default: False).
gget info ENSG00000034713 ENSG00000104853 ENSG00000170296
+# Python
+gget.info(["ENSG00000034713", "ENSG00000104853", "ENSG00000170296"])
+→ Returns extensive information about each requested Ensembl ID:
+| uniprot_id | ncbi_gene_id | primary_gene_name | synonyms | protein_names | ensembl_description | uniprot_description | ncbi_description | biotype | canonical_transcript | ... | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ENSG00000034713 | P60520 | 11345 | GABARAPL2 | [ATG8, ATG8C, FLC3A, GABARAPL2, GATE-16, GATE16, GEF-2, GEF2] | Gamma-aminobutyric acid receptor-associated protein like 2 (GABA(A) receptor-associated protein-like 2)... | GABA type A receptor associated protein like 2 [Source:HGNC Symbol;Acc:HGNC:13291] | FUNCTION: Ubiquitin-like modifier involved in intra- Golgi traffic (By similarity). Modulates intra-Golgi transport through coupling between NSF activity and ... | Enables ubiquitin protein ligase binding activity. Involved in negative regulation of proteasomal protein catabolic process and protein... | protein_coding | ENST00000037243.7 | ... |
| . . . | . . . | . . . | . . . | . . . | . . . | . . . | . . . | . . . | . . . | . . . | ... |
If you use gget info in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Martin FJ, Amode MR, Aneja A, Austine-Orimoloye O, Azov AG, Barnes I, Becker A, Bennett R, Berry A, Bhai J, Bhurji SK, Bignell A, Boddu S, Branco Lins PR, Brooks L, Ramaraju SB, Charkhchi M, Cockburn A, Da Rin Fiorretto L, Davidson C, Dodiya K, Donaldson S, El Houdaigui B, El Naboulsi T, Fatima R, Giron CG, Genez T, Ghattaoraya GS, Martinez JG, Guijarro C, Hardy M, Hollis Z, Hourlier T, Hunt T, Kay M, Kaykala V, Le T, Lemos D, Marques-Coelho D, Marugán JC, Merino GA, Mirabueno LP, Mushtaq A, Hossain SN, Ogeh DN, Sakthivel MP, Parker A, Perry M, Piližota I, Prosovetskaia I, Pérez-Silva JG, Salam AIA, Saraiva-Agostinho N, Schuilenburg H, Sheppard D, Sinha S, Sipos B, Stark W, Steed E, Sukumaran R, Sumathipala D, Suner MM, Surapaneni L, Sutinen K, Szpak M, Tricomi FF, Urbina-Gómez D, Veidenberg A, Walsh TA, Walts B, Wass E, Willhoft N, Allen J, Alvarez-Jarreta J, Chakiachvili M, Flint B, Giorgetti S, Haggerty L, Ilsley GR, Loveland JE, Moore B, Mudge JM, Tate J, Thybert D, Trevanion SJ, Winterbottom A, Frankish A, Hunt SE, Ruffier M, Cunningham F, Dyer S, Finn RD, Howe KL, Harrison PW, Yates AD, Flicek P. Ensembl 2023. Nucleic Acids Res. 2023 Jan 6;51(D1):D933-D941. doi: 10.1093/nar/gkac958. PMID: 36318249; PMCID: PMC9825606.
+Sayers EW, Beck J, Bolton EE, Brister JR, Chan J, Comeau DC, Connor R, DiCuccio M, Farrell CM, Feldgarden M, Fine AM, Funk K, Hatcher E, Hoeppner M, Kane M, Kannan S, Katz KS, Kelly C, Klimke W, Kim S, Kimchi A, Landrum M, Lathrop S, Lu Z, Malheiro A, Marchler-Bauer A, Murphy TD, Phan L, Prasad AB, Pujar S, Sawyer A, Schmieder E, Schneider VA, Schoch CL, Sharma S, Thibaud-Nissen F, Trawick BW, Venkatapathi T, Wang J, Pruitt KD, Sherry ST. Database resources of the National Center for Biotechnology Information. Nucleic Acids Res. 2024 Jan 5;52(D1):D33-D43. doi: 10.1093/nar/gkad1044. PMID: 37994677; PMCID: PMC10767890.
+The UniProt Consortium , UniProt: the Universal Protein Knowledgebase in 2023, Nucleic Acids Research, Volume 51, Issue D1, 6 January 2023, Pages D523–D531, https://doi.org/10.1093/nar/gkac1052
+pip install --upgrade gget
+Alternative:
+conda install -c bioconda gget
+For use in Jupyter Lab / Google Colab:
+import gget
+gget is a free, open-source command-line tool and Python package that enables efficient querying of genomic databases.
+
+gget consists of a collection of separate but interoperable modules, each designed to facilitate one type of database querying in a single line of code.
+
+NOTE: The databases queried by gget are continuously being updated which sometimes changes their structure. gget modules are tested automatically on a biweekly basis and updated to match new database structures when necessary. If you encounter a problem, please upgrade to the latest gget version using pip install --upgrade gget. If the problem persists, please report the issue.
+
+
Request a new feature
+
+
+++
gget infoandgget seqare currently unable to fetch information for WormBase and FlyBase IDs (all other IDs are functioning normally). This issue arose due to a bug in Ensembl release 112. We appreciate Ensembl's efforts in addressing this issue and expect a fix soon. Thank you for your patience.
These are the gget core modules. Click on any module to access detailed documentation.
| gget alphafold Predict 3D protein structure from an amino acid sequence. |
+ gget archs4 What is the expression of my gene in tissue X? |
+ gget bgee Find all orthologs of a gene. |
+ gget blast BLAST a nucleotide or amino acid sequence. |
+
| gget blat Find the genomic location of a nucleotide or amino acid sequence. |
+ gget cbio Explore a gene's expression in the specified cancers. |
+ gget cellxgene Get ready-to-use single-cell RNA seq count matrices from certain tissues/ diseases/ etc. |
+ gget cosmic Search for genes, mutations, and other factors associated with certain cancers. |
+
| gget diamond Align amino acid sequences to a reference. |
+ gget elm Find protein interaction domains and functions in an amino acid sequence. |
+ gget enrichr Check if a list of genes is associated with a specific celltype/ pathway/ disease/ etc. |
+ gget info Fetch all of the information associated with an Ensembl ID. |
+
| gget muscle Align multiple nucleotide or amino acid sequences to each other. |
+ gget mutate Mutate nucleotide sequences based on specified mutations. |
+ gget opentargets Explore which diseases and drugs a gene is associated with. |
+ gget pdb Fetch data from the Protein Data Bank (PDB) based on a PDB ID. |
+
| gget ref Get reference genomes from Ensembl. |
+ gget search Find Ensembl IDs associated with the specified search word. |
+ gget seq Fetch the nucleotide or amino acid sequence of a gene. |
+
If you use gget in a publication, please cite*:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Read the article here: https://doi.org/10.1093/bioinformatics/btac836
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Align multiple nucleotide or amino acid sequences to each other using Muscle5.
+Return format: ClustalW formatted standard out or aligned FASTA (.afa).
Positional argument
+fasta
+List of sequences or path to FASTA or .txt file containing the nucleotide or amino acid sequences to be aligned.
Optional arguments
+-o --out
+Path to the aligned FASTA file the results will be saved in, e.g. path/to/directory/results.afa. Default: Standard out.
+Python: save=True will save the output in the current working directory.
Flags
+-s5 --super5
+Aligns input using the Super5 algorithm instead of the Parallel Perturbed Probcons (PPP) algorithm to decrease time and memory.
+Use for large inputs (a few hundred sequences).
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
gget muscle MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS
+# Python
+gget.muscle(["MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS", "MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS"])
+gget muscle fasta.fa
+# Python
+gget.muscle("fasta.fa")
+→ Returns an overview of the aligned sequences with ClustalW coloring. (To return an aligned FASTA (.afa) file, use --out argument (or save=True in Jupyter Lab/Google Colab).) In the above example, the 'fasta.fa' includes several sequences to be aligned (e.g. isoforms returned from gget seq).

You can also view aligned fasta files returned by gget.muscle using programs like alv, as shown below:
# Python
+!pip install biopython
+!pip install alv
+from Bio import AlignIO
+import alv
+
+gget.muscle("fasta.fa", out="fasta_aligned.afa")
+msa = AlignIO.read("fasta_aligned.afa", "fasta")
+alv.view(msa)
+If you use gget muscle in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Edgar RC (2021), MUSCLE v5 enables improved estimates of phylogenetic tree confidence by ensemble bootstrapping, bioRxiv 2021.06.20.449169. https://doi.org/10.1101/2021.06.20.449169
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Takes in nucleotide sequences and mutations (in standard mutation annotation and returns mutated versions of the input sequences according to the provided mutations.
+Return format: Saves mutated sequences in FASTA format (or returns a list containing the mutated sequences if out=None).
This module was written by Joseph Rich.
+Positional argument
+sequences
+Path to the FASTA file containing the sequences to be mutated, e.g., 'path/to/seqs.fa'.
+Sequence identifiers following the '>' character must correspond to the identifiers in the seq_ID column of mutations.
Example format of the FASTA file:
+>seq1 (or ENSG00000106443)
+ACTGCGATAGACT
+>seq2
+AGATCGCTAG
+Alternatively: Input sequence(s) as a string or list, e.g. 'AGCTAGCT'.
+NOTE: Only the letters until the first space or dot will be used as sequence identifiers - Version numbers of Ensembl IDs will be ignored.
+NOTE: When the sequences input is a genome fasta file, also see the gtf argument below.
Required arguments
+-m --mutations
+Path to the csv or tsv file (e.g., 'path/to/mutations.csv') or data frame (DataFrame object) containing information about the mutations in the following format (the 'notes' and 'mut_ID' columns are optional):
| mutation | mut_ID | seq_ID | notes |
|---|---|---|---|
| c.2C>T | mut1 | seq1 | -> Apply mutation 1 to sequence 1 |
| c.9_13inv | mut2 | seq2 | -> Apply mutation 2 to sequence 2 |
| c.9_13inv | mut2 | seq4 | -> Apply mutation 2 to sequence 4 |
| c.9_13delinsAAT | mut3 | seq4 | -> Apply mutation 3 to sequence 4 |
| ... | ... | ... |
'mutation' = Column containing the mutations to be performed written in standard mutation annotation
+'mut_ID' = Column containing the identifier for each mutation
+'seq_ID' = Column containing the identifiers of the sequences to be mutated (must correspond to the string following the '>' character in the 'sequences' FASTA file; do NOT include spaces or dots)
Alternatively: Input mutation(s) as a string or list, e.g., 'c.2C>T'.
+If a list is provided, the number of mutations must equal the number of input sequences.
For use from the terminal (bash): Enclose individual mutation annotations in quotation marks to prevent parsing errors.
+Optional input-related arguments
+-mc --mut_column
+Name of the column containing the mutations to be performed in mutations. Default: 'mutation'.
-sic --seq_id_column
+Name of the column containing the IDs of the sequences to be mutated in mutations. Default: 'seq_ID'.
-mic --mut_id_column
+Name of the column containing the IDs of each mutation in mutations. Default: Same as mut_column.
-gtf --gtf
+Path to a .gtf file. When providing a genome fasta file as input for 'sequences', you can provide a .gtf file here and the input sequences will be defined according to the transcript boundaries, e.g. 'path/to/genome_annotation.gtf'. Default: None
-gtic --gtf_transcript_id_column
+Column name in the input mutations file containing the transcript ID. In this case, column seq_id_column should contain the chromosome number.
+Required when gtf is provided. Default: None
Optional mutant sequence generation/filtering arguments
+-k --k
+Length of sequences flanking the mutation. Default: 30.
+If k > total length of the sequence, the entire sequence will be kept.
-msl --min_seq_len
+Minimum length of the mutant output sequence, e.g. 100. Mutant sequences smaller than this will be dropped. Default: None
-ma --max_ambiguous
+Maximum number of 'N' (or 'n') characters allowed in the output sequence, e.g. 10. Default: None (no ambiguous character filter will be applied)
Optional mutant sequence generation/filtering flags
+-ofr --optimize_flanking_regions
+Removes nucleotides from either end of the mutant sequence to ensure (when possible) that the mutant sequence does not contain any k-mers also found in the wildtype/input sequence.
-rswk --remove_seqs_with_wt_kmers
+Removes output sequences where at least one k-mer is also present in the wildtype/input sequence in the same region.
+When used with --optimize_flanking_regions, only sequences for which a wildtype k-mer is still present after optimization will be removed.
-mio --merge_identical_off
+Do not merge identical mutant sequences in the output (by default, identical sequences will be merged by concatenating the sequence headers for all identical sequences).
Optional arguments to generate additional output
+This output is activated using the --update_df flag and will be stored in a copy of the mutations DataFrame.
-udf_o --update_df_out
+Path to output csv file containing the updated DataFrame, e.g. 'path/to/mutations_updated.csv'. Only valid when used with --update_df.
+Default: None -> the new csv file will be saved in the same directory as the mutations DataFrame with appendix '_updated'
-ts --translate_start
+(int or str) The position in the input nucleotide sequence to start translating, e.g. 5. If a string is provided, it should correspond to a column name in mutations containing the open reading frame start positions for each sequence/mutation. Only valid when used with --translate.
+Default: translates from the beginning of each sequence
-te --translate_end
+(int or str) The position in the input nucleotide sequence to end translating, e.g. 35. If a string is provided, it should correspond to a column name in mutations containing the open reading frame end positions for each sequence/mutation. Only valid when used with --translate.
+Default: translates until the end of each sequence
Optional flags to modify additional output
+-udf --update_df
+Updates the input mutations DataFrame to include additional columns with the mutation type, wildtype nucleotide sequence, and mutant nucleotide sequence (only valid if mutations is a .csv or .tsv file).
-sfs --store_full_sequences
+Includes the complete wildtype and mutant sequences in the updated mutations DataFrame (not just the sub-sequence with k-length flanks). Only valid when used with --update_df.
-tr --translate
+Adds additional columns to the updated mutations DataFrame containing the wildtype and mutant amino acid sequences. Only valid when used with --store_full_sequences.
Optional general arguments
+-o --out
+Path to output FASTA file containing the mutated sequences, e.g., 'path/to/output_fasta.fa'.
+Default: None -> returns a list of the mutated sequences to standard out.
+The identifiers (following the '>') of the mutated sequences in the output FASTA will be '>[seq_ID]_[mut_ID]'.
Optional general flags
+-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
gget mutate ATCGCTAAGCT -m 'c.4G>T'
+# Python
+gget.mutate("ATCGCTAAGCT", "c.4G>T")
+→ Returns ATCTCTAAGCT.
+List of sequences with a mutation for each sequence provided in a list:
+gget mutate ATCGCTAAGCT TAGCTA -m 'c.4G>T' 'c.1_3inv' -o mut_fasta.fa
+# Python
+gget.mutate(["ATCGCTAAGCT", "TAGCTA"], ["c.4G>T", "c.1_3inv"], out="mut_fasta.fa")
+→ Saves 'mut_fasta.fa' file containing:
+>seq1_mut1
+ATCTCTAAGCT
+>seq2_mut2
+GATCTA
+One mutation applied to several sequences with adjusted k:
gget mutate ATCGCTAAGCT TAGCTA -m 'c.1_3inv' -k 3
+# Python
+gget.mutate(["ATCGCTAAGCT", "TAGCTA"], "c.1_3inv", k=3)
+→ Returns ['CTAGCT', 'GATCTA'].
+Add mutations to an entire genome with extended output
+Main input:
mutations CSV (by having seq_id_column contain chromosome information, and mut_column contain mutation information with respect to genome coordinates)sequences fileSince we are passing the path to a gtf file to the gtf argument, transcript boundaries will be respected (the genome will be split into transcripts). gtf_transcript_id_column specifies the name of the column in mutations containing the transcript IDs corresponding to the transcript IDs in the gtf file.
The optimize_flanking_regions argument maximizes the length of the resulting mutation-containing sequences while maintaining specificity (no wildtype k-mer will be retained).
update_df activates the creation of a new CSV file with updated information about each input and output sequence. This new CSV file will be saved as update_df_out. Since store_full_sequences is activated, this new CSV file will not only contain the output sequences (restricted in size by flanking regiong of size k), but also the complete input and output sequences. This allows us to observe the mutation in the context of the entire sequence. Lastly, we are also adding the translated versions of the complete sequences by adding the with the translate flag, so we can observe how the resulting amino acid sequence is changed. The translate_start and translate_end arguments specify the names of the columns in mutations that contain the start and end positions of the open reading frame (start and end positions for translating the nucleotide sequence to an amino acid sequence), respectively.
gget mutate \
+ -m mutations_input.csv \
+ -o mut_fasta.fa \
+ -k 4 \
+ -sic Chromosome \
+ -mic Mutation \
+ -gtf genome_annotation.gtf \
+ -gtic Ensembl_Transcript_ID \
+ -ofr \
+ -update_df \
+ -udf_o mutations_updated.csv \
+ -sfs \
+ -tr \
+ -ts Translate_Start \
+ -te Translate_End \
+ genome_reference.fa
+# Python
+gget.mutate(sequences="genome_reference.fa", mutations="mutations_input.csv", out="mut_fasta.fa", k=4, seq_id_column="Chromosome", mut_column="Mutation", gtf="genome_annotation.gtf", gtf_transcript_id_column="Ensembl_Transcript_ID", optimize_flanking_regions=True, update_df=True, update_df_out="mutations_updated.csv", store_full_sequences=True, translate=True, translate_start="Translate_Start", translate_end="Translate_End")
+→ Takes as input 'mutations_input.csv' file containing:
+| Chromosome | Mutation | Ensembl_Transcript_ID | Translate_Start | Translate_End |
+|------------|-------------------|------------------------|-----------------|---------------|
+| 1 | g.224411A>C | ENST00000193812 | 0 | 100 |
+| 8 | g.25111del | ENST00000174411 | 0 | 294 |
+| X | g.1011_1012insAA | ENST00000421914 | 9 | 1211 |
+→ Saves 'mut_fasta.fa' file containing:
+>1:g.224411A>C
+TGCTCTGCT
+>8:g.25111del
+GAGTCGAT
+>X:g.1011_1012insAA
+TTAGAACTT
+→ Saves 'mutations_updated.csv' file containing:
+
+| Chromosome | Mutation | Ensembl_Transcript_ID | mutation_type | wt_sequence | mutant_sequence | wt_sequence_full | mutant_sequence_full | wt_sequence_aa_full | mutant_sequence_aa_full |
+|------------|-------------------|------------------------|---------------|-------------|-----------------|-------------------|----------------------|---------------------|-------------------------|
+| 1 | g.224411A>C | ENSMUST00000193812 | Substitution | TGCTATGCT | TGCTCTGCT | ...TGCTATGCT... | ...TGCTCTGCT... | ...CYA... | ...CSA... |
+| 8 | g.25111del | ENST00000174411 | Deletion | GAGTCCGAT | GAGTCGAT | ...GAGTCCGAT... | ...GAGTCGAT... | ...ESD... | ...ES... |
+| X | g.1011_1012insAA | ENST00000421914 | Insertion | TTAGCTT | TTAGAACTT | ...TTAGCTT... | ...TTAGAACTT... | ...A... | ...EL... |
+
+If you use gget mutate in a publication, please cite the following articles:
++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Fetch associated diseases or drugs from OpenTargets using Ensembl IDs.
+Return format: JSON/CSV (command-line) or data frame (Python).
This module was written by Sam Wagenaar.
+Positional argument
+ens_id
+Ensembl gene ID, e.g ENSG00000169194.
Optional arguments
+-r --resource
+Defines the type of information to return in the output. Default: 'diseases'.
+Possible resources are:
| Resource | Return Value | Valid Filters | Sources |
|---|---|---|---|
diseases | Associated diseases | None | Various:etc. |
drugs | Associated drugs | disease_id | ChEMBL |
tractability | Tractability data | None | Open Targets |
pharmacogenetics | Pharmacogenetic responses | drug_id | PharmGKB |
expression | Gene expression data (by tissues, organs, and anatomical systems) | tissue_idanatomical_systemorgan | |
depmap | DepMap gene→disease-effect data. | tissue_id | DepMap Portal |
interactions | Protein⇄protein interactions | protein_a_idprotein_b_idgene_b_id |
-l --limit
+Limit the number of results, e.g 10. Default: No limit.
+Note: Not compatible with the tractability and depmap resources.
-o --out
+Path to the JSON file the results will be saved in, e.g. path/to/directory/results.json. Default: Standard out.
+Python: save=True will save the output in the current working directory.
Optional filter arguments
+-fd --filter_disease disease_id
+Filter by disease ID, e.g. 'EFO_0000274'. Only valid for the drugs resource.
-fc --filter_drug drug_id
+Filter by drug ID, e.g. 'CHEMBL1743081'. Only valid for the pharmacogenetics resource.
-ft --filter_tissue tissue_id
+Filter by tissue ID, e.g. 'UBERON_0000473'. Only valid for the expression and depmap resources.
-fa --filter_anat_sys
+Filter by anatomical system, e.g. 'nervous system'. Only valid for the expression resource.
-fo --filter_organ anatomical_system
+Filter by organ, e.g. 'brain'. Only valid for the expression resource.
-fpa --filter_protein_a protein_a_id
+Filter by the protein ID of the first protein in the interaction, e.g. 'ENSP00000304915'. Only valid for the interactions resource.
-fpb --filter_protein_b protein_b_id
+Filter by the protein ID of the second protein in the interaction, e.g. 'ENSP00000379111'. Only valid for the interactions resource.
-fgb --filter_gene_b gene_b_id
+Filter by the gene ID of the second protein in the interaction, e.g. 'ENSG00000077238'. Only valid for the interactions resource.
filters
+Python only. A dictionary of filters, e.g.
{'disease_id': ['EFO_0000274', 'HP_0000964']}
+filter_mode
+Python only. filter_mode='or' combines filters of different IDs with OR logic.
+filter_mode='and' combines filters of different IDs with AND logic (default).
Flags
+-csv --csv
+Command-line only. Returns the output in CSV format, instead of JSON format.
+Python: Use json=True to return output in JSON format.
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
-or --or
+Command-line only. Filters are combined with OR logic. Default: AND logic.
wrap_text
+Python only. wrap_text=True displays data frame with wrapped text for easy reading (default: False).
Get associated diseases for a specific gene:
+gget opentargets ENSG00000169194 -r diseases -l 1
+# Python
+import gget
+gget.opentargets('ENSG00000169194', resource='diseases', limit=1)
+→ Returns the top disease associated with the gene ENSG00000169194.
+| id | name | description | score |
|---|---|---|---|
| EFO_0000274 | atopic eczema | A chronic inflammatory genetically determined disease of the skin ... | 0.66364347241831 |
Get associated drugs for a specific gene:
+gget opentargets ENSG00000169194 -r drugs -l 2
+# Python
+import gget
+gget.opentargets('ENSG00000169194', resource='drugs', limit=2)
+→ Returns the top 2 drugs associated with the gene ENSG00000169194.
+| id | name | type | action_mechanism | description | synonyms | trade_names | disease_id | disease_name | trial_phase | trial_status | trial_ids | approved |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CHEMBL1743081 | TRALOKINUMAB | Antibody | Interleukin‑13 inhibitor | Antibody drug with a maximum clinical trial phase of IV ... | ['CAT-354', 'Tralokinumab'] | ['Adbry', 'Adtralza'] | EFO_0000274 | atopic eczema | 4 | [] | True | |
| CHEMBL4297864 | CENDAKIMAB | Antibody | Interleukin‑13 inhibitor | Antibody drug with a maximum clinical trial phase of III ... | [ABT-308, Abt-308, CC-93538, Cendakimab, RPC-4046] | [] | EFO_0004232 | eosinophilic esophagitis | 3 | Recruiting | [NCT04991935] | False |
Note: Returned trial_ids are ClinicalTrials.gov identifiers
Get tractability data for a specific gene:
+gget opentargets ENSG00000169194 -r tractability
+# Python
+import gget
+gget.opentargets('ENSG00000169194', resource='tractability')
+→ Returns tractability data for the gene ENSG00000169194.
+| label | modality |
|---|---|
| High-Quality Pocket | Small molecule |
| Approved Drug | Antibody |
| GO CC high conf | Antibody |
| UniProt loc med conf | Antibody |
| UniProt SigP or TMHMM | Antibody |
Get pharmacogenetic responses for a specific gene:
+gget opentargets ENSG00000169194 -r pharmacogenetics -l 1
+# Python
+import gget
+gget.opentargets('ENSG00000169194', resource='pharmacogenetics', limit=1)
+→ Returns pharmacogenetic responses for the gene ENSG00000169194.
+| rs_id | genotype_id | genotype | variant_consequence_id | variant_consequence_label | drugs | phenotype | genotype_annotation | response_category | direct_target | evidence_level | source | literature |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| rs1295686 | 5_132660151_T_T,T | TT | SO:0002073 | no_sequence_alteration | id name 0 None hepatitis vaccines | increased risk for non‑immune response to the hepatitis B vaccine | Patients with the TT genotype may be at increased risk for non-immune response to the hepatitis B vaccine... | efficacy | False | 3 | pharmgkb | [21111021] |
Note: Returned literature ids are Europe PMC identifiers
Get tissues where a gene is most expressed:
+gget opentargets ENSG00000169194 -r expression -l 2
+# Python
+import gget
+gget.opentargets('ENSG00000169194', resource='expression', limit=2)
+→ Returns the top 2 tissues where the gene ENSG00000169194 is most expressed.
+| tissue_id | tissue_name | rna_zscore | rna_value | rna_unit | rna_level | anatomical_systems | organs |
|---|---|---|---|---|---|---|---|
| UBERON_0000473 | testis | 5 | 1026 | 3 | [reproductive system] | [reproductive organ, reproductive structure] | |
| CL_0000542 | EBV‑transformed lymphocyte | 1 | 54 | 2 | [hemolymphoid system, immune system, lymphoid system] | [immune organ] |
Get DepMap gene-disease effect data for a specific gene:
+gget opentargets ENSG00000169194 -r depmap
+# Python
+import gget
+gget.opentargets('ENSG00000169194', resource='depmap')
+→ Returns DepMap gene-disease effect data for the gene ENSG00000169194.
+| depmap_id | expression | effect | tissue_id | tissue_name | cell_line_name | disease_cell_line_id | disease_name | mutation |
|---|---|---|---|---|---|---|---|---|
| ACH‑001532 | 0.176323 | 0.054950 | UBERON_0002113 | kidney | JMU-RTK-2 | None | Rhabdoid Cancer | None |
Get protein-protein interactions for a specific gene:
+gget opentargets ENSG00000169194 -r interactions -l 2
+# Python
+import gget
+gget.opentargets('ENSG00000169194', resource='interactions', limit=2)
+→ Returns the top 2 protein-protein interactions for the gene ENSG00000169194.
+| evidence_score | evidence_count | source_db | protein_a_id | gene_a_id | gene_a_symbol | role_a | taxon_a | protein_b_id | gene_b_id | gene_b_symbol | role_b | taxon_b |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0.999 | 3 | string | ENSP00000304915 | ENSG00000169194 | IL13 | unspecified role | 9606 | ENSP00000379111 | ENSG00000077238 | IL4R | unspecified role | 9606 |
| 0.999 | 3 | string | ENSP00000304915 | ENSG00000169194 | IL13 | unspecified role | 9606 | ENSP00000360730 | ENSG00000131724 | IL13RA1 | unspecified role | 9606 |
Get protein-protein interactions for a specific gene, filtering by protein and gene IDs:
+gget opentargets ENSG00000169194 -r interactions -fpa P35225 --filter_gene_b ENSG00000077238
+# Python
+import gget
+gget.opentargets('ENSG00000169194', resource='interactions', filters={'protein_a_id': 'P35225', 'gene_b_id': 'ENSG00000077238'})
+→ Returns protein-protein interactions for the gene ENSG00000169194, where the first protein is P35225 and the second gene is ENSG00000077238.
+| evidence_score | evidence_count | source_db | protein_a_id | gene_a_id | gene_a_symbol | role_a | taxon_a | protein_b_id | gene_b_id | gene_b_symbol | role_b | taxon_b |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| None | 3 | reactome | P35225 | ENSG00000169194 | IL13 | unspecified role | 9606 | P24394 | ENSG00000077238 | IL4R | unspecified role | 9606 |
| None | 2 | signor | P35225 | ENSG00000169194 | IL13 | regulator | 9606 | P24394 | ENSG00000077238 | IL4R | regulator target | 9606 |
Get protein-protein interactions for a specific gene, filtering by protein or gene IDs:
+gget opentargets ENSG00000169194 -r interactions -fpa P35225 --filter_gene_b ENSG00000077238 ENSG00000111537 --or -l 5
+# Python
+import gget
+gget.opentargets(
+ 'ENSG00000169194',
+ resource='interactions',
+ filters={'protein_a_id': 'P35225', 'gene_b_id': ['ENSG00000077238', 'ENSG00000111537']},
+ filter_mode='or',
+ limit=5
+)
+→ Returns protein-protein interactions for the gene ENSG00000169194, where the first protein is P35225 or the second gene is either ENSG00000077238 or ENSG00000111537.
+| evidence_score | evidence_count | source_db | protein_a_id | gene_a_id | gene_a_symbol | role_a | taxon_a | protein_b_id | gene_b_id | gene_b_symbol | role_b | taxon_b |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0.999 | 3 | string | ENSP00000304915 | ENSG00000169194 | IL13 | unspecified role | 9606 | ENSP00000379111 | ENSG00000077238 | IL4R | unspecified role | 9606 |
| 0.961 | 2 | string | ENSP00000304915 | ENSG00000169194 | IL13 | unspecified role | 9606 | ENSP00000229135 | ENSG00000111537 | IFNG | unspecified role | 9606 |
| 0.800 | 9 | intact | P35225 | ENSG00000169194 | IL13 | unspecified role | 9606 | Q14627 | ENSG00000123496 | IL13RA2 | unspecified role | 9606 |
| 0.740 | 6 | intact | P35225 | ENSG00000169194 | IL13 | unspecified role | 9606 | P78552 | ENSG00000131724 | IL13RA1 | unspecified role | 9606 |
| 0.400 | 1 | intact | P35225 | ENSG00000169194 | IL13 | unspecified role | 9606 | Q86XT9 | ENSG00000149932 | TMEM219 | stimulator | 9606 |
If you use gget opentargets in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Ochoa D, Hercules A, Carmona M, Suveges D, Baker J, Malangone C, Lopez I, Miranda A, Cruz-Castillo C, Fumis L, Bernal-Llinares M, Tsukanov K, Cornu H, Tsirigos K, Razuvayevskaya O, Buniello A, Schwartzentruber J, Karim M, Ariano B, Martinez Osorio RE, Ferrer J, Ge X, Machlitt-Northen S, Gonzalez-Uriarte A, Saha S, Tirunagari S, Mehta C, Roldán-Romero JM, Horswell S, Young S, Ghoussaini M, Hulcoop DG, Dunham I, McDonagh EM. The next-generation Open Targets Platform: reimagined, redesigned, rebuilt. Nucleic Acids Res. 2023 Jan 6;51(D1):D1353-D1359. doi: 10.1093/nar/gkac1046. PMID: 36399499; PMCID: PMC9825572.
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Query RCSB Protein Data Bank (PDB) for the protein structure/metadata of a given PDB ID.
+Return format: Resource 'pdb' is returned in PDB format. All other resources are returned in JSON format.
Positional argument
+pdb_id
+PDB ID to be queried, e.g. '7S7U'.
Optional arguments
+-r --resource
+Defines type of information to be returned. One of the following:
+'pdb': Returns the protein structure in PDB format (default).
+'entry': Information about PDB structures at the top level of PDB structure hierarchical data organization.
+'pubmed': Get PubMed annotations (data integrated from PubMed) for a given entry's primary citation.
+'assembly': Information about PDB structures at the quaternary structure level.
+'branched_entity': Get branched entity description (define entity ID as 'identifier').
+'nonpolymer_entity': Get non-polymer entity data (define entity ID as 'identifier').
+'polymer_entity': Get polymer entity data (define entity ID as 'identifier').
+'uniprot': Get UniProt annotations for a given macromolecular entity (define entity ID as 'identifier').
+'branched_entity_instance': Get branched entity instance description (define chain ID as 'identifier').
+'polymer_entity_instance': Get polymer entity instance (a.k.a chain) data (define chain ID as 'identifier').
+'nonpolymer_entity_instance': Get non-polymer entity instance description (define chain ID as 'identifier').
-i --identifier
+Can be used to define assembly, entity or chain ID (default: None). Assembly/entity IDs are numbers (e.g. 1), and chain IDs are letters (e.g. 'A').
-o --out
+Path to the file the results will be saved in, e.g. path/to/directory/7S7U.pdb or path/to/directory/7S7U_entry.json. Default: Standard out.
+Python: save=True will save the output in the current working directory.
gget pdb 7S7U -o 7S7U.pdb
+# Python
+gget.pdb("7S7U", save=True)
+→ Saves the structure of 7S7U in PDB format as '7S7U.pdb' in the current working directory.
+Find PDB crystal structures for a comparative analysis of protein structure:
+# Find PDB IDs associated with an Ensembl ID
+gget info ENSG00000130234
+
+# Alternatively: Since many entries in the PDB do not have linked Ensembl IDs,
+# you will likely find more PDB entries by BLASTing the sequence agains the PDB.
+
+# Get the amino acid sequence of a transcript from an Ensembl ID
+gget seq --translate ENSG00000130234 -o gget_seq_results.fa
+
+# BLAST an amino acid sequence to find similar structures in the PDB
+gget blast --database pdbaa gget_seq_results.fa
+
+# Get PDB files from the PDB IDs returned by gget blast for comparative analysis
+gget pdb 7DQA -o 7DQA.pdb
+gget pdb 7CT5 -o 7CT5.pdb
+# Find PDB IDs associated with an Ensembl ID
+gget.info("ENSG00000130234")
+
+# Alternatively: Since many entries in the PDB do not have linked Ensembl IDs,
+# you will likely find more PDB entries by BLASTing the sequence agains the PDB.
+
+# Get the amino acid sequence of a transcript from an Ensembl ID
+gget.seq("ENSG00000130234", translate=True, save=True)
+
+# BLAST an amino acid sequence to find similar structures in the PDB
+gget.blast("gget_seq_results.fa", database="pdbaa")
+
+# Get PDB files from the PDB IDs returned by gget blast for comparative analysis
+gget.pdb("7DQA", save=True)
+gget.pdb("7CT5", save=True)
+→ The use case above exemplifies how to find PDB files for comparative analysis of protein structure starting with Ensembl IDs or amino acid sequences. The fetched PDB files can also be compared to predicted structures generated by gget alphafold. PDB files can be viewed interactively in 3D online, or using programs like PyMOL or Blender. To compare two PDB files, you can use this website.
If you use gget pdb in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Berman HM, Westbrook J, Feng Z, Gilliland G, Bhat TN, Weissig H, Shindyalov IN, Bourne PE. The Protein Data Bank. Nucleic Acids Res. 2000 Jan 1;28(1):235-42. doi: 10.1093/nar/28.1.235. PMID: 10592235; PMCID: PMC102472.
+Command line:
+# Fetch all Homo sapiens reference and annotation FTPs from the latest Ensembl release
+$ gget ref homo_sapiens
+
+# Get Ensembl IDs of human genes with "ace2" or "angiotensin converting enzyme 2" in their name/description
+$ gget search -s homo_sapiens 'ace2' 'angiotensin converting enzyme 2'
+
+# Look up gene ENSG00000130234 (ACE2) and its transcript ENST00000252519
+$ gget info ENSG00000130234 ENST00000252519
+
+# Fetch the amino acid sequence of the canonical transcript of gene ENSG00000130234
+$ gget seq --translate ENSG00000130234
+
+# Quickly find the genomic location of (the start of) that amino acid sequence
+$ gget blat MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS
+
+# BLAST (the start of) that amino acid sequence
+$ gget blast MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS
+
+# Align multiple nucleotide or amino acid sequences against each other (also accepts path to FASTA file)
+$ gget muscle MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS
+
+# Align one or more amino acid sequences against a reference (containing one or more sequences) (local BLAST) (also accepts paths to FASTA files)
+$ gget diamond MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS -ref MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS
+
+# Use Enrichr for an ontology analysis of a list of genes
+$ gget enrichr -db ontology ACE2 AGT AGTR1 ACE AGTRAP AGTR2 ACE3P
+
+# Get the human tissue expression of gene ACE2
+$ gget archs4 -w tissue ACE2
+
+# Get the protein structure (in PDB format) of ACE2 as stored in the Protein Data Bank (PDB ID returned by gget info)
+$ gget pdb 1R42 -o 1R42.pdb
+
+# Find Eukaryotic Linear Motifs (ELMs) in a protein sequence
+$ gget setup elm # setup only needs to be run once
+$ gget elm -o results MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS
+
+# Fetch a scRNAseq count matrix (AnnData format) based on specified gene(s), tissue(s), and cell type(s) (default species: human)
+$ gget setup cellxgene # setup only needs to be run once
+$ gget cellxgene --gene ACE2 SLC5A1 --tissue lung --cell_type 'mucus secreting cell' -o example_adata.h5ad
+
+# Predict the protein structure of GFP from its amino acid sequence
+$ gget setup alphafold # setup only needs to be run once
+$ gget alphafold MSKGEELFTGVVPILVELDGDVNGHKFSVSGEGEGDATYGKLTLKFICTTGKLPVPWPTLVTTFSYGVQCFSRYPDHMKQHDFFKSAMPEGYVQERTIFFKDDGNYKTRAEVKFEGDTLVNRIELKGIDFKEDGNILGHKLEYNYNSHNVYIMADKQKNGIKVNFKIRHNIEDGSVQLADHYQQNTPIGDGPVLLPDNHYLSTQSALSKDPNEKRDHMVLLEFVTAAGITHGMDELYK
+Python (Jupyter Lab / Google Colab):
+import gget
+gget.ref("homo_sapiens")
+gget.search(["ace2", "angiotensin converting enzyme 2"], "homo_sapiens")
+gget.info(["ENSG00000130234", "ENST00000252519"])
+gget.seq("ENSG00000130234", translate=True)
+gget.blat("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS")
+gget.blast("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS")
+gget.muscle(["MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS", "MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS"])
+gget.diamond("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS", reference="MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS")
+gget.enrichr(["ACE2", "AGT", "AGTR1", "ACE", "AGTRAP", "AGTR2", "ACE3P"], database="ontology", plot=True)
+gget.archs4("ACE2", which="tissue")
+gget.pdb("1R42", save=True)
+
+gget.setup("elm") # setup only needs to be run once
+ortho_df, regex_df = gget.elm("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS")
+
+gget.setup("cellxgene") # setup only needs to be run once
+gget.cellxgene(gene = ["ACE2", "SLC5A1"], tissue = "lung", cell_type = "mucus secreting cell")
+
+gget.setup("alphafold") # setup only needs to be run once
+gget.alphafold("MSKGEELFTGVVPILVELDGDVNGHKFSVSGEGEGDATYGKLTLKFICTTGKLPVPWPTLVTTFSYGVQCFSRYPDHMKQHDFFKSAMPEGYVQERTIFFKDDGNYKTRAEVKFEGDTLVNRIELKGIDFKEDGNILGHKLEYNYNSHNVYIMADKQKNGIKVNFKIRHNIEDGSVQLADHYQQNTPIGDGPVLLPDNHYLSTQSALSKDPNEKRDHMVLLEFVTAAGITHGMDELYK")
+Call gget from R using reticulate:
system("pip install gget")
+install.packages("reticulate")
+library(reticulate)
+gget <- import("gget")
+
+gget$ref("homo_sapiens")
+gget$search(list("ace2", "angiotensin converting enzyme 2"), "homo_sapiens")
+gget$info(list("ENSG00000130234", "ENST00000252519"))
+gget$seq("ENSG00000130234", translate=TRUE)
+gget$blat("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS")
+gget$blast("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS")
+gget$muscle(list("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS", "MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS"), out="out.afa")
+gget$diamond("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS", reference="MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS")
+gget$enrichr(list("ACE2", "AGT", "AGTR1", "ACE", "AGTRAP", "AGTR2", "ACE3P"), database="ontology")
+gget$archs4("ACE2", which="tissue")
+gget$pdb("1R42", save=TRUE)
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Fetch FTPs and their respective metadata (or use flag ftp to only return the links) for reference genomes and annotations from Ensembl by species.
+Return format: dictionary/JSON.
Positional argument
+species
+Species for which the FTPs will be fetched in the format genus_species, e.g. homo_sapiens.
+Supports all available vertebrate and invertebrate (plants, fungi, protists, and invertebrate metazoa) genomes from Ensembl, except bacteria.
+Note: Not required when using flags --list_species or --list_iv_species.
+Supported shortcuts: 'human', 'mouse', 'human_grch37' (accesses the GRCh37 genome assembly)
Optional arguments
+-w --which
+Defines which results to return. Default: 'all' -> Returns all available results.
+Possible entries are one or a combination (as comma-separated list) of the following:
+'gtf' - Returns the annotation (GTF).
+'cdna' - Returns the trancriptome (cDNA).
+'dna' - Returns the genome (DNA).
+'cds' - Returns the coding sequences corresponding to Ensembl genes. (Does not contain UTR or intronic sequence.)
+'cdrna' - Returns transcript sequences corresponding to non-coding RNA genes (ncRNA).
+'pep' - Returns the protein translations of Ensembl genes.
-r --release
+Defines the Ensembl release number from which the files are fetched, e.g. 104. Default: latest Ensembl release.
-od --out_dir
+Path to the directory where the FTPs will be saved, e.g. path/to/directory/. Default: Current working directory.
-o --out
+Path to the JSON file the results will be saved in, e.g. path/to/directory/results.json. Default: Standard out.
+Python: save=True will save the output in the current working directory.
Flags
+-l --list_species
+Lists all available vertebrate species. (Python: combine with species=None.)
-liv --list_iv_species
+Lists all available invertebrate species. (Python: combine with species=None.)
-ftp --ftp
+Returns only the requested FTP links.
-d --download
+Command-line only. Downloads the requested FTPs to the directory specified by out_dir (requires curl to be installed).
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
Use gget ref in combination with kallisto | bustools to build a reference index:
kb ref -i INDEX -g T2G -f1 FASTA $(gget ref --ftp -w dna,gtf homo_sapiens)
+→ kb ref builds a reference index using the latest DNA and GTF files of species Homo sapiens passed to it by gget ref.
List all available genomes from Ensembl release 103:
+gget ref --list_species -r 103
+# Python
+gget.ref(species=None, list_species=True, release=103)
+→ Returns a list with all available genomes (checks if GTF and FASTAs are available) from Ensembl release 103.
+(If no release is specified, gget ref will always return information from the latest Ensembl release.)
Get the genome reference for a specific species:
+gget ref -w gtf,dna homo_sapiens
+# Python
+gget.ref("homo_sapiens", which=["gtf", "dna"])
+→ Returns a JSON with the latest human GTF and FASTA FTPs, and their respective metadata, in the format:
+{
+ "homo_sapiens": {
+ "annotation_gtf": {
+ "ftp": "http://ftp.ensembl.org/pub/release-106/gtf/homo_sapiens/Homo_sapiens.GRCh38.106.gtf.gz",
+ "ensembl_release": 106,
+ "release_date": "28-Feb-2022",
+ "release_time": "23:27",
+ "bytes": "51379459"
+ },
+ "genome_dna": {
+ "ftp": "http://ftp.ensembl.org/pub/release-106/fasta/homo_sapiens/dna/Homo_sapiens.GRCh38.dna.primary_assembly.fa.gz",
+ "ensembl_release": 106,
+ "release_date": "21-Feb-2022",
+ "release_time": "09:35",
+ "bytes": "881211416"
+ }
+ }
+}
+If you use gget ref in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Martin FJ, Amode MR, Aneja A, Austine-Orimoloye O, Azov AG, Barnes I, Becker A, Bennett R, Berry A, Bhai J, Bhurji SK, Bignell A, Boddu S, Branco Lins PR, Brooks L, Ramaraju SB, Charkhchi M, Cockburn A, Da Rin Fiorretto L, Davidson C, Dodiya K, Donaldson S, El Houdaigui B, El Naboulsi T, Fatima R, Giron CG, Genez T, Ghattaoraya GS, Martinez JG, Guijarro C, Hardy M, Hollis Z, Hourlier T, Hunt T, Kay M, Kaykala V, Le T, Lemos D, Marques-Coelho D, Marugán JC, Merino GA, Mirabueno LP, Mushtaq A, Hossain SN, Ogeh DN, Sakthivel MP, Parker A, Perry M, Piližota I, Prosovetskaia I, Pérez-Silva JG, Salam AIA, Saraiva-Agostinho N, Schuilenburg H, Sheppard D, Sinha S, Sipos B, Stark W, Steed E, Sukumaran R, Sumathipala D, Suner MM, Surapaneni L, Sutinen K, Szpak M, Tricomi FF, Urbina-Gómez D, Veidenberg A, Walsh TA, Walts B, Wass E, Willhoft N, Allen J, Alvarez-Jarreta J, Chakiachvili M, Flint B, Giorgetti S, Haggerty L, Ilsley GR, Loveland JE, Moore B, Mudge JM, Tate J, Thybert D, Trevanion SJ, Winterbottom A, Frankish A, Hunt SE, Ruffier M, Cunningham F, Dyer S, Finn RD, Howe KL, Harrison PW, Yates AD, Flicek P. Ensembl 2023. Nucleic Acids Res. 2023 Jan 6;51(D1):D933-D941. doi: 10.1093/nar/gkac958. PMID: 36318249; PMCID: PMC9825606.
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Fetch genes and transcripts from Ensembl using free-form search terms.
+Results are matched based on the "gene name" and "description" sections in the Ensembl database. gget version >= 0.27.9 also includes results that match the Ensembl "synonym" section.
+Return format: JSON (command-line) or data frame/CSV (Python).
Positional argument
+searchwords
+One or more free form search words, e.g. gaba nmda. (Note: Search is not case-sensitive.)
Other required arguments
+-s --species
+Species or database to be searched.
+A species can be passed in the format 'genus_species', e.g. 'homo_sapiens' or 'arabidopsis_thaliana'.
+To pass a specific database, pass the name of the CORE database, e.g. 'mus_musculus_dba2j_core_105_1'.
All available core databases can be found here:
+Vertebrates: http://ftp.ensembl.org/pub/current/mysql/
+Invertebrates: http://ftp.ensemblgenomes.org/pub/current/ + select kingdom + go to mysql/
Supported shortcuts: 'human', 'mouse'
+Optional arguments
+-r --release
+Defines the Ensembl release number from which the files are fetched, e.g. 104. Default: None -> latest Ensembl release is used.
Note: The release argument does not apply to invertebrate species (you can pass a specific core database (which includes a release number) to the species argument instead). For invertebrate species, Ensembl only stores databases from 10 releases prior to the current release.
This argument is overwritten if a specific database (which includes a release number) is passed to the species argument.
+-t --id_type
+'gene' (default) or 'transcript'
+Returns genes or transcripts, respectively.
-ao --andor
+'or' (default) or 'and'
+'or': Returns all genes that INCLUDE AT LEAST ONE of the searchwords in their name/description.
+'and': Returns only genes that INCLUDE ALL of the searchwords in their name/description.
-l --limit
+Limits the number of search results, e.g. 10. Default: None.
-o --out
+Path to the csv the results will be saved in, e.g. path/to/directory/results.csv (or .json). Default: Standard out.
+Python: save=True will save the output in the current working directory.
Flags
+-csv --csv
+Command-line only. Returns results in CSV format.
+Python: Use json=True to return output in JSON format.
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
wrap_text
+Python only. wrap_text=True displays data frame with wrapped text for easy reading (default: False).
gget search -s human gaba gamma-aminobutyric
+# Python
+gget.search(["gaba", "gamma-aminobutyric"], "homo_sapiens")
+→ Returns all genes that contain at least one of the search words in their name or Ensembl/external reference description:
+| ensembl_id | gene_name | ensembl_description | ext_ref_description | biotype | url |
|---|---|---|---|---|---|
| ENSG00000034713 | GABARAPL2 | GABA type A receptor associated protein like 2 [Source:HGNC Symbol;Acc:HGNC:13291] | GABA type A receptor associated protein like 2 | protein_coding | https://uswest.ensembl.org/homo_sapiens/Gene/Summary?g=ENSG00000034713 |
| . . . | . . . | . . . | . . . | . . . | . . . |
If you use gget search in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Martin FJ, Amode MR, Aneja A, Austine-Orimoloye O, Azov AG, Barnes I, Becker A, Bennett R, Berry A, Bhai J, Bhurji SK, Bignell A, Boddu S, Branco Lins PR, Brooks L, Ramaraju SB, Charkhchi M, Cockburn A, Da Rin Fiorretto L, Davidson C, Dodiya K, Donaldson S, El Houdaigui B, El Naboulsi T, Fatima R, Giron CG, Genez T, Ghattaoraya GS, Martinez JG, Guijarro C, Hardy M, Hollis Z, Hourlier T, Hunt T, Kay M, Kaykala V, Le T, Lemos D, Marques-Coelho D, Marugán JC, Merino GA, Mirabueno LP, Mushtaq A, Hossain SN, Ogeh DN, Sakthivel MP, Parker A, Perry M, Piližota I, Prosovetskaia I, Pérez-Silva JG, Salam AIA, Saraiva-Agostinho N, Schuilenburg H, Sheppard D, Sinha S, Sipos B, Stark W, Steed E, Sukumaran R, Sumathipala D, Suner MM, Surapaneni L, Sutinen K, Szpak M, Tricomi FF, Urbina-Gómez D, Veidenberg A, Walsh TA, Walts B, Wass E, Willhoft N, Allen J, Alvarez-Jarreta J, Chakiachvili M, Flint B, Giorgetti S, Haggerty L, Ilsley GR, Loveland JE, Moore B, Mudge JM, Tate J, Thybert D, Trevanion SJ, Winterbottom A, Frankish A, Hunt SE, Ruffier M, Cunningham F, Dyer S, Finn RD, Howe KL, Harrison PW, Yates AD, Flicek P. Ensembl 2023. Nucleic Acids Res. 2023 Jan 6;51(D1):D933-D941. doi: 10.1093/nar/gkac958. PMID: 36318249; PMCID: PMC9825606.
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Fetch nucleotide or amino acid sequence(s) of a gene (and all its isoforms) or a transcript by Ensembl ID.
+Return format: FASTA.
Positional argument
+ens_ids
+One or more Ensembl IDs.
Optional arguments
+-o --out
+Path to the file the results will be saved in, e.g. path/to/directory/results.fa. Default: Standard out.
+Python: save=True will save the output in the current working directory.
Flags
+-t --translate
+Returns amino acid (instead of nucleotide) sequences.
+Nucleotide sequences are fetched from Ensembl.
+Amino acid sequences are fetched from UniProt.
-iso --isoforms
+Returns the sequences of all known transcripts.
+(Only for gene IDs.)
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
gget seq ENSG00000034713 ENSG00000104853 ENSG00000170296
+# Python
+gget.seq(["ENSG00000034713", "ENSG00000104853", "ENSG00000170296"])
+→ Returns the nucleotide sequences of ENSG00000034713, ENSG00000104853, and ENSG00000170296 in FASTA format.
+gget seq -t -iso ENSG00000034713
+# Python
+gget.seq("ENSG00000034713", translate=True, isoforms=True)
+→ Returns the amino acid sequences of all known transcripts of ENSG00000034713 in FASTA format.
+If you use gget seq in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Martin FJ, Amode MR, Aneja A, Austine-Orimoloye O, Azov AG, Barnes I, Becker A, Bennett R, Berry A, Bhai J, Bhurji SK, Bignell A, Boddu S, Branco Lins PR, Brooks L, Ramaraju SB, Charkhchi M, Cockburn A, Da Rin Fiorretto L, Davidson C, Dodiya K, Donaldson S, El Houdaigui B, El Naboulsi T, Fatima R, Giron CG, Genez T, Ghattaoraya GS, Martinez JG, Guijarro C, Hardy M, Hollis Z, Hourlier T, Hunt T, Kay M, Kaykala V, Le T, Lemos D, Marques-Coelho D, Marugán JC, Merino GA, Mirabueno LP, Mushtaq A, Hossain SN, Ogeh DN, Sakthivel MP, Parker A, Perry M, Piližota I, Prosovetskaia I, Pérez-Silva JG, Salam AIA, Saraiva-Agostinho N, Schuilenburg H, Sheppard D, Sinha S, Sipos B, Stark W, Steed E, Sukumaran R, Sumathipala D, Suner MM, Surapaneni L, Sutinen K, Szpak M, Tricomi FF, Urbina-Gómez D, Veidenberg A, Walsh TA, Walts B, Wass E, Willhoft N, Allen J, Alvarez-Jarreta J, Chakiachvili M, Flint B, Giorgetti S, Haggerty L, Ilsley GR, Loveland JE, Moore B, Mudge JM, Tate J, Thybert D, Trevanion SJ, Winterbottom A, Frankish A, Hunt SE, Ruffier M, Cunningham F, Dyer S, Finn RD, Howe KL, Harrison PW, Yates AD, Flicek P. Ensembl 2023. Nucleic Acids Res. 2023 Jan 6;51(D1):D933-D941. doi: 10.1093/nar/gkac958. PMID: 36318249; PMCID: PMC9825606
+The UniProt Consortium , UniProt: the Universal Protein Knowledgebase in 2023, Nucleic Acids Research, Volume 51, Issue D1, 6 January 2023, Pages D523–D531, https://doi.org/10.1093/nar/gkac1052
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Function to install/download third-party dependencies for a specified gget module.
+Positional argument
+module
+gget module for which dependencies should be installed.
Optional arguments
+-o --out
+Path to the folder downloaded files will be saved in (currently only applies to module = 'elm').
+NOTE: Do NOT use this argument when downloading the files for use with gget.elm.
+Default: None (downloaded files are saved inside the gget package installation folder).
Flags
+-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
gget setup alphafold
+# Python
+gget.setup("alphafold")
+→ Installs all (modified) third-party dependencies and downloads model parameters (~4GB) required to run gget alphafold.
Version ≥ 0.29.0 (Sep 25, 2024):
+gget enrichr now also supports species other than human and mouse (fly, yeast, worm, and fish) via modEnrichRgget mutate:gget mutate will now merge identical sequences in the final file by default. Mutation creation was vectorized to decrease runtime. Improved flanking sequence check for non-substitution mutations to make sure no wildtype kmer is retained in the mutation-containing sequence. Addition of several new arguments to customize sequence generation and output.gget cosmic:gget ref:gget info and gget seq:Version ≥ 0.28.6 (Jun 2, 2024):
+gget mutategget cosmic: You can now download entire COSMIC databases using the argument download_cosmic argumentgget ref: Can now fetch the GRCh37 genome assembly using species='human_grch37'gget search: Adjust access of human data to the structure of Ensembl release 112 (fixes issue 129)Version ≥ 0.28.5 (May 29, 2024):
gget.setup("alphafold") + inversion mutations in gget mutate only reverse the string instead of also computing the complementary strandVersion ≥ 0.28.4 (January 31, 2024):
+gget setup: Fix bug with filepath when running gget.setup("elm") on Windows OS.Version ≥ 0.28.3 (January 22, 2024):
+gget search and gget ref now also support fungi 🍄, protists 🌝, and invertebrate metazoa 🐝 🐜 🐌 🐙 (in addition to vertebrates and plants)gget cosmicgget enrichr: Fix duplicate scatter dots in plot when pathway names are duplicatedgget elm:
+gget modules.gget setup: Use the out argument to specify a directory the ELM database will be downloaded into. Completes this feature request.gget diamond: The DIAMOND command is now run with --ignore-warnings flag, allowing niche sequences such as amino acid sequences that only contain nucleotide characters and repeated sequences. This is also true for DIAMOND alignments performed within gget elm.gget ref and gget search back-end change: the current Ensembl release is fetched from the new release file on the Ensembl FTP site to avoid errors during uploads of new releases.gget search:
+--ftp) are saved in txt file format instead of json.gget ref:
+list_iv_species to list all available invertebrate species (can be combined with the release argument to fetch all species available from a specific Ensembl release)Version ≥ 0.28.2 (November 15, 2023):
+gget info: Return a logging error message when the NCBI server fails for a reason other than a fetch fail (this is an error on the server side rather than an error with gget)BeautifulSoupgget elm: Remove false positive and true negative instances from returned resultsgget elm: Add expand argumentVersion ≥ 0.28.0 (November 5, 2023):
+gget muscle to add a tutorial on how to visualize sequences with varying sequence name lengths + slight change to returned visualization so it's a bit more robust to varying sequence namesgget muscle now also allows a list of sequences as input (as an alternative to providing the path to a FASTA file)gget cellxgene (fixes bug)gget seq: Allow missing gene names (fixes https://github.com/pachterlab/gget/issues/107)gget enrichr: Use new arguments kegg_out and kegg_rank to create an image of the KEGG pathway with the genes from the enrichment analysis highlighted (thanks to this PR by Noriaki Sato)gget elm and gget diamondVersion ≥ 0.27.9 (August 7, 2023):
+gget enrichr: Use new argument background_list to provide a list of background genesgget search now also searches Ensembl synonyms (in addition to gene descriptions and names) to return more comprehensive search results (thanks to Samuel Klein for the suggestion)Version ≥ 0.27.8 (July 12, 2023):
+gget search: Specify the Ensembl release from which information is fetched with new argument -r --releasegget pdb (this bug was introduced in version 0.27.5)Version ≥ 0.27.7 (May 15, 2023):
+gget gpt and gget cellxgene from automatically installed requirements to gget setup.gget alphafold dependencies for compatibility with Python >= 3.10.census_version argument to gget cellxgene.Version ≥ 0.27.6 (May 1, 2023) (YANKED due to problems with dependencies -> replaced with version 0.27.7):
+gget search can now also query plant 🌱 Ensembl IDs.gget cellxgeneVersion ≥ 0.27.5 (April 6, 2023):
+gget search to function correctly with new Pandas version 2.0.0 (released on April 3rd, 2023) as well as older versions of Pandasgget info with new flags uniprot and ncbi which allow turning off results from these databases independently to save runtime (note: flag ensembl_only was deprecated)-q / --quiet (Python: verbose=False) flag to turn off progress informationVersion ≥ 0.27.4 (March 19, 2023):
+gget gptVersion ≥ 0.27.3 (March 11, 2023):
+gget info excludes PDB IDs by default to increase speed (PDB results can be included using flag --pdb / pdb=True).Version ≥ 0.27.2 (January 1, 2023):
+gget alphafold to DeepMind's AlphaFold v2.3.0 (including new arguments multimer_for_monomer and multimer_recycles)Version ≥ 0.27.0 (December 10, 2022):
+gget alphafold to match recent changes by DeepMindVersion ≥ 0.3.13 (November 11, 2022):
+gget enrichr and gget archs4 when used with Ensembl IDsVersion ≥ 0.3.12 (November 10, 2022):
+gget info now also returns subcellular localisation data from UniProtgget info flag ensembl_only returns only Ensembl resultsgget info and gget seqVersion ≥ 0.3.11 (September 7, 2022):
+gget pdbVersion ≥ 0.3.10 (September 2, 2022):
+gget alphafold now also returns pLDDT values for generating plots from output without rerunning the program (also see the gget alphafold FAQ)Version ≥ 0.3.9 (August 25, 2022):
+gget alphafoldVersion ≥ 0.3.8 (August 12, 2022):
+Version ≥ 0.3.7 (August 9, 2022):
+gget version ≥ 0.3.7 if you use gget refVersion ≥ 0.3.5 (August 6, 2022):
+gget alphafoldVersion ≥ 0.2.6 (July 7, 2022):
+gget ref now supports plant genomes! 🌱Version ≥ 0.2.5 (June 30, 2022):
+gget version ≥ 0.2.5 if you use any of the modules querying data from UniProt (gget info and gget seq).Version ≥ 0.2.3: (June 26, 2022):
+[-csv][--csv]). Data frame/CSV remains the default output for Jupyter Lab / Google Colab (and can be converted to JSON with json=True).gget ref -s human → gget ref human.gget info: [--expand] is deprecated. The module will now always return all of the available information.gget info, including the return of versioned Ensembl IDs.gget info and gget seq now support 🪱 WormBase and 🪰 FlyBase IDs.gget archs4 and gget enrichr now also take Ensembl IDs as input with added flag [-e][--ensembl] (ensembl=True in Jupyter Lab / Google Colab).gget seq argument seqtype was replaced by flag [-t][--translate] (translate=True/False in Jupyter Lab / Google Colab) which will return either nucleotide (False) or amino acid (True) sequences.gget search argument seqtype was renamed to id_type for clarity (still taking the same arguments 'gene' or 'transcript').++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Predice la estructura en 3D de cualquier proteína derivada de su secuencia de aminoácidos usando una versión simplificada del algoritmo AlphaFold2 de DeepMind, originalmente producido y publicado para AlphaFold Colab.
+Resultado: Predicción de la estructura (en formato PDB) y el errór de alineación (en formato json).
Antes de usar gget alphafold por primera vez:
conda install -qy conda==4.13.0 && conda install -qy -c conda-forge openmm=7.5.1openmm=7.7.0 para Python >= 3.10)conda update -qy conda para actualizar conda a la última versión.gget setup alphafold / gget.setup("alphafold") (ver también gget setup). Al ejecutar gget setup alphafold / gget.setup("alphafold") se descargará e instalará la última versión de AlphaFold2 alojada en el AlphaFold GitHub Repo. Puede volver a ejecutar este comando en cualquier momento para actualizar el software cuando hay una nueva versión de AlphaFold.Parámetro posicional
+sequence
+Secuencia de aminoácidos (str), o una lista de secuencias (gget alphafold automaticamente usa el algoritmo del multímero si múltiples secuencias son ingresadas), o una ruta a un archivo formato FASTA.
Parámetros optionales
+-mr --multimer_recycles
+El algoritmo de multímero se reciclara hasta que las predicciones dejen de cambiar, el limite de ciclos esta indicado aqui. Por defecto: 3
+Para obtener más exactitud, ajusta este limite a 20 (al costo de ejecuciones mas tardadas).
-o --out
+Ruta a la carpeta para guardar los resultados de la predicción (str). Por defecto: "./[fecha_tiempo]_gget_alphafold_prediction".
Banderas
+-mfm --multimer_for_monomer
+Usa el algoritmo de multímero para un monómero.
-r --relax
+Relaja el mejor modelo con el algoritmo AMBER.
-q --quiet
+Uso limitado para Terminal. Impide la información de progreso de ser exhibida durante la ejecución del programa.
+Para Python, usa verbose=False.
plot
+Solo para Python. plot=True provée una visualización interactiva de la predicción con el errór de alineación en 3D con py3Dmol y matplotlib (por defecto: True).
show_sidechains
+Solo para Python. show_sidechains=True incluye las cadenas laterales de proteínas en el esquema (por defecto: True).
# Predice la estructura de una proteína derivada de su secuencia de aminoácidos
+gget alphafold MAAHKGAEHHHKAAEHHEQAAKHHHAAAEHHEKGEHEQAAHHADTAYAHHKHAEEHAAQAAKHDAEHHAPKPH
+
+# Encuentra secuencias similares previamente depositadas en el PDB para análisis comparativo
+gget blast --database pdbaa MAAHKGAEHHHKAAEHHEQAAKHHHAAAEHHEKGEHEQAAHHADTAYAHHKHAEEHAAQAAKHDAEHHAPKPH
+
+# Busca los archivos PDB de estructuras similares resultantes de gget blast para comparar y obtener una medida de calidad del modelo predecido.
+gget pdb 3UQ3 -o 3UQ3.pdb
+gget pdb 2K42 -o 2K42.pdb
+# Python
+# Predice la estructura de una proteína derivada de su secuencia de aminoácidos
+gget.alphafold("MAAHKGAEHHHKAAEHHEQAAKHHHAAAEHHEKGEHEQAAHHADTAYAHHKHAEEHAAQAAKHDAEHHAPKPH")
+
+# Encuentra secuencias similares previamente depositadas en el PDB para análisis comparativo
+gget.blast("MAAHKGAEHHHKAAEHHEQAAKHHHAAAEHHEKGEHEQAAHHADTAYAHHKHAEEHAAQAAKHDAEHHAPKPH", database="pdbaa")
+
+# Busca los archivos PDB de estructuras similares resultantes de gget blast para comparar y obtener una medida de calidad del modelo predecido.
+gget.pdb("3UQ3", save=True)
+gget.pdb("2K42", save=True)
+→ gget alphafold produce la estructura predecida (en formato PDB) y el errór de alineación (en formato json) en una nueva carpeta ("./[fecha_tiempo]_gget_alphafold_prediction"). Este ejemplo demuestra como usar gget blast y gget pdb para correr un análisis comparativo. Los archivos PDB se pueden ver en 3D con RCSB 3D view, o usando programas como PyMOL o Blender. Para comparar múltiples archivos PDB, use RCSB alignment. Python también produce esquemas interactivos, los cuales se pueden generar de los archivos PDB y JSON, como es describido en gget alphafold FAQ Q4.
Si utiliza gget alphafold en una publicación, favor de citar los siguientes artículos:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Jumper, J., Evans, R., Pritzel, A. et al. Highly accurate protein structure prediction with AlphaFold. Nature 596, 583–589 (2021). https://doi.org/10.1038/s41586-021-03819-2
+Y, si corresponde:
+++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no es especificado de otra manera. Las banderas son designadas como cierto o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede obtener desde Terminal con la bandera-h--help.
Encuentra los genes más correlacionados a un gen de interés, o bién, encuentra los tejidos donde un gen se expresa usando la base de datos ARCHS4.
+Produce: Resultados en formato JSON (Terminal) o Dataframe/CSV (Python).
Parámetro posicional
+gene
+Nombre corto (símbolo del gen) del gen de interés, p. ej. STAT4.
+Alternativamente: usa la bandera --ensembl para ingresar un ID tipo Ensembl, p. ej. ENSG00000138378.
Parámetros optionales
+-w --which
+'correlation' (correlación; se usa por defecto) o 'tissue' (tejido).
+'correlation' produce una tabla que contiene los 100 genes más correlacionados con el gen de interés. La correlación de Pearson se calcula de todas las muestras y tejidos en ARCHS4.
+'tissue' produce un atlas de expresión tisular calculado de todas las muestras humanas o de ratón (según lo definido usando el parámetro --species (especies)) en ARCHS4.
-s --species
+'human' (humano; se usa por defecto) o 'mouse' (ratón).
+Define si se usan muestras humanas o de ratón de ARCHS4.
+(Solo aplica para el atlas de expresión tisular.)
-o --out
+Ruta al archivo en el que se guardarán los resultados, p. ej. ruta/al/directorio/resultados.csv (o .json). Por defecto: salida estándar (STDOUT).
+Para Python, use save=True para guardar los resultados en el directorio de trabajo actual.
Banderas
+-e --ensembl
+Usa esta bandera si gene se ingresa como ID tipo Ensembl.
-csv --csv
+Solo para Terminal. Produce los resultados en formato CSV.
+Para Python, usa json=True para obtener los resultados en formato JSON.
-q --quiet
+Solo para Terminal. Impide la información de progreso de ser exhibida durante la ejecución del programa.
+Para Python, usa verbose=False para impedir la información de progreso de ser exhibida durante la ejecución del programa.
gget archs4 ACE2
+# Python
+gget.archs4("ACE2")
+→ Produce los 100 genes más correlacionados con el gen ACE2:
+| gene_symbol | pearson_correlation |
|---|---|
| SLC5A1 | 0.579634 |
| CYP2C18 | 0.576577 |
| . . . | . . . |
gget archs4 -w tissue ACE2
+# Python
+gget.archs4("ACE2", which="tissue")
+→ Produce la expresión tisular de ACE2 (por defecto, se utilizan datos humanos):
+| id | min | q1 | median | q3 | max |
|---|---|---|---|---|---|
| System.Urogenital/Reproductive System.Kidney.RENAL CORTEX | 0.113644 | 8.274060 | 9.695840 | 10.51670 | 11.21970 |
| System.Digestive System.Intestine.INTESTINAL EPITHELIAL CELL | 0.113644 | 5.905560 | 9.570450 | 13.26470 | 13.83590 |
| . . . | . . . | . . . | . . . | . . . | . . . |
+Consulte este tutorial de Dave Tang, quien escribió un script R para crear esta visualización con los resultados de gget archs4 en formato JSON:
Si utiliza gget archs4 en una publicación, favor de citar los siguientes artículos:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Lachmann A, Torre D, Keenan AB, Jagodnik KM, Lee HJ, Wang L, Silverstein MC, Ma’ayan A. Massive mining of publicly available RNA-seq data from human and mouse. Nature Communications 9. Article number: 1366 (2018), doi:10.1038/s41467-018-03751-6
+Bray NL, Pimentel H, Melsted P and Pachter L, Near optimal probabilistic RNA-seq quantification, Nature Biotechnology 34, p 525--527 (2016). https://doi.org/10.1038/nbt.3519
+++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Las banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
BLAST una secuencia de nucleótidos o aminoácidos a cualquier base de datos BLAST.
+Produce: Resultados en formato JSON (Terminal) o Dataframe/CSV (Python).
Parámetro posicional
+sequence
+Secuencia de nucleótidos o aminoácidos, o una ruta a un archivo tipo FASTA o .txt.
Parámetros optionales
+-p --program
+'blastn', 'blastp', 'blastx', 'tblastn', o 'tblastx'.
+Por defecto: 'blastn' para secuencias de nucleótidos; 'blastp' para secuencias de aminoácidos.
-db --database
+'nt', 'nr', 'refseq_rna', 'refseq_protein', 'swissprot', 'pdbaa', o 'pdbnt'.
+Por defecto: 'nt' para secuencias de nucleótidos; 'nr' para secuencias de aminoácidos.
+Más información sobre los bases de datos BLAST
-l --limit
+Limita el número de resultados producidos. Por defecto: 50.
-e --expect
+Define el umbral de 'expect value'. Por defecto: 10.0.
-o --out
+Ruta al archivo en el que se guardarán los resultados, p. ej. ruta/al/directorio/resultados.csv (o .json). Por defecto: salida estándar (STDOUT).
+Para Python, usa save=True para guardar los resultados en el directorio de trabajo actual.
Banderas
+-lcf --low_comp_filt
+Activa el 'low complexity filter' (filtro de baja complejidad).
-mbo --megablast_off
+Desactiva el algoritmo MegaBLAST. Por defecto: MegaBLAST esta activado (solo aplicable para blastn).
-csv --csv
+Solo para Terminal. Produce los resultados en formato CSV.
+Para Python, usa json=True para producir los resultados en formato JSON.
-q --quiet
+Solo para Terminal. Impide la información de progreso de ser exhibida durante la ejecución del programa.
+Para Python, usa verbose=False para imipidir la informacion de progreso de ser exhibida durante la ejecución del programa.
wrap_text
+Solo para Python. wrap_text=True muestra los resultados con texto envuelto para facilitar la lectura (por defecto: False).
gget blast MKWMFKEDHSLEHRCVESAKIRAKYPDRVPVIVEKVSGSQIVDIDKRKYLVPSDITVAQFMWIIRKRIQLPSEKAIFLFVDKTVPQSR
+# Python
+gget.blast("MKWMFKEDHSLEHRCVESAKIRAKYPDRVPVIVEKVSGSQIVDIDKRKYLVPSDITVAQFMWIIRKRIQLPSEKAIFLFVDKTVPQSR")
+→ Produce los resultados BLAST de la secuencia de interés. gget blast automáticamente detecta esta secuencia como una secuencia de aminoácidos y, por lo tanto, establece el programa BLAST en blastp con la base de datos nr.
| Description | Scientific Name | Common Name | Taxid | Max Score | Total Score | Query Cover | ... |
|---|---|---|---|---|---|---|---|
| PREDICTED: gamma-aminobutyric acid receptor-as... | Colobus angolensis palliatus | NaN | 336983 | 180 | 180 | 100% | ... |
| . . . | . . . | . . . | . . . | . . . | . . . | . . . | ... |
BLAST desde un archivo .fa o .txt:
+gget blast fasta.fa
+# Python
+gget.blast("fasta.fa")
+→ Produce los resultados BLAST de la primera secuencia contenida en el archivo 'fasta.fa'.
+Si utiliza gget blast en una publicación, favor de citar los siguientes artículos:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ. Basic local alignment search tool. J Mol Biol. 1990 Oct 5;215(3):403-10. doi: 10.1016/S0022-2836(05)80360-2. PMID: 2231712.
+++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Encuentra la ubicación genómica de una secuencia de nucleótidos o aminoácidos usando BLAT.
+Produce: Resultados en formato JSON (Terminal) o Dataframe/CSV (Python).
Parámetro posicional
+sequence
+Secuencia de nucleótidos o aminoácidos, o una ruta a un archivo tipo FASTA o .txt.
Parámetros optionales
+-st --seqtype
+'DNA', 'protein', 'translated%20RNA', o 'translated%20DNA'.
+Por defecto: 'DNA' para secuencias de nucleótidos; 'protein' para secuencias de aminoácidos.
-a --assembly
+Ensamblaje del genoma. 'human' (hg38) (se usa por defecto), 'mouse' (mm39) (ratón), 'zebrafish' (taeGut2) (pinzón cebra),
+o cualquiera de los ensamblajes de especies disponibles aquí (use el nombre corto del ensamblado, p. ej. 'hg38').
-o --out
+Ruta al archivo en el que se guardarán los resultados, p. ej. ruta/al/directorio/resultados.csv (o .json). Por defecto: salida estándar (STDOUT).
+Para Python, usa save=True para guardar los resultados en el directorio de trabajo actual.
Banderas
+-csv --csv
+Solo para Terminal. Produce los resultados en formato CSV.
+Para Python, usa json=True para producir los resultados en formato JSON.
-q --quiet
+Solo para Terminal. Impide la información de progreso de ser exhibida durante la ejecución del programa.
+Para Python, usa verbose=False para impedir la información de progreso de ser exhibida durante la ejecución del programa.
gget blat -a taeGut2 MKWMFKEDHSLEHRCVESAKIRAKYPDRVPVIVEKVSGSQIVDIDKRKYLVPSDITVAQFMWIIRKRIQLPSEKAIFLFVDKTVPQSR
+# Python
+gget.blat("MKWMFKEDHSLEHRCVESAKIRAKYPDRVPVIVEKVSGSQIVDIDKRKYLVPSDITVAQFMWIIRKRIQLPSEKAIFLFVDKTVPQSR", assembly="taeGut2")
+→ Produce los resultados de BLAT para el ensamblaje taeGut2 (pinzón cebra). En este ejemplo, gget blat automáticamente detecta esta secuencia como una secuencia de aminoácidos y, por lo tanto, establece el tipo de secuencia (--seqtype) como proteína.
| genome | query_size | aligned_start | aligned_end | matches | mismatches | %_aligned | ... |
|---|---|---|---|---|---|---|---|
| taeGut2 | 88 | 12 | 88 | 77 | 0 | 87.5 | ... |
Si utiliza gget blat en una publicación, favor de citar los siguientes artículos:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Kent WJ. BLAT--the BLAST-like alignment tool. Genome Res. 2002 Apr;12(4):656-64. doi: 10.1101/gr.229202. PMID: 11932250; PMCID: PMC187518.
+++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Las banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Query data de la base de datos CZ CELLxGENE Discover usando CZ CELLxGENE Discover Census.
+Produce: Un objeto AnnData que contiene la matriz de recuentos de genes y los metadatos de resultados de single cell RNA-seq de los tejidos/genes/etcetera previamente definidos.
Antes de usar gget cellxgene por primera vez, corre gget setup cellxgene / gget.setup("cellxgene") (ver también gget setup).
Parámetros opcionales
+-s --species
+'homo_sapiens' o 'mus_musculus'. Por defecto: 'homo_sapiens'.
-g --gene
+Str o lista de genes de interés o ID(s) tipo Ensembl. Por defecto: None (ninguno).
+Atención: Utilice la bandera -e / --ensembl (Python: ensembl=True) cuando ingrese ID(s) tipo Ensembl.
+Ver https://cellxgene.cziscience.com/gene-expression para ejemplos de genes.
-cv --census_version
+Versión del CZ CELLxGENE Discover Census (str), p. ej. "2023-05-15", o "latest" (ultima) o "stable" (estable). Por defecto: "stable" (estable).
-cn --column_names
+Lista de columnas de metadatos a obtener (almacenadas en AnnData.obs).
+Por defecto: ['dataset_id', 'assay', 'suspension_type', 'sex', 'tissue_general', 'tissue', 'cell_type']
+Para más opciones, ver: https://api.cellxgene.cziscience.com/curation/ui/#/ -> 'Schemas' -> 'dataset'
-o --out
+Ruta al archivo para guardar el objeto AnnData formato .h5ad (o .csv con bandera -mo / --meta_only).
+¡Requerido cuando se usa desde Terminal!
Banderas
+-e --ensembl
+Usa esta bandera si gene se ingresa como ID tipo Ensembl.
-mo --meta_only
+Solo produce la tabla (Dataframe) con metadatos (corresponde a AnnData.obs).
-q --quiet
+Solo para Terminal. Impide la información de progreso de ser exhibida durante la ejecución del programa.
+Para Python, usa verbose=False para impedir la información de progreso de ser exhibida durante la ejecución del programa.
Parámetros opcionales correspondientes a los atributos de metadatos de CZ CELLxGENE Discover
+--tissue
+Str o lista de tejido(s), p. ej. ['lung', 'blood']. Por defecto: None.
+Ver https://cellxgene.cziscience.com/gene-expression para ejemplos de tejidos.
--cell_type
+Str o lista de tipo(s) de célula(s), p. ej. ['mucus secreting cell', 'neuroendocrine cell']. Por defecto: None.
+Ver https://cellxgene.cziscience.com/gene-expression y seleccione un tejido para ejemplos de tipos de células.
--development_stage
+Str o lista de etapa(s) de desarrollo. Por defecto: None.
--disease
+Str o lista de enfermedad(es). Por defecto: None.
--sex
+Str o lista de sexo(s), p. ej. 'female' (femenina). Por defecto: None.
--dataset_id
+Str o lista de CELLxGENE ID(s). Por defecto: None.
--tissue_general_ontology_term_id
+Str o lista de tejido(s) del tipo high-level UBERON ID. Por defecto: None.
+Tejidos y sus IDs tipo UBERON se enumeran aquí.
--tissue_general
+Str o lista de tejido(s) del tipo high-level. Por defecto: None.
+Tejidos y sus IDs de UBERON se enumeran aquí.
--tissue_ontology_term_id
+Str o lista de ID(s) de 'tissue ontology term' como están definidos en el esquema de datos del CELLxGENE. Por defecto: None.
--assay_ontology_term_id
+Str o lista de ID(s) de 'assay ontology term' como están definidos en el esquema de datos del CELLxGENE. Por defecto: None.
--assay
+Str o lista de 'assays' (métodos) como están definidos en el esquema de datos del CELLxGENE. Por defecto: None.
--cell_type_ontology_term_id
+Str o lista de ID(s) de 'celltype ontology term' como están definidos en el esquema de datos del CELLxGENE. Por defecto: None.
--development_stage_ontology_term_id
+Str o lista de ID(s) de 'development stage ontology term' como están definidos en el esquema de datos del CELLxGENE. Por defecto: None.
--disease_ontology_term_id
+Str o lista de ID(s) de 'disease ontology term' como están definidos en el esquema de datos del CELLxGENE. Por defecto: None.
--donor_id
+Str o lista de ID(s) de 'donor' (donador) como están definidos en el esquema de datos del CELLxGENE. Por defecto: None.
--self_reported_ethnicity_ontology_term_id
+Str o lista de ID(s) de 'self-reported ethnicity ontology' como están definidos en el esquema de datos del CELLxGENE. Por defecto: None.
--self_reported_ethnicity
+Str o lista de etnias autoinformadas como están definidas en el esquema de datos del CELLxGENE. Por defecto: None.
--sex_ontology_term_id
+Str o lista de ID(s) de 'sex ontology' como están definidos en el esquema de datos del CELLxGENE. Por defecto: None.
--suspension_type
+Str o lista de tipo(s) de suspensión como están definidos en el esquema de datos del CELLxGENE. Por defecto: None.
gget cellxgene --gene ACE2 ABCA1 SLC5A1 --tissue lung --cell_type 'mucus secreting cell' 'neuroendocrine cell' -o example_adata.h5ad
+# Python
+adata = gget.cellxgene(
+ gene = ["ACE2", "ABCA1", "SLC5A1"],
+ tissue = "lung",
+ cell_type = ["mucus secreting cell", "neuroendocrine cell"]
+)
+adata
+→ Produce un objeto AnnData que contiene la matriz de recuentos de scRNAseq de los genes ACE2, ABCA1 y SLC5A1 en 3322 células secretoras de mucosidad y neuroendocrinas pulmonares humanas y sus metadatos correspondientes.
+Obtiene solo los metadatos (corresponde a AnnData.obs):
+gget cellxgene --meta_only --gene ENSMUSG00000015405 --ensembl --tissue lung --species mus_musculus -o example_meta.csv
+# Python
+df = gget.cellxgene(
+ meta_only = True,
+ gene = "ENSMUSG00000015405",
+ ensembl = True,
+ tissue = "lung",
+ species = "mus_musculus"
+)
+df
+→ Produce solo los metadatos de los conjuntos de datos de ENSMUSG00000015405 (ACE2), los cuales corresponden a células pulmonares murinas.
+Ver también: https://chanzuckerberg.github.io/cellxgene-census/notebooks/api_demo/census_gget_demo.html
+Si utiliza gget cellxgene en una publicación, favor de citar los siguientes artículos:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Chanzuckerberg Initiative. (n.d.). CZ CELLxGENE Discover. Retrieved [insert date here], from https://cellxgene.cziscience.com/
+Si utiliza gget en una publicación, favor de citar:
+Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
Si utiliza gget alphafold, favor de citar también:
Y si aplica:
+Si utiliza gget archs4, favor de citar también:
Lachmann A, Torre D, Keenan AB, Jagodnik KM, Lee HJ, Wang L, Silverstein MC, Ma’ayan A. Massive mining of publicly available RNA-seq data from human and mouse. Nature Communications 9. Article number: 1366 (2018), doi:10.1038/s41467-018-03751-6
+Bray NL, Pimentel H, Melsted P and Pachter L, Near optimal probabilistic RNA-seq quantification, Nature Biotechnology 34, p 525--527 (2016). https://doi.org/10.1038/nbt.3519
+Si utiliza gget bgee, favor de citar también:
Si utiliza gget blast, favor de citar también:
Si utiliza gget blat, favor de citar también:
Si utiliza gget cbio, favor de citar también:
Cerami E, Gao J, Dogrusoz U, Gross BE, Sumer SO, Aksoy BA, Jacobsen A, Byrne CJ, Heuer ML, Larsson E, Antipin Y, Reva B, Goldberg AP, Sander C, Schultz N. The cBio cancer genomics portal: an open platform for exploring multidimensional cancer genomics data. Cancer Discov. 2012 May;2(5):401-4. doi: 10.1158/2159-8290.CD-12-0095. Erratum in: Cancer Discov. 2012 Oct;2(10):960. PMID: 22588877; PMCID: PMC3956037.
+Gao J, Aksoy BA, Dogrusoz U, Dresdner G, Gross B, Sumer SO, Sun Y, Jacobsen A, Sinha R, Larsson E, Cerami E, Sander C, Schultz N. Integrative analysis of complex cancer genomics and clinical profiles using the cBioPortal. Sci Signal. 2013 Apr 2;6(269):pl1. doi: 10.1126/scisignal.2004088. PMID: 23550210; PMCID: PMC4160307.
+de Bruijn I, Kundra R, Mastrogiacomo B, Tran TN, Sikina L, Mazor T, Li X, Ochoa A, Zhao G, Lai B, Abeshouse A, Baiceanu D, Ciftci E, Dogrusoz U, Dufilie A, Erkoc Z, Garcia Lara E, Fu Z, Gross B, Haynes C, Heath A, Higgins D, Jagannathan P, Kalletla K, Kumari P, Lindsay J, Lisman A, Leenknegt B, Lukasse P, Madela D, Madupuri R, van Nierop P, Plantalech O, Quach J, Resnick AC, Rodenburg SYA, Satravada BA, Schaeffer F, Sheridan R, Singh J, Sirohi R, Sumer SO, van Hagen S, Wang A, Wilson M, Zhang H, Zhu K, Rusk N, Brown S, Lavery JA, Panageas KS, Rudolph JE, LeNoue-Newton ML, Warner JL, Guo X, Hunter-Zinck H, Yu TV, Pilai S, Nichols C, Gardos SM, Philip J; AACR Project GENIE BPC Core Team, AACR Project GENIE Consortium; Kehl KL, Riely GJ, Schrag D, Lee J, Fiandalo MV, Sweeney SM, Pugh TJ, Sander C, Cerami E, Gao J, Schultz N. Analysis and Visualization of Longitudinal Genomic and Clinical Data from the AACR Project GENIE Biopharma Collaborative in cBioPortal. Cancer Res. 2023 Dec 1;83(23):3861-3867. doi: 10.1158/0008-5472.CAN-23-0816. PMID: 37668528; PMCID: PMC10690089.
+Please also cite the source of the data if you are using a publicly available dataset.
+Si utiliza gget cellxgene, favor de citar también:
Si utiliza gget cosmic, favor de citar también:
Si utiliza gget diamond, favor de citar también:
Si utiliza gget elm, favor de citar también:
Laura Luebbert, Chi Hoang, Manjeet Kumar, Lior Pachter, Fast and scalable querying of eukaryotic linear motifs with gget elm, Bioinformatics, 2024, btae095, https://doi.org/10.1093/bioinformatics/btae095
+Manjeet Kumar, Sushama Michael, Jesús Alvarado-Valverde, Bálint Mészáros, Hugo Sámano‐Sánchez, András Zeke, Laszlo Dobson, Tamas Lazar, Mihkel Örd, Anurag Nagpal, Nazanin Farahi, Melanie Käser, Ramya Kraleti, Norman E Davey, Rita Pancsa, Lucía B Chemes, Toby J Gibson, The Eukaryotic Linear Motif resource: 2022 release, Nucleic Acids Research, Volume 50, Issue D1, 7 January 2022, Pages D497–D508, https://doi.org/10.1093/nar/gkab975
+Si utiliza gget enrichr, favor de citar también:
Chen EY, Tan CM, Kou Y, Duan Q, Wang Z, Meirelles GV, Clark NR, Ma'ayan A. Enrichr: interactive and collaborative HTML5 gene list enrichment analysis tool. BMC Bioinformatics. 2013; 128(14). https://doi.org/10.1186/1471-2105-14-128
+Kuleshov MV, Jones MR, Rouillard AD, Fernandez NF, Duan Q, Wang Z, Koplev S, Jenkins SL, Jagodnik KM, Lachmann A, McDermott MG, Monteiro CD, Gundersen GW, Ma'ayan A. Enrichr: a comprehensive gene set enrichment analysis web server 2016 update. Nucleic Acids Research. 2016; gkw377. doi: 10.1093/nar/gkw377
+Xie Z, Bailey A, Kuleshov MV, Clarke DJB., Evangelista JE, Jenkins SL, Lachmann A, Wojciechowicz ML, Kropiwnicki E, Jagodnik KM, Jeon M, & Ma’ayan A. Gene set knowledge discovery with Enrichr. Current Protocols, 1, e90. 2021. doi: 10.1002/cpz1.90.
+Si trabaja con conjuntos de datos no humanos/ratón, cite también:
+Si utiliza gget info, favor de citar también:
Martin FJ, Amode MR, Aneja A, Austine-Orimoloye O, Azov AG, Barnes I, Becker A, Bennett R, Berry A, Bhai J, Bhurji SK, Bignell A, Boddu S, Branco Lins PR, Brooks L, Ramaraju SB, Charkhchi M, Cockburn A, Da Rin Fiorretto L, Davidson C, Dodiya K, Donaldson S, El Houdaigui B, El Naboulsi T, Fatima R, Giron CG, Genez T, Ghattaoraya GS, Martinez JG, Guijarro C, Hardy M, Hollis Z, Hourlier T, Hunt T, Kay M, Kaykala V, Le T, Lemos D, Marques-Coelho D, Marugán JC, Merino GA, Mirabueno LP, Mushtaq A, Hossain SN, Ogeh DN, Sakthivel MP, Parker A, Perry M, Piližota I, Prosovetskaia I, Pérez-Silva JG, Salam AIA, Saraiva-Agostinho N, Schuilenburg H, Sheppard D, Sinha S, Sipos B, Stark W, Steed E, Sukumaran R, Sumathipala D, Suner MM, Surapaneni L, Sutinen K, Szpak M, Tricomi FF, Urbina-Gómez D, Veidenberg A, Walsh TA, Walts B, Wass E, Willhoft N, Allen J, Alvarez-Jarreta J, Chakiachvili M, Flint B, Giorgetti S, Haggerty L, Ilsley GR, Loveland JE, Moore B, Mudge JM, Tate J, Thybert D, Trevanion SJ, Winterbottom A, Frankish A, Hunt SE, Ruffier M, Cunningham F, Dyer S, Finn RD, Howe KL, Harrison PW, Yates AD, Flicek P. Ensembl 2023. Nucleic Acids Res. 2023 Jan 6;51(D1):D933-D941. doi: 10.1093/nar/gkac958. PMID: 36318249; PMCID: PMC9825606.
+Sayers EW, Beck J, Bolton EE, Brister JR, Chan J, Comeau DC, Connor R, DiCuccio M, Farrell CM, Feldgarden M, Fine AM, Funk K, Hatcher E, Hoeppner M, Kane M, Kannan S, Katz KS, Kelly C, Klimke W, Kim S, Kimchi A, Landrum M, Lathrop S, Lu Z, Malheiro A, Marchler-Bauer A, Murphy TD, Phan L, Prasad AB, Pujar S, Sawyer A, Schmieder E, Schneider VA, Schoch CL, Sharma S, Thibaud-Nissen F, Trawick BW, Venkatapathi T, Wang J, Pruitt KD, Sherry ST. Database resources of the National Center for Biotechnology Information. Nucleic Acids Res. 2024 Jan 5;52(D1):D33-D43. doi: 10.1093/nar/gkad1044. PMID: 37994677; PMCID: PMC10767890.
+The UniProt Consortium , UniProt: the Universal Protein Knowledgebase in 2023, Nucleic Acids Research, Volume 51, Issue D1, 6 January 2023, Pages D523–D531, https://doi.org/10.1093/nar/gkac1052
+Si utiliza gget muscle, favor de citar también:
Si utiliza gget opentargets, favor de citar también:
Si utiliza gget pdb, favor de citar también:
Si utiliza gget ref o gget search, favor de citar también:
Si utiliza gget seq, favor de citar también:
Martin FJ, Amode MR, Aneja A, Austine-Orimoloye O, Azov AG, Barnes I, Becker A, Bennett R, Berry A, Bhai J, Bhurji SK, Bignell A, Boddu S, Branco Lins PR, Brooks L, Ramaraju SB, Charkhchi M, Cockburn A, Da Rin Fiorretto L, Davidson C, Dodiya K, Donaldson S, El Houdaigui B, El Naboulsi T, Fatima R, Giron CG, Genez T, Ghattaoraya GS, Martinez JG, Guijarro C, Hardy M, Hollis Z, Hourlier T, Hunt T, Kay M, Kaykala V, Le T, Lemos D, Marques-Coelho D, Marugán JC, Merino GA, Mirabueno LP, Mushtaq A, Hossain SN, Ogeh DN, Sakthivel MP, Parker A, Perry M, Piližota I, Prosovetskaia I, Pérez-Silva JG, Salam AIA, Saraiva-Agostinho N, Schuilenburg H, Sheppard D, Sinha S, Sipos B, Stark W, Steed E, Sukumaran R, Sumathipala D, Suner MM, Surapaneni L, Sutinen K, Szpak M, Tricomi FF, Urbina-Gómez D, Veidenberg A, Walsh TA, Walts B, Wass E, Willhoft N, Allen J, Alvarez-Jarreta J, Chakiachvili M, Flint B, Giorgetti S, Haggerty L, Ilsley GR, Loveland JE, Moore B, Mudge JM, Tate J, Thybert D, Trevanion SJ, Winterbottom A, Frankish A, Hunt SE, Ruffier M, Cunningham F, Dyer S, Finn RD, Howe KL, Harrison PW, Yates AD, Flicek P. Ensembl 2023. Nucleic Acids Res. 2023 Jan 6;51(D1):D933-D941. doi: 10.1093/nar/gkac958. PMID: 36318249; PMCID: PMC9825606.
+The UniProt Consortium , UniProt: the Universal Protein Knowledgebase in 2023, Nucleic Acids Research, Volume 51, Issue D1, 6 January 2023, Pages D523–D531, https://doi.org/10.1093/nar/gkac1052
+gget es tan preciso como la base de datos/servidores/APIs que utiliza. La exactitud o fiabilidad de los datos no es garantizada por ningún motivo. Los proveedores por ningún motivo seran responsables de (incluyendo, sin limite alguno) la calidad, ejecución, o comerciabilidad para cualquier propósito particular surgiendo del uso o la incapacidad de usar los datos.
Nosotros, como miembros, contribuyentes y administradores nos comprometemos a hacer de la participación en nuestra comunidad sea una experiencia libre de acoso para todo el mundo, independientemente de la edad, dimensión corporal, discapacidad visible o invisible, etnicidad, características sexuales, identidad y expresión de género, nivel de experiencia, educación, nivel socio-económico, nacionalidad, apariencia personal, raza, casta, color, religión, o identidad u orientación sexual.
+Nos comprometemos a actuar e interactuar de maneras que contribuyan a una comunidad abierta, acogedora, diversa, inclusiva y sana.
+Ejemplos de comportamientos que contribuyen a crear un ambiente positivo para nuestra comunidad:
+Ejemplos de comportamiento inaceptable:
+Los administradores de la comunidad son responsables de aclarar y hacer cumplir nuestros estándares de comportamiento aceptable y tomarán acciones apropiadas y correctivas de forma justa en respuesta a cualquier comportamiento que consideren inapropiado, amenazante, ofensivo o dañino.
+Los administradores de la comunidad tendrán el derecho y la responsabilidad de eliminar, editar o rechazar comentarios, commits, código, ediciones de páginas de wiki, issues y otras contribuciones que no se alineen con este Código de Conducta, y comunicarán las razones para sus decisiones de moderación cuando sea apropiado.
+Este código de conducta aplica tanto a espacios del proyecto como a espacios públicos donde un individuo esté en representación del proyecto o comunidad. Ejemplos de esto incluyen el uso de la cuenta oficial de correo electrónico, publicaciones a través de las redes sociales oficiales, o presentaciones con personas designadas en eventos en línea o no.
+Instancias de comportamiento abusivo, acosador o inaceptable de otro modo podrán ser reportadas a los administradores de la comunidad responsables del cumplimiento a través de lpachter@caltech.edu. Todas las quejas serán evaluadas e investigadas de una manera puntual y justa.
+Todos los administradores de la comunidad están obligados a respetar la privacidad y la seguridad de quienes reporten incidentes.
+Los administradores de la comunidad seguirán estas Guías de Impacto en la Comunidad para determinar las consecuencias de cualquier acción que juzguen como un incumplimiento de este Código de Conducta:
+Impacto en la Comunidad: El uso de lenguaje inapropiado u otro comportamiento considerado no profesional o no acogedor en la comunidad.
+Consecuencia: Un aviso escrito y privado por parte de los administradores de la comunidad, proporcionando claridad alrededor de la naturaleza de este incumplimiento y una explicación de por qué el comportamiento es inaceptable. Una disculpa pública podría ser solicitada.
+Impacto en la Comunidad: Un incumplimiento causado por un único incidente o por una cadena de acciones.
+Consecuencia: Un aviso con consecuencias por comportamiento prolongado. No se interactúa con las personas involucradas, incluyendo interacción no solicitada con quienes se encuentran aplicando el Código de Conducta, por un periodo especificado de tiempo. Esto incluye evitar las interacciones en espacios de la comunidad, así como a través de canales externos como las redes sociales. Incumplir estos términos puede conducir a una expulsión temporal o permanente.
+Impacto en la Comunidad: Una serie de incumplimientos de los estándares de la comunidad, incluyendo comportamiento inapropiado continuo.
+Consecuencia: Una expulsión temporal de cualquier forma de interacción o comunicación pública con la comunidad durante un intervalo de tiempo especificado. No se permite interactuar de manera pública o privada con las personas involucradas, incluyendo interacciones no solicitadas con quienes se encuentran aplicando el Código de Conducta, durante este periodo. Incumplir estos términos puede conducir a una expulsión permanente.
+Impacto en la Comunidad: Demostrar un patrón sistemático de incumplimientos de los estándares de la comunidad, incluyendo conductas inapropiadas prolongadas en el tiempo, acoso de individuos, o agresiones o menosprecio a grupos de individuos.
+Consecuencia: Una expulsión permanente de cualquier tipo de interacción pública con la comunidad del proyecto.
+Este Código de Conducta es una adaptación del Contributor Covenant, versión 2.1, +disponible en https://www.contributor-covenant.org/es/version/2/1/code_of_conduct.html
+Las Guías de Impacto en la Comunidad están inspiradas en la escalera de aplicación del código de conducta de Mozilla.
+Para respuestas a las preguntas frecuentes de este código de conducta, consulta las FAQ en +https://www.contributor-covenant.org/faq. Hay traducciones disponibles en https://www.contributor-covenant.org/translationshttps://www.contributor-covenant.org/translations
+ +¡Gracias por invertir su tiempo en contribuir con nuestro proyecto! Cualquier contribución que hagas se verá reflejada en el repositorio de GitHub de gget. ✨
+Lea nuestro Código de conducta para mantener nuestra comunidad accesible y respetable.
+En esta guía, obtendrá una descripción general del flujo de trabajo de contribución desde la creación de un GitHub Issue (asunto) o la creación de un GitHub Pull Request (PR) hasta la revisión y fusión de un PR.
+Si detecta un problema con gget o tiene una idea para una nueva función, comproba si ya existe un Issue para este problema/sugerencia. Si no existe un Issue relacionado, puede abrir un nuevo Issue utilizando el formulario correspondiente.
+Explore nuestros Issues existentes para encontrar uno que le interese. Puede restringir la búsqueda utilizando "labels" como filtros. Si encuentra un Issue en el que desea trabajar, puede abrir un PR con una solución.
+Usando GitHub Desktop:
+Usando la Terminal:
+Confirme sus cambios una vez que esté satisfecho con ellos.
+pip install -r dev-requirements.txtcoverage run -m pytest -ra -v tests && coverage report --omit=main.py,tests*Si tiene alguna pregunta, no dude en iniciar una discusión o crear un Issue como se describe anteriormente.
+Cuando haya terminado con los cambios, cree un Pull Request, también conocido como "PR".
+‼️ Realice todos los PRs contra la rama dev del repositorio gget
Una vez que envíe su PR, un miembro del equipo gget revisará su propuesta. Podemos hacer preguntas o solicitar información adicional.
+¡Felicidades! 🎉 El equipo de gget te lo agradece. ✨
+Una vez que su PR se fusione, sus contribuciones serán visibles públicamente en el repositorio de gget.
+ +++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Las banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Busque genes, mutaciones, etc. asociados con cánceres utilizando la base de datos COSMIC (Catálogo de mutaciones somáticas en cáncer).
+Produce: JSON (línea de comandos) o marco de datos/CSV (Python) cuando download_cosmic=False. Cuando download_cosmic=True, descarga la base de datos solicitada en la carpeta especificada.
Este módulo fue escrito en parte por @AubakirovArman (consulta de información) y @josephrich98 (descarga de base de datos).
+NOTA: Se aplican tarifas de licencia para el uso comercial de COSMIC. Puede leer más sobre la concesión de licencias de datos COSMIC aquí.
+Parámetro posicional (para consultar información)
+searchterm
+Término de búsqueda, que puede ser una mutación, un nombre de gen (o ID de Ensembl), una muestra, etc.
+Ejemplos para los argumentos de searchterm y entidad:
| searchterm | entidad | |
|---|---|---|
| EGFR | mutaciones | -> Encuentra mutaciones en el gen EGFR asociadas con el cáncer |
| v600e | mutaciones | -> Encuentra genes para los cuales una mutación v600e está asociada con el cáncer |
| COSV57014428 | mutaciones | -> Encuentra mutaciones asociadas con esta ID de mutaciones COSMIC |
| EGFR | genes | -> Obtiene el número de muestras, mutaciones simples/codificantes y fusiones observadas en COSMIC para EGFR |
| prostate | cáncer | -> Obtiene el número de muestras probadas y mutaciones para el cáncer de próstata |
| prostate | sitio_tumoral | -> Obtiene el número de muestras probadas, genes, mutaciones, fusiones, etc. con 'próstata' como sitio de tejido primario |
| ICGC | estudios | -> Obtiene el código de proyecto y descripciones de todos los estudios de ICGC (Consortio Internacional del Genoma del Cáncer) |
| EGFR | pubmed | -> Encuentra publicaciones de PubMed sobre EGFR y cáncer |
| ICGC | muestras | -> Obtiene metadatos sobre todas las muestras de ICGC (Consortio Internacional del Genoma del Cáncer) |
| COSS2907494 | muestras | -> Obtiene metadatos sobre esta ID de muestra COSMIC (tipo de cáncer, tejido, # genes analizados, # mutaciones, etc.) |
NOTA: (Solo Python) Establezca en None cuando se descarguen bases de datos COSMIC con download_cosmic=True.
Parámetros opcionales (para consultar información)
+-e --entity
+'mutations' (predeterminado), 'genes', 'cáncer', 'sitio_tumoral', 'estudios', 'pubmed' o 'muestras'.
+Define el tipo de resultados a devolver.
-l --limit
+Limita el número de resultados a devolver. Predeterminado: 100.
Banderas (para descargar bases de datos COSMIC)
+-d --download_cosmic
+Conmuta al modo de descarga de base de datos.
-gm --gget_mutate
+DESACTIVA la creación de una versión modificada de la base de datos para usar con gget mutate.
+Python: gget_mutate es Verdadero por defecto. Establezca gget_mutate=False para deshabilitar.
Parámetros opcionales (para descargar bases de datos COSMIC)
+-mc --mutation_class
+'cancer' (predeterminado), 'cell_line', 'census', 'resistance', 'genome_screen', 'targeted_screen', o 'cancer_example'
+Tipo de base de datos COSMIC para descargar.
-cv --cosmic_version
+Versión de la base de datos COSMIC. Predeterminado: Ninguno -> Se establece en la última versión por defecto.
-gv --grch_version
+Versión del genoma de referencia humano GRCh en el que se basó la base de datos COSMIC (37 o 38). Predeterminado: 37
Parámetros opcionales (generales)
+-o --out
+Ruta al archivo (o carpeta cuando se descargan bases de datos con el flag download_cosmic) donde se guardarán los resultados, p. ej. 'ruta/a/resultados.json'.
+Predeterminado: None
+-> Cuando download_cosmic=False: Los resultados se devolverán a la salida estándar
+-> Cuando download_cosmic=True: La base de datos se descargará en el directorio de trabajo actual
Banderas (generales)
+-csv --csv
+Solo para Terminal. Produce los resultados en formato CSV.
+Para Python, usa json=True para producir los resultados en formato JSON.
-q --quiet
+Solo para Terminal. Impide la información de progreso de ser exhibida durante la ejecución del programa.
+Para Python, usa verbose=False para imipidir la informacion de progreso de ser exhibida durante la ejecución del programa.
gget cosmic -e genes EGFR
+# Python
+gget.cosmic("EGFR", entity="genes")
+→ Devuelve mutaciones en el gen EGFR asociadas con el cáncer en el formato:
+| Gene | Syntax | Alternate IDs | Canonical |
|---|---|---|---|
| EGFR | c.*2446A>G | EGFR c.*2446A>G, EGFR p.?, ... | y |
| EGFR | c.(2185_2283)ins(18) | EGFR c.(2185_2283)ins(18), EGFR p.?, ... | y |
| . . . | . . . | . . . | . . . |
gget cosmic --download_cosmic
+# Python
+gget.cosmic(searchterm=None, download_cosmic=True)
+→ Descargue la base de datos sobre cáncer de COSMIC de la última versión de COSMIC.
+Si utiliza gget alphafold en una publicación, favor de citar los siguientes artículos:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Tate JG, Bamford S, Jubb HC, Sondka Z, Beare DM, Bindal N, Boutselakis H, Cole CG, Creatore C, Dawson E, Fish P, Harsha B, Hathaway C, Jupe SC, Kok CY, Noble K, Ponting L, Ramshaw CC, Rye CE, Speedy HE, Stefancsik R, Thompson SL, Wang S, Ward S, Campbell PJ, Forbes SA. COSMIC: the Catalogue Of Somatic Mutations In Cancer. Nucleic Acids Res. 2019 Jan 8;47(D1):D941-D947. doi: 10.1093/nar/gky1015. PMID: 30371878; PMCID: PMC6323903.
+++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Las banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Alinee múltiples proteínas o secuencias de ADN traducidas usando DIAMOND (DIAMOND es similar a BLAST, pero este es un cálculo local).
+Produce: Resultados en formato JSON (Terminal) o Dataframe/CSV (Python).
Parámetro posicional
+query
+Secuencia(s) (str o lista) de aminoácidos, o una ruta a un archivo tipo FASTA.
Parámetro requerido
+-ref --reference
+Secuencias de aminoácidos de referencia (str o lista), o una ruta a un archivo tipo FASTA.
Parámetros optionales
+-db --diamond_db
+Ruta para guardar la base de datos DIAMOND creada a partir de reference (str).
+Por defecto: None -> El archivo de base de datos DIAMOND temporal se eliminará después de la alineación o se guardará en out si se proporciona out.
-s --sensitivity
+Sensibilidad de la alineación (str). Por defecto: "very-sensitive" (muy sensible).
+Uno de los siguientes: fast, mid-sensitive, sensitive, more-sensitive, very-sensitive, or ultra-sensitive.
-t --threads
+Número de hilos de procesamiento utilizados (int). Por defecto: 1.
-db --diamond_binary
+Ruta al binario DIAMOND (str). Por defecto: None -> Utiliza el binario DIAMOND instalado automáticamente con gget.
-o --out
+Ruta al archivo en el que se guardarán los resultados (str), p. ej. "ruta/al/directorio". Por defecto: salida estándar (STDOUT); los archivos temporales se eliminan.
Banderas
+-u --uniprot
+Use esta bandera cuando sequence es un ID de Uniprot en lugar de una secuencia de aminoácidos.
-csv --csv
+Solo para Terminal. Produce los resultados en formato CSV.
+Para Python, usa json=True para producir los resultados en formato JSON.
-q --quiet
+Solo para Terminal. Impide la información de progreso de ser exhibida durante la ejecución del programa.
+Para Python, usa verbose=False para impedir la información de progreso de ser exhibida durante la ejecución del programa.
# !!! Asegúrese de enumerar primero el argumento posicional aquí para que no se agregue como secuencia de referencia
+gget diamond GGETISAWESQME ELVISISALIVE LQVEFRANKLIN PACHTERLABRQCKS -ref GGETISAWESQMEELVISISALIVELQVEFRANKLIN PACHTERLABRQCKS
+# Python
+gget.diamond(["GGETISAWESQME", "ELVISISALIVE", "LQVEFRANKLIN", "PACHTERLABRQCKS"], reference=["GGETISAWESQMEELVISISALIVELQVEFRANKLIN", "PACHTERLABRQCKS"])
+→ Produce los resultados de la alineación en formato JSON (Terminal) o Dataframe/CSV:
+| query_accession | subject_accession | identity_percentage | query_seq_length | subject_seq_length | length | mismatches | gap_openings | query_start | query_end | subject_start | subject_end | e-value | bit_score |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Seq0 | Seq0 | 100 | 13 | 37 | 13 | 0 | 0 | 1 | 13 | 1 | 13 | 2.82e-09 | 30.8 |
| Seq2 | Seq0 | 100 | 12 | 37 | 12 | 0 | 0 | 1 | 12 | 26 | 37 | 4.35e-08 | 27.7 |
| Seq3 | Seq1 | 100 | 15 | 15 | 15 | 0 | 0 | 1 | 15 | 1 | 15 | 2.01e-11 | 36.2 |
Si utiliza gget diamond en una publicación, favor de citar los siguientes artículos:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Buchfink, B., Xie, C. & Huson, D. Fast and sensitive protein alignment using DIAMOND. Nat Methods 12, 59–60 (2015). https://doi.org/10.1038/nmeth.3176
+++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Prediga localmente motivos lineales eucarióticos (ELMs) a partir de una secuencia de aminoácidos o UniProt Acc utilizando datos de la base de datos ELM.
+Produce: Resultados en formato JSON (Terminal) o Dataframe/CSV (Python). Este módulo devuelve dos tipos de resultados (ver ejemplos).
Los datos de ELM se pueden descargar y distribuir para uso no comercial de acuerdo con el acuerdo de licencia de software de ELM.
+Si utiliza gget elm en una publicación, favor de citar:
Antes de usar gget elm por primera vez, ejecute gget setup elm / gget.setup("elm") una vez (consulte también gget setup).
Parámetro posicional
+sequence
+Secuencia de aminoácidos o Uniprot Acc (str).
+Al proporcionar una Uniprot Acc, use la bandera --uniprot (Python: uniprot=True).
Parámetros optionales
+-s sensitivity
+Sensibilidad de la alineación DIAMOND (str). Por defecto: "very-sensitive" (muy sensible).
+Uno de los siguientes: fast, mid-sensitive, sensitive, more-sensitive, very-sensitive, or ultra-sensitive.
-t threads
+Número de hilos de procesamiento utilizados en la alineación de secuencias con DIAMOND (int). Por defecto: 1.
-bin diamond_binary
+Ruta al binario DIAMOND (str). Por defecto: None -> Utiliza el binario DIAMOND instalado automáticamente con gget.
-o --out
+Ruta al archivo en el que se guardarán los resultados (str), p. ej. "ruta/al/directorio". Por defecto: salida estándar (STDOUT); los archivos temporales se eliminan.
Banderas
+-u --uniprot
+Use esta bandera cuando sequence es una Uniprot Acc en lugar de una secuencia de aminoácidos.
-e --expand
+Amplíe la información devuelta en el marco de datos de expresiones regulares para incluir los nombres de proteínas, los organismos y las referencias en las que se validó originalmente el motivo.
-csv --csv
+Solo para Terminal. Produce los resultados en formato CSV.
+Para Python, usa json=True para producir los resultados en formato JSON.
-q --quiet
+Solo para Terminal. Impide la información de progreso de ser exhibida durante la ejecución del programa.
+Para Python, usa verbose=False para impedir la información de progreso de ser exhibida durante la ejecución del programa.
Encuentre ELM en una secuencia de aminoácidos:
+gget setup elm # Descarga/actualiza la base de datos ELM local
+gget elm -o gget_elm_results LIAQSIGQASFV
+# Python
+gget.setup(“elm”) # Descarga/actualiza la base de datos ELM local
+ortholog_df, regex_df = gget.elm("LIAQSIGQASFV")
+Encuentre ELM que proporcionen a una UniProt Acc:
+gget setup elm # Descarga/actualiza la base de datos ELM local
+gget elm -o gget_elm_results --uniprot Q02410 -e
+# Python
+gget.setup(“elm”) # Descarga/actualiza la base de datos ELM local
+ortholog_df, regex_df = gget.elm("Q02410", uniprot=True, expand=True)
+→ Produce dos resultados con información extensa sobre ELMs asociados con proteínas ortólogas y motivos encontrados en la secuencia de entrada directamente en función de sus expresiones regex:
+ortholog_df:
+| Ortholog_UniProt_Acc | ProteinName | class_accession | ELMIdentifier | FunctionalSiteName | Description | Organism | … |
|---|---|---|---|---|---|---|---|
| Q02410 | APBA1_HUMAN | ELME000357 | LIG_CaMK_CASK_1 | CASK CaMK domain binding ligand motif | Motif that mediates binding to the calmodulin-dependent protein kinase (CaMK) domain of the peripheral plasma membrane protein CASK/Lin2. | Homo sapiens | … |
| Q02410 | APBA1_HUMAN | ELME000091 | LIG_PDZ_Class_2 | PDZ domain ligands | The C-terminal class 2 PDZ-binding motif is classically represented by a pattern such as | Homo sapiens | … |
regex_df:
+| Instance_accession | ELMIdentifier | FunctionalSiteName | ELMType | Description | Instances (Matched Sequence) | Organism | … |
|---|---|---|---|---|---|---|---|
| ELME000321 | CLV_C14_Caspase3-7 | Caspase cleavage motif | CLV | Caspase-3 and Caspase-7 cleavage site. | ERSDG | Mus musculus | … |
| ELME000102 | CLV_NRD_NRD_1 | NRD cleavage site | CLV | N-Arg dibasic convertase (NRD/Nardilysin) cleavage site. | RRA | Rattus norvegicus | … |
| ELME000100 | CLV_PCSK_PC1ET2_1 | PCSK cleavage site | CLV | NEC1/NEC2 cleavage site. | KRD | Mus musculus | … |
| ELME000146 | CLV_PCSK_SKI1_1 | PCSK cleavage site | CLV | Subtilisin/kexin isozyme-1 (SKI1) cleavage site. | RLLTA | Homo sapiens | … |
| ELME000231 | DEG_APCC_DBOX_1 | APCC-binding Destruction motifs | DEG | An RxxL-based motif that binds to the Cdh1 and Cdc20 components of APC/C thereby targeting the protein for destruction in a cell cycle dependent manner | SRVKLNIVR | Saccharomyces cerevisiae S288c | … |
| … | … | … | … | … | … | … | … |
(Los motivos que aparecen en muchas especies diferentes pueden parecer repetidos, pero todas las filas deben ser únicas.)
+++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Las banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Realice un análisis de enriquecimiento de una lista de genes utilizando Enrichr.
+Produce: Resultados en formato JSON (Terminal) o Dataframe/CSV (Python).
Parámetro posicional
+genes
+Lista de nombres cortos (símbolos) de los genes de interés para realizar el análisis de enriquecimiento, p. PHF14 RBM3 MSL1 PHF21A.
+Alternativamente: use la bandera --ensembl para ingresar IDs tipo Ensembl, p. ENSG00000106443 ENSG00000102317 ENSG00000188895.
Otros parámetros requeridos
+-db --database
+Base de datos que será utilizada como referencia para el análisis de enriquecimiento.
+Admite cualquier base de datos enumerada aquí o uno de los siguientes accesos directos:
+'pathway' (KEGG_2021_Human)
+'transcription' (ChEA_2016)
+'ontology' (GO_Biological_Process_2021)
+'diseases_drugs' (GWAS_Catalog_2019)
+'celltypes' (PanglaoDB_Augmented_2021)
+'kinase_interactions' (KEA_2015)
Parámetros opcionales
+-bkg_l --background_list
+Lista de nombres cortos (símbolos) de genes de 'background' (de fondo/control), p. NSUN3 POLRMT NLRX1.
+Alternativamente: usa la bandera --ensembl_background para ingresar IDs tipo Ensembl.
-o --out
+Ruta al archivo en el que se guardarán los resultados, p. ruta/al/directorio/resultados.csv (o .json). Por defecto: salida estándar (STDOUT).
+Para Python, usa save=True para guardar los resultados en el directorio de trabajo actual.
-ko --kegg_out
+Ruta al archivo png en el que se guardará la imágen de la vía de señalización celular KEGG, p. ej. ruta/al/directorio/KEGG.png. (Por defecto: None)
-kr --kegg_rank
+Rango de la ruta KEGG que se va a trazar. (Por defecto: 1)
figsize
+Solo para Python. (ancho, alto) de la visualización en pulgadas. (Por defecto: (10,10))
ax
+Solo para Python. Ingresa un objeto de ejes matplotlib para personalizar la visualización.(Por defecto: None)
Banderas
+-e --ensembl
+Usa esta bandera si genes se ingresa como una lista de IDs tipo Ensembl.
-e_b --ensembl_background
+Usa esta bandera si background_list se ingresa como una lista de IDs tipo Ensembl.
-bkg --background
+Use un conjunto de 20,625 genes 'background'
+listados aquí.
-csv --csv
+Solo para Terminal. Produce los resultados en formato CSV.
+Para Python, usa json=True produce los resultados en formato JSON.
-q --quiet
+Solo para Terminal. Impide la información de progreso de ser exhibida durante la ejecución del programa.
+Para Python, usa verbose=False para imipidir la información de progreso de ser exhibida durante la ejecución del programa.
plot
+Solo para Python. plot=True provée la visualización de los primeros 15 resultados (por defecto: False).
gget enrichr -db ontology ACE2 AGT AGTR1
+# Python
+gget.enrichr(["ACE2", "AGT", "AGTR1"], database="ontology", plot=True)
+→ Produce vías/funciones celulares relacionadas con los genes ACE2, AGT y AGTR1 de la base de datos GO Biological Process 2021. En Python, plot=True provee la visualización de resultados:

Use gget enrichr con una lista de genes 'background':
# Aquí, primero ingresamos los genes de interés (parámetro posicional 'genes'), para que no se agreguen a la lista de genes 'background' detrás del parámetro '-bkgr_l'
+gget enrichr \
+ PHF14 RBM3 MSL1 PHF21A ARL10 INSR JADE2 P2RX7 LINC00662 CCDC101 PPM1B KANSL1L CRYZL1 ANAPC16 TMCC1 CDH8 RBM11 CNPY2 HSPA1L CUL2 PLBD2 LARP7 TECPR2 ZNF302 CUX1 MOB2 CYTH2 SEC22C EIF4E3 ROBO2 ADAMTS9-AS2 CXXC1 LINC01314 ATF7 ATP5F1 \
+ -db ChEA_2022 \
+ -bkg_l NSUN3 POLRMT NLRX1 SFXN5 ZC3H12C SLC25A39 ARSG DEFB29 PCMTD2 ACAA1A LRRC1 2810432D09RIK SEPHS2 SAC3D1 TMLHE LOC623451 TSR2 PLEKHA7 GYS2 ARHGEF12 HIBCH LYRM2 ZBTB44 ENTPD5 RAB11FIP2 LIPT1 INTU ANXA13 KLF12 SAT2 GAL3ST2 VAMP8 FKBPL AQP11 TRAP1 PMPCB TM7SF3 RBM39 BRI3 KDR ZFP748 NAP1L1 DHRS1 LRRC56 WDR20A STXBP2 KLF1 UFC1 CCDC16 9230114K14RIK RWDD3 2610528K11RIK ACO1 CABLES1 LOC100047214 YARS2 LYPLA1 KALRN GYK ZFP787 ZFP655 RABEPK ZFP650 4732466D17RIK EXOSC4 WDR42A GPHN 2610528J11RIK 1110003E01RIK MDH1 1200014M14RIK AW209491 MUT 1700123L14RIK 2610036D13RIK PHF14 RBM3 MSL1 PHF21A ARL10 INSR JADE2 P2RX7 LINC00662 CCDC101 PPM1B KANSL1L CRYZL1 ANAPC16 TMCC1 CDH8 RBM11 CNPY2 HSPA1L CUL2 PLBD2 LARP7 TECPR2 ZNF302 CUX1 MOB2 CYTH2 SEC22C EIF4E3 ROBO2 ADAMTS9-AS2 CXXC1 LINC01314 ATF7 ATP5F1COX15 TMEM30A NSMCE4A TM2D2 RHBDD3 ATXN2 NFS1 3110001I20RIK BC038156 C330002I19RIK ZFYVE20 POLI TOMM70A LOC100047782 2410012H22RIK RILP A230062G08RIK PTTG1IP RAB1 AFAP1L1 LYRM5 2310026E23RIK SLC7A6OS MAT2B 4932438A13RIK LRRC8A SMO NUPL2
+# Python
+gget.enrichr(
+ genes = [
+ "PHF14", "RBM3", "MSL1", "PHF21A", "ARL10", "INSR", "JADE2", "P2RX7",
+ "LINC00662", "CCDC101", "PPM1B", "KANSL1L", "CRYZL1", "ANAPC16", "TMCC1",
+ "CDH8", "RBM11", "CNPY2", "HSPA1L", "CUL2", "PLBD2", "LARP7", "TECPR2",
+ "ZNF302", "CUX1", "MOB2", "CYTH2", "SEC22C", "EIF4E3", "ROBO2",
+ "ADAMTS9-AS2", "CXXC1", "LINC01314", "ATF7", "ATP5F1"
+ ],
+ database = "ChEA_2022",
+ background_list = [
+ "NSUN3","POLRMT","NLRX1","SFXN5","ZC3H12C","SLC25A39","ARSG",
+ "DEFB29","PCMTD2","ACAA1A","LRRC1","2810432D09RIK","SEPHS2",
+ "SAC3D1","TMLHE","LOC623451","TSR2","PLEKHA7","GYS2","ARHGEF12",
+ "HIBCH","LYRM2","ZBTB44","ENTPD5","RAB11FIP2","LIPT1",
+ "INTU","ANXA13","KLF12","SAT2","GAL3ST2","VAMP8","FKBPL",
+ "AQP11","TRAP1","PMPCB","TM7SF3","RBM39","BRI3","KDR","ZFP748",
+ "NAP1L1","DHRS1","LRRC56","WDR20A","STXBP2","KLF1","UFC1",
+ "CCDC16","9230114K14RIK","RWDD3","2610528K11RIK","ACO1",
+ "CABLES1", "LOC100047214","YARS2","LYPLA1","KALRN","GYK",
+ "ZFP787","ZFP655","RABEPK","ZFP650","4732466D17RIK","EXOSC4",
+ "WDR42A","GPHN","2610528J11RIK","1110003E01RIK","MDH1","1200014M14RIK",
+ "AW209491","MUT","1700123L14RIK","2610036D13RIK",
+ "PHF14", "RBM3", "MSL1", "PHF21A", "ARL10", "INSR", "JADE2",
+ "P2RX7", "LINC00662", "CCDC101", "PPM1B", "KANSL1L", "CRYZL1",
+ "ANAPC16", "TMCC1","CDH8", "RBM11", "CNPY2", "HSPA1L", "CUL2",
+ "PLBD2", "LARP7", "TECPR2", "ZNF302", "CUX1", "MOB2", "CYTH2",
+ "SEC22C", "EIF4E3", "ROBO2", "ADAMTS9-AS2", "CXXC1", "LINC01314", "ATF7",
+ "ATP5F1""COX15","TMEM30A","NSMCE4A","TM2D2","RHBDD3","ATXN2","NFS1",
+ "3110001I20RIK","BC038156","C330002I19RIK","ZFYVE20","POLI","TOMM70A",
+ "LOC100047782","2410012H22RIK","RILP","A230062G08RIK",
+ "PTTG1IP","RAB1","AFAP1L1", "LYRM5","2310026E23RIK",
+ "SLC7A6OS","MAT2B","4932438A13RIK","LRRC8A","SMO","NUPL2"
+ ],
+ plot=True
+)
+→ Provée factores de transcripción relacionados a los genes de interés y controlados con la lista de genes background de la base de datos ChEA 2022. En Python, plot=True permite la visualización de resultados:

Genere una imagen de la vía de señalización de células KEGG con los genes del análisis de enriquecimiento resaltados:
+Esta función está disponible gracias a un PR de Noriaki Sato.
gget enrichr -db pathway --kegg_out kegg.png --kegg_rank 1 ZBP1 IRF3 RIPK1
+# Python
+gget.enrichr(["ZBP1", "IRF3", "RIPK1"], database="pathway", kegg_out="kegg.png", kegg_rank=1)
+→ Además de los resultados estándar gget enrichr, el argumento kegg_out guarda una imagen con los genes del análisis de enriquecimiento resaltados guardado como kegg.png:
El siguiente ejemplo fue enviado por Dylan Lawless a través de un PR (con ajustes de Laura Luebbert):
+Use gget enrichr en R y cree unq visualización similar usando ggplot.
+TENGA EN CUENTA el cambio de ejes en comparación con la visualización en Python.
system("pip install gget")
+install.packages("reticulate")
+library(reticulate)
+gget <- import("gget")
+
+# Perform enrichment analysis on a list of genes
+df <- gget$enrichr(list("ACE2", "AGT", "AGTR1"), database = "ontology")
+
+# Count number of overlapping genes
+df$overlapping_genes_count <- lapply(df$overlapping_genes, length) |> as.numeric()
+
+# Only keep the top 15 results
+df <- df[1:15, ]
+
+# Plot
+library(ggplot2)
+
+df |>
+ ggplot() +
+ geom_bar(aes(
+ x = -log10(adj_p_val),
+ y = reorder(path_name, -adj_p_val)
+ ),
+ stat = "identity",
+ fill = "lightgrey",
+ width = 0.5,
+ color = "black") +
+ geom_text(
+ aes(
+ y = path_name,
+ x = (-log10(adj_p_val)),
+ label = overlapping_genes_count
+ ),
+ nudge_x = 0.75,
+ show.legend = NA,
+ color = "red"
+ ) +
+ geom_text(
+ aes(
+ y = Inf,
+ x = Inf,
+ hjust = 1,
+ vjust = 1,
+ label = "# of overlapping genes"
+ ),
+ show.legend = NA,
+ color = "red"
+ ) +
+ geom_vline(linetype = "dotted", linewidth = 1, xintercept = -log10(0.05)) +
+ ylab("Pathway name") +
+ xlab("-log10(adjusted P value)")
+++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Genera texto en lenguaje natural basado en mensaje de entrada. gget gpt use la API 'openai.ChatCompletion.create' de OpenAI.
+Este módulo, incluido su código, documentación y pruebas unitarias, fue escrito en parte por Chat-GTP3 de OpenAI.
TENGA EN CUENTA:
+Las llamadas a la API de OpenAI solo son 'gratuitas' durante los primeros tres meses después de generar su cuenta de OpenAI (OpenAI proporciona un crédito de $5 que vence).
+Puede definir un límite de facturación mensual estricto (por ejemplo, $1) aquí.
+Vea sus precios y preguntas frecuentes aquí.
+Obtenga su clave API de OpenAI aquí.
Regresa: El texto generado (str).
+Antes de usar gget gpt por primera vez, corre gget setup gpt / gget.setup("gpt") (ver también gget setup).
Parámetros posicionales
+prompt
+Mensaje de entrada basado en el cual generar texto (str).
api_key
+Su clave API de OpenAI (str) (obtenga su clave API).
Parámetros optionales
+-m --model
+El nombre del algoritmo GPT que se usará para generar el texto (str). Por defecto: "gpt-3.5-turbo".
+See https://platform.openai.com/docs/models/gpt-4 for more information on the available models.
-temp --temperature
+Valor entre 0 y 2 que controla el nivel de aleatoriedad y creatividad en el texto generado (float).
+Los valores más altos resultan en un texto más creativo y variado. Por defecto: 1.
-tp --top_p
+Controla la diversidad del texto generado como alternativa al muestreo con --temperature (float).
+Los valores más altos resultan en un texto más diverso e inesperado. Por defecto: 1.
+Tenga en cuenta que OpenAI recomienda modificar --top_p o el parámetro --temperature, pero no ambas.
-s --stop
+Una secuencia de tokens para marcar el final del texto generado (str). Por defecto: None.
-mt --max_tokens
+Controla la longitud máxima del texto generado, en tokens (int). Por defecto: 200.
-pp --presence_penalty
+Número entre -2.0 y 2.0. Los valores más altos aumentan la probabilidad de que el modelo hable sobre temas nuevos (float). Por defecto: 0.
-fp --frequency_penalty
+Número entre -2.0 y 2.0. Los valores más altos reducen la probabilidad de que el modelo repita la misma línea palabra por palabra (float). Por defecto: 0.
-lb --logit_bias
+Un diccionario que especifica un sesgo hacia ciertos tokens en el texto generado (dict). Por defecto: None.
-o --out
+Ruta al archivo en el que se guardarán los resultados, p. ej. ruta/al/directorio/resultados.txt. Por defecto: salida estándar (STDOUT).
gget gpt "Cómo estás hoy GPT?" su_clave_api
+# Python
+print(gget.gpt("Cómo estás hoy GPT?", "su_clave_api"))
+ +
+
+
+ ++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Obtenga información detallada sobre genes y transcripciones de Ensembl, UniProt y NCBI utilizando sus IDs del tipo Ensembl.
+Regresa: Resultados en formato JSON (Terminal) o Dataframe/CSV (Python).
Parámetro posicional
+ens_ids
+Uno o más ID del tipo Ensembl.
Parámetros optionales
+-o --out
+Ruta al archivo en el que se guardarán los resultados, p. ej. ruta/al/directorio/resultados.csv (o .json). Por defecto: salida estándar (STDOUT).
+Para Python, usa save=True para guardar los resultados en el directorio de trabajo actual.
Banderas
+-n --ncbi
+DESACTIVA los resultados de NCBI.
+Para Python: ncbi=False evita la incluida de datos de NCBI (por defecto: True).
-u --uniprot
+DESACTIVA los resultados de UniProt.
+Para Python: uniprot=False evita la incluida de datos de UniProt (por defecto: True).
-pdb --pdb
+INCLUYE PDB IDs en los resultados (podría aumentar el tiempo de ejecución).
+Para Python: pdb=True incluye IDs de PDB en los resultados (por defecto: False).
-csv --csv
+Solo para la Terminal. Regresa los resultados en formato CSV.
+Para Python, usa json=True para regresar los resultados en formato JSON.
-q --quiet
+Solo para la Terminal. Impide la informacion de progreso de ser exhibida durante la corrida.
+Para Python, usa verbose=False para imipidir la informacion de progreso de ser exhibida durante la corrida.
wrap_text
+Solo para Python. wrap_text=True muestra los resultados con texto envuelto para facilitar la lectura (por defecto: False).
gget info ENSG00000034713 ENSG00000104853 ENSG00000170296
+# Python
+gget.info(["ENSG00000034713", "ENSG00000104853", "ENSG00000170296"])
+→ Regresa información detallada sobre cada ID de Ensembl ingresada:
+| uniprot_id | ncbi_gene_id | primary_gene_name | synonyms | protein_names | ensembl_description | uniprot_description | ncbi_description | biotype | canonical_transcript | ... | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ENSG00000034713 | P60520 | 11345 | GABARAPL2 | [ATG8, ATG8C, FLC3A, GABARAPL2, GATE-16, GATE16, GEF-2, GEF2] | Gamma-aminobutyric acid receptor-associated protein like 2 (GABA(A) receptor-associated protein-like 2)... | GABA type A receptor associated protein like 2 [Source:HGNC Symbol;Acc:HGNC:13291] | FUNCTION: Ubiquitin-like modifier involved in intra- Golgi traffic (By similarity). Modulates intra-Golgi transport through coupling between NSF activity and ... | Enables ubiquitin protein ligase binding activity. Involved in negative regulation of proteasomal protein catabolic process and protein... | protein_coding | ENST00000037243.7 | ... |
| . . . | . . . | . . . | . . . | . . . | . . . | . . . | . . . | . . . | . . . | . . . | ... |
pip install --upgrade gget
+Alternativa:
+conda install -c bioconda gget
+Para uso en Jupyter Lab / Google Colab:
+import gget
+gget es un programa gratuito de código fuente abierta de Terminal y Python que permite la consulta eficiente de bases de datos genómicas.
+
+gget consiste en un conjunto de módulos separados pero interoperables, cada uno diseñado para facilitar un tipo de consulta de base de datos en una sola línea de código.
+
+Las bases de datos consultadas por gget se actualizan continuamente, lo que a veces cambia su estructura. Los módulos gget se prueban automáticamente cada dos semanas y se actualizan para que coincidan con las nuevas estructuras de la base de datos cuando es necesario. Si encuentra algún problema, actualice a la última versión de gget usando pip install --upgrade gget. Si el problema persiste, informa el problema.
+
+
Solicitar una nueva función
+
Estos son los módulos principales de gget. Haga clic en cualquier módulo para acceder a la documentación detallada.
| gget alphafold Predecir la estructura 3D de una proteína a partir de una secuencia de aminoácidos. |
+ gget archs4 ¿Cuál es la expresión de mi gen en el tejido X? |
+ gget bgee Encontrar todos los ortólogos de un gen. |
+ gget blast Realizar un BLAST de una secuencia de nucleótidos o aminoácidos. |
+
| gget blat Encontrar la ubicación genómica de una secuencia de nucleótidos o aminoácidos. |
+ gget cbio Explorar la expresión de un gen en los cánceres especificados. |
+ gget cellxgene Obtener matrices de conteo de ARN de células individuales listas para usar para ciertos tejidos/enfermedades/etc. |
+ gget cosmic Buscar genes, mutaciones y otros factores asociados con ciertos cánceres. |
+
| gget diamond Alinear secuencias de aminoácidos a una referencia. |
+ gget elm Encontrar dominios y funciones de interacción de proteínas en una secuencia de aminoácidos. |
+ gget enrichr Verificar si una lista de genes está asociada con un tipo celular específico/ vía/ enfermedad/ etc. |
+ gget info Recuperar toda la información asociada con un ID de Ensembl. |
+
| gget muscle Alinear múltiples secuencias de nucleótidos o aminoácidos entre sí. |
+ gget mutate Mutar secuencias de nucleótidos según mutaciones específicas. |
+ gget opentargets Explorar qué enfermedades y medicamentos están asociados con un gen. |
+ gget pdb Recuperar datos de la Base de Datos de Proteínas (PDB) según un ID de PDB. |
+
| gget ref Obtener genomas de referencia de Ensembl. |
+ gget search Encontrar IDs de Ensembl asociados con la palabra de búsqueda especificada. |
+ gget seq Recuperar la secuencia de nucleótidos o aminoácidos de un gen. |
+
Si usa gget en una publicación, por favor cite*:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Lea el artículo aquí: https://doi.org/10.1093/bioinformatics/btac836
+Gracias a Victor Garcia-Ruiz y Anna Karen Orta por su ayuda con la traduccion del sitio web.
+++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Alinea múltiples secuencias de nucleótidos o aminoácidos usando el algoritmo Muscle5.
+Regresa: Salida estándar (STDOUT) en formato ClustalW o archivo de tipo 'aligned FASTA' (.afa).
Parámetro posicional
+fasta
+Lista de secuencias o ruta al archivo FASTA o .txt que contiene las secuencias de nucleótidos o aminoácidos que se van a alinear.
Parámetros optionales
+-o --out
+Ruta al archivo en el que se guardarán los resultados, p. ej. ruta/al/directorio/resultados.afa. Por defecto: salida estándar (STDOUT).
+Para Python, usa save=True para guardar los resultados en el directorio de trabajo actual.
Banderas
+-s5 --super5
+Alinea las secuencies usando el algoritmo Super5 en lugar del algoritmo Parallel Perturbed Probcons (PPP) para disminuir el tiempo y la memoria usada durante la corrida.
+Use para ingresos grandes (unos cientos secuencias).
-q --quiet
+Solo para la Terminal. Impide la informacion de progreso de ser exhibida durante la corrida.
+Para Python, usa verbose=False para imipidir la informacion de progreso de ser exhibida durante la corrida.
gget muscle MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS
+# Python
+gget.muscle(["MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS", "MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS"])
+gget muscle fasta.fa
+# Python
+gget.muscle("fasta.fa")
+→ Regresa las secuencias alineadas con coloración ClustalW. (Para devolver un archivo FASTA alineado (.afa), use el argumento --out (o save=True en Python).) En este ejemplo, el archivo 'fasta.fa' incluye varias secuencias para alineación (por ejemplo, isoformas devueltas desde gget seq).

También puede ver archivos FASTA alineados devueltos por gget.muscle usando programas como alv:
# Python
+!pip install biopython
+!pip install alv
+from Bio import AlignIO
+import alv
+
+gget.muscle("fasta.fa", out="fasta_aligned.afa")
+msa = AlignIO.read("fasta_aligned.afa", "fasta")
+alv.view(msa)
+++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Recibe secuencias de nucleótidos y mutaciones (en anotación de mutación estándar) y devuelve versiones mutadas de las secuencias según las mutaciones proporcionadas.
+Formato de devolución: Guarda las secuencias mutadas en formato FASTA (o devuelve una lista que contiene las secuencias mutadas si out=None).
Este módulo fue coescrito por Joseph Rich.
+Parámetro posicional
+sequences
+Ruta al archivo FASTA que contiene las secuencias a mutar, por ejemplo, 'path/to/seqs.fa'.
+Los identificadores de secuencia que siguen al carácter '>' deben corresponder a los identificadores en la columna seq_ID de mutations.
+NOTA: Solo se utilizará la cadena que sigue al '>' hasta el primer espacio o punto como identificador de secuencia. -> Se ignorarán los números de versión de los IDs de Ensembl.
Ejemplo de formato del archivo FASTA:
+>seq1 (o ENSG00000106443)
+ACTGCGATAGACT
+>seq2
+AGATCGCTAG
+Alternativamente: Proporcione secuencia(s) como una cadena o lista, por ejemplo, 'AGCTAGCT'.
+Otros parámetros requeridos
+-m --mutations
+Ruta al archivo csv o tsv (por ejemplo, 'path/to/mutations.csv') o marco de datos (objeto DataFrame) que contiene información sobre las mutaciones en el siguiente formato (la columna 'notas' no es necesaria):
| mutation | mut_ID | seq_ID | notas |
|---|---|---|---|
| c.2C>T | mut1 | seq1 | -> Aplicar la mutación 1 a la secuencia 1 |
| c.9_13inv | mut2 | seq2 | -> Aplicar la mutación 2 a la secuencia 2 |
| c.9_13inv | mut2 | seq4 | -> Aplicar la mutación 2 a la secuencia 4 |
| c.9_13delinsAAT | mut3 | seq4 | -> Aplicar la mutación 3 a la secuencia 4 |
| ... | ... | ... |
'mutation' = Columna que contiene las mutaciones a realizar, escritas en anotación de mutación estándar
+'mut_ID' = Columna que contiene el identificador de cada mutación
+'seq_ID' = Columna que contiene los identificadores de las secuencias a mutar (deben corresponder a la cadena que sigue al carácter '>' en el archivo FASTA de 'sequences'; NO incluir espacios ni puntos)
Alternativamente: Mutación(es) de entrada como una cadena o lista, por ejemplo, 'c.2C>T'.
+Si se proporciona una lista, el número de mutaciones debe ser igual al número de secuencias de entrada.
+Para uso desde el terminal (bash): Encierre las anotaciones de mutación individuales entre comillas para evitar errores.
Parámetros opcionales
+-k --k
+Longitud de las secuencias que flanquean la mutación. Por defecto: 30.
+Si k > longitud total de la secuencia, se mantendrá toda la secuencia.
-mc --mut_column
+Nombre de la columna que contiene las mutaciones a realizar en mutations. Por defecto: 'mutation'.
-mic --mut_id_column
+Nombre de la columna que contiene los IDs de cada mutación en mutations. Por defecto: 'mut_ID'.
-sic --seq_id_column
+Nombre de la columna que contiene los IDs de las secuencias a mutar en mutations. Por defecto: 'seq_ID'.
-o --out
+Ruta al archivo FASTA de salida que contiene las secuencias mutadas, por ejemplo, 'path/to/output_fasta.fa'.
+Por defecto: None -> devuelve una lista de las secuencias mutadas a la salida estándar.
+Los identificadores (después del '>') de las secuencias mutadas en el FASTA de salida serán '>[seq_ID]_[mut_ID]'.
Flags
+-q --quiet
+Solo para Terminal. Impide la información de progreso de ser exhibida durante la ejecución del programa.
+Para Python, usa verbose=False para imipidir la información de progreso de ser exhibida durante la ejecución del programa.
gget mutate ATCGCTAAGCT -m 'c.4G>T'
+# Python
+gget.mutate("ATCGCTAAGCT", "c.4G>T")
+→ Devuelve ATCTCTAAGCT.
+Lista de secuencias con una mutación para cada secuencia proporcionada en una lista:
+gget mutate ATCGCTAAGCT TAGCTA -m 'c.4G>T' 'c.1_3inv' -o mut_fasta.fa
+# Python
+gget.mutate(["ATCGCTAAGCT", "TAGCTA"], ["c.4G>T", "c.1_3inv"], out="mut_fasta.fa")
+→ Guarda el archivo 'mut_fasta.fa' que contiene:
+>seq1_mut1
+ATCTCTAAGCT
+>seq2_mut2
+GATCTA
+Una mutación aplicada a varias secuencias con k ajustado:
+gget mutate ATCGCTAAGCT TAGCTA -m 'c.1_3inv' -k 3
+# Python
+gget.mutate(["ATCGCTAAGCT", "TAGCTA"], "c.1_3inv", k=3)
+→ Devuelve ['CTAGCT', 'GATCTA'].
+ +++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Obtenga la estructura o los metadatos de una proteína usando data de RCSB Protein Data Bank (PDB).
+Regresa: El archivo 'pdb' se regresa en formato PDB. Todos los demás datos se regresan en formato JSON.
Parámetro posicional
+pdb_id
+ID del tipo PDB, p. ej. '7S7U'.
Parámetros optionales
+-r --resource
+Define el tipo de información a regresar. Uno de los siguientes:
+'pdb': Regresa la estructura de la proteína en formato PDB (regresa por defecto).
+'entry': Regresa información sobre las estructuras PDB en el nivel superior de la organización de datos PDB jerárquicos.
+'pubmed': Regresa anotaciones de PubMed (datos integrados de PubMed) para la cita principal de un ID PDB.
+'assembly': Regresa información sobre estructuras PDB en el nivel de estructura cuaternaria.
+'branched_entity': Regresa la descripción de la entidad ramificada (defina el ID de la entidad como identifier).
+'nonpolymer_entity': Regresa datos de entidades no poliméricas (defina el ID de la entidad como identifier).
+'polymer_entity': Regresa datos de entidades poliméricas (defina el ID de la entidad como identifier).
+'uniprot': Regresa anotaciones UniProt para una entidad macromolecular (defina el ID de la entidad como identifier).
+'branched_entity_instance': Regresa la descripción de instancia de entidad ramificada (defina el ID de cadena como identifier).
+'polymer_entity_instance': Regresa datos de instancia de entidad polimérica (también conocida como cadena) (defina el ID de cadena como identifier).
+'nonpolymer_entity_instance': Regresa datos de instancia de entidad no polimérica (defina el ID de cadena como identifier).
-i --identifier
+Este parámetro se puede utilizar para definir el ID de ensamblaje, entidad o cadena (po defecto: None). Los IDs de ensamblaje/entidad son números (p. ej., 1) y los IDs de cadena son letras (p. ej., 'A').
-o --out
+Ruta al archivo en el que se guardarán los resultados, p. ej. ruta/al/directorio/7S7U.pdb (o 7S7U_entry.json). Por defecto: salida estándar (STDOUT).
+Para Python, usa save=True para guardar los resultados en el directorio de trabajo actual.
gget pdb 7S7U -o 7S7U.pdb
+# Python
+gget.pdb("7S7U", save=True)
+→ Guarda la estructura de 7S7U en formato PDB como '7S7U.pdb' en el directorio de trabajo actual.
+Encuentre estructuras cristalinas de PDB para un análisis comparativo de la estructura de proteínas:
+# Encuentre IDs de PDB asociados con un ID de Ensembl
+gget info ENSG00000130234
+
+# Alternativamente: como que muchas entradas en el PDB no tienen ID de Ensembl vinculados,
+# es probable que encuentre más entradas de PDB BLASTing la secuencia contra el PDB:
+
+# Obtenga la secuencia de aminoácidos
+gget seq --translate ENSG00000130234 -o gget_seq_results.fa
+
+# BLAST la secuencia de aminoácidos para encontrar estructuras similares en el PDB
+gget blast --database pdbaa gget_seq_results.fa
+
+# Obtenga archivos PDB de los IDs de PDB regresados por gget blast para un análisis comparativo
+gget pdb 7DQA -o 7DQA.pdb
+gget pdb 7CT5 -o 7CT5.pdb
+# Encuentre IDs de PDB asociados con un ID de Ensembl
+gget.info("ENSG00000130234")
+
+# Alternativamente: como que muchas entradas en el PDB no tienen ID de Ensembl vinculados,
+# es probable que encuentre más entradas de PDB BLASTing la secuencia contra el PDB:
+
+# Obtenga la secuencia de aminoácidos
+gget.seq("ENSG00000130234", translate=True, save=True)
+
+# BLAST la secuencia de aminoácidos para encontrar estructuras similares en el PDB
+gget.blast("gget_seq_results.fa", database="pdbaa")
+
+# Obtenga archivos PDB de los IDs de PDB regresados por gget blast para un análisis comparativo
+gget.pdb("7DQA", save=True)
+gget.pdb("7CT5", save=True)
+→ Este caso de uso ejemplifica cómo encontrar archivos PDB para un análisis comparativo de la estructura de las proteínas asociado con IDs de Ensembl o secuencias de aminoácidos. Los archivos PDB obtenidos también se pueden comparar con las estructuras predichas generadas por gget alphafold. Los archivos PDB se pueden ver de forma interactiva en 3D aquí, o usando programas como PyMOL o Blender. Múltiple archivos PDB se pueden visualizar para comparación aquí.
Terminal:
+# Obtenga todos los FTP de anotaciones y referencias de Homo sapiens de la última versión de Ensembl
+$ gget ref homo_sapiens
+
+# Obtenga IDs de Ensembl de genes humanos con "ace2" o "angiotensin converting enzyme 2" en su nombre/descripción
+$ gget search -s homo_sapiens 'ace2' 'angiotensin converting enzyme 2'
+
+# Busque el gen ENSG00000130234 (ACE2) y su transcripción ENST00000252519
+$ gget info ENSG00000130234 ENST00000252519
+
+# Obtenga la secuencia de aminoácidos de la transcripción canónica del gen ENSG00000130234
+$ gget seq --translate ENSG00000130234
+
+# Rápidamente encuentra la ubicación genómica de la secuencia de aminoácidos
+$ gget blat MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS
+
+# BLAST la secuencia de aminoácidos
+$ gget blast MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS
+
+# Alinee múltiples secuencias de nucleótidos o aminoácidos entre sí (también acepta la ruta al archivo FASTA)
+$ gget muscle MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS
+
+# Alinee una o más secuencias de aminoácidos con una referencia (que contiene una o más secuencias) (BLAST local) (también acepta rutas a archivos FASTA)
+$ gget diamond MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS -ref MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS
+
+# Alinea secuencias de nucleótidos o aminoácidos en un archivo FASTA
+$ gget muscle path/to/file.fa
+
+# Use Enrichr para un análisis de ontología de una lista de genes
+$ gget enrichr -db ontology ACE2 AGT AGTR1 ACE AGTRAP AGTR2 ACE3P
+
+# Obtene la expresión en tejido humano del gen ACE2
+$ gget archs4 -w tissue ACE2
+
+# Obtenga la estructura de la proteína (en formato PDB) de ACE2 (ID de PDB devuelta por gget info)
+$ gget pdb 1R42 -o 1R42.pdb
+
+# Encuentre motivos lineales eucarióticos (ELM) en una secuencia de aminoácidos
+$ gget setup elm # solo debe ejecutarse una vez
+$ gget elm -o results MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS
+
+# Obtene una matriz de recuento de scRNAseq (formato AnnData) basada en genes, tejidos y tipos de células especificados (especie predeterminada: humano)
+$ gget setup cellxgene # solo debe ejecutarse una vez
+$ gget cellxgene --gene ACE2 SLC5A1 --tissue lung --cell_type 'mucus secreting cell' -o example_adata.h5ad
+
+# Predice la estructura proteica de GFP a partir de su secuencia de aminoácidos
+$ gget setup alphafold # solo debe ejecutarse una vez
+$ gget alphafold MSKGEELFTGVVPILVELDGDVNGHKFSVSGEGEGDATYGKLTLKFICTTGKLPVPWPTLVTTFSYGVQCFSRYPDHMKQHDFFKSAMPEGYVQERTIFFKDDGNYKTRAEVKFEGDTLVNRIELKGIDFKEDGNILGHKLEYNYNSHNVYIMADKQKNGIKVNFKIRHNIEDGSVQLADHYQQNTPIGDGPVLLPDNHYLSTQSALSKDPNEKRDHMVLLEFVTAAGITHGMDELYK
+Python (Jupyter Lab / Google Colab):
+import gget
+gget.ref("homo_sapiens")
+gget.search(["ace2", "angiotensin converting enzyme 2"], "homo_sapiens")
+gget.info(["ENSG00000130234", "ENST00000252519"])
+gget.seq("ENSG00000130234", translate=True)
+gget.blat("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS")
+gget.blast("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS")
+gget.muscle(["MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS", "MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS"])
+gget.diamond("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS", reference="MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS")
+gget.enrichr(["ACE2", "AGT", "AGTR1", "ACE", "AGTRAP", "AGTR2", "ACE3P"], database="ontology", plot=True)
+gget.archs4("ACE2", which="tissue")
+gget.pdb("1R42", save=True)
+
+gget.setup("elm") # solo debe ejecutarse una vez
+ortho_df, regex_df = gget.elm("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS")
+
+gget.setup("cellxgene") # solo debe ejecutarse una vez
+gget.cellxgene(gene = ["ACE2", "SLC5A1"], tissue = "lung", cell_type = "mucus secreting cell")
+
+gget.setup("alphafold") # solo debe ejecutarse una vez
+gget.alphafold("MSKGEELFTGVVPILVELDGDVNGHKFSVSGEGEGDATYGKLTLKFICTTGKLPVPWPTLVTTFSYGVQCFSRYPDHMKQHDFFKSAMPEGYVQERTIFFKDDGNYKTRAEVKFEGDTLVNRIELKGIDFKEDGNILGHKLEYNYNSHNVYIMADKQKNGIKVNFKIRHNIEDGSVQLADHYQQNTPIGDGPVLLPDNHYLSTQSALSKDPNEKRDHMVLLEFVTAAGITHGMDELYK")
+Use a gget con R usando reticulate:
system("pip install gget")
+install.packages("reticulate")
+library(reticulate)
+gget <- import("gget")
+
+gget$ref("homo_sapiens")
+gget$search(list("ace2", "angiotensin converting enzyme 2"), "homo_sapiens")
+gget$info(list("ENSG00000130234", "ENST00000252519"))
+gget$seq("ENSG00000130234", translate=TRUE)
+gget$blat("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS")
+gget$blast("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS")
+gget$muscle(list("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS", "MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS"), out="out.afa")
+gget$diamond("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS", reference="MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS")
+gget$enrichr(list("ACE2", "AGT", "AGTR1", "ACE", "AGTRAP", "AGTR2", "ACE3P"), database="ontology")
+gget$archs4("ACE2", which="tissue")
+gget$pdb("1R42", save=TRUE)
+++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Obtenga enlaces FTP y sus respectivos metadatos (o use la bandera ftp para regresar solo los enlaces) para referenciar genomas y anotaciones de Ensembl.
+Regresa: Resultados en formato JSON.
Parámetro posicional
+species
+La especie por la cual que se buscará los FTP en el formato género_especies, p. ej. homo_sapiens.
+Nota: No se requiere cuando se llama a la bandera --list_species.
+Accesos directos: 'human', 'mouse', 'human_grch37' (accede al ensamblaje del genoma GRCh37)
Parámetros optionales
+-w --which
+Define qué resultados devolver. Por defecto: 'all' -> Regresa todos los resultados disponibles.
+Las entradas posibles son uno solo o una combinación de las siguientes (como lista separada por comas):
+'gtf' - Regresa la anotación (GTF).
+'cdna' - Regresa el transcriptoma (cDNA).
+'dna' - Regresa el genoma (DNA).
+'cds' - Regresa las secuencias codificantes correspondientes a los genes Ensembl. (No contiene UTR ni secuencia intrónica).
+'cdrna' - Regresa secuencias de transcripción correspondientes a genes de ARN no codificantes (ncRNA).
+'pep' - Regresa las traducciones de proteínas de los genes Ensembl.
-r --release
+Define el número de versión de Ensembl desde el que se obtienen los archivos, p. ej. 104. Default: latest Ensembl release.
-o --out
+Ruta al archivo en el que se guardarán los resultados, p. ej. ruta/al/directorio/resultados.json. Por defecto: salida estándar (STDOUT).
+Para Python, usa save=True para guardar los resultados en el directorio de trabajo actual.
Banderas
+-l --list_species
+Enumera todas las especies disponibles. (Para Python: combina con species=None.)
-ftp --ftp
+Regresa solo los enlaces FTP solicitados.
-d --download
+Solo para Terminal. Descarga los FTP solicitados al directorio actual (requiere curl para ser instalado).
-q --quiet
+Solo para la Terminal. Impide la informacion de progreso de ser exhibida durante la corrida.
+Para Python, usa verbose=False para imipidir la informacion de progreso de ser exhibida durante la corrida.
Use gget ref en combinación con kallisto | bustools para construir un índice de referencia:
kb ref -i INDEX -g T2G -f1 FASTA $(gget ref --ftp -w dna,gtf homo_sapiens)
+→ kb ref crea un índice de referencia utilizando los últimos archivos de ADN y GTF de especies Homo sapiens que le ha pasado gget ref.
Enumere todos los genomas disponibles de la versión 103 de Ensembl:
+gget ref --list_species -r 103
+# Python
+gget.ref(species=None, list_species=True, release=103)
+→ Regresa una lista con todos los genomas disponibles (gget ref verifica si GTF y FASTA están disponibles) de la versión 103 de Ensembl.
+(Si no se especifica ninguna versión, gget ref siempre devolverá información de la última versión de Ensembl).
Obtenga la referencia del genoma para una especie específica:
+gget ref -w gtf,dna homo_sapiens
+# Python
+gget.ref("homo_sapiens", which=["gtf", "dna"])
+→ Regresa un JSON con los últimos FTP humanos GTF y FASTA, y sus respectivos metadatos, en el formato:
+{
+ "homo_sapiens": {
+ "annotation_gtf": {
+ "ftp": "http://ftp.ensembl.org/pub/release-106/gtf/homo_sapiens/Homo_sapiens.GRCh38.106.gtf.gz",
+ "ensembl_release": 106,
+ "release_date": "28-Feb-2022",
+ "release_time": "23:27",
+ "bytes": "51379459"
+ },
+ "genome_dna": {
+ "ftp": "http://ftp.ensembl.org/pub/release-106/fasta/homo_sapiens/dna/Homo_sapiens.GRCh38.dna.primary_assembly.fa.gz",
+ "ensembl_release": 106,
+ "release_date": "21-Feb-2022",
+ "release_time": "09:35",
+ "bytes": "881211416"
+ }
+ }
+}
+++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Obtenga genes y transcripciones de Ensembl usando términos de búsqueda de forma libre.
+Los resultados se comparan según las secciones "nombre del gen" y "descripción" en la base de datos de Ensembl. gget versión >= 0.27.9 también incluye resultados que coinciden con la sección "sinónimo" de Ensembl.
+Regresa: Resultados en formato JSON (Terminal) o Dataframe/CSV (Python).
Parámetro posicional
+searchwords
+Una o más palabras de búsqueda de forma libre, p. ej. gaba nmda. (Nota: la búsqueda no distingue entre mayúsculas y minúsculas).
Otros parámetros requeridos
+-s --species
+Especies o base de datos a buscar.
+Una especie se puede pasar en el formato 'género_especie', p. ej. 'homo_sapiens' o 'arabidopsis_thaliana'.
+Para pasar una base de datos específica, pase el nombre de la base de datos CORE, p. ej. 'mus_musculus_dba2j_core_105_1'.
Todas las bases de datos disponibles para cada versión de Ensembl se pueden encontrar aquí:
+Vertebrados: http://ftp.ensembl.org/pub/current/mysql/
+Invertebrados: http://ftp.ensemblgenomes.org/pub/current/ + selecciona reino animal + selecciona mysql/
Accesos directos: 'human', 'mouse'
+Parámetros optionales
+-r --release
+Define el número de versión de Ensembl desde el que se obtienen los archivos, p. ej. 104. Por defecto: None -> se usa la última versión de Ensembl.
Nota: No se aplica a las especies invertebrados (en su lugar, puede pasar una base de datos de una especies específica (incluyen un número de versión) al argumento species). Para especies de invertebrados, Ensembl solo almacena bases de datos de 10 versiones anteriores a la versión actual.
Este argumento se sobrescribe si se pasa una base de datos específica (que incluye un número de publicación) al argumento species.
-t --id_type
+'gene' (esto se use por defecto) o 'transcript'
+Regesa genes o transcripciones, respectivamente.
-ao --andor
+'or' (esto se use por defecto) o 'and'
+'or' ('o'): Regresa todos los genes que INCLUYEN AL MENOS UNA de las palabras de búsqueda en su nombre/descripción.
+'and' ('y'): Regresa solo los genes que INCLUYEN TODAS las palabras de búsqueda en su nombre/descripción.
-l --limit
+Limita el número de resultados de búsqueda, p. ej. 10. Por defecto: None.
-o --out
+Ruta al archivo en el que se guardarán los resultados, p. ej. ruta/al/directorio/resultados.csv (o .json). Por defecto: salida estándar (STDOUT).
+Para Python, usa save=True para guardar los resultados en el directorio de trabajo actual.
Banderas
+-csv --csv
+Solo para la Terminal. Regresa los resultados en formato CSV.
+Para Python, usa json=True para regresar los resultados en formato JSON.
-q --quiet
+Solo para la Terminal. Impide la informacion de progreso de ser exhibida durante la corrida.
+Para Python, usa verbose=False para imipidir la informacion de progreso de ser exhibida durante la corrida.
wrap_text
+Solo para Python. wrap_text=True muestra los resultados con texto envuelto para facilitar la lectura (por defecto: False).
gget search -s human gaba gamma-aminobutyric
+# Python
+gget.search(["gaba", "gamma-aminobutyric"], "homo_sapiens")
+→ Regresa todos los genes que contienen al menos una de las palabras de búsqueda en su nombre o descripción de Ensembl/referencia externa:
+| ensembl_id | gene_name | ensembl_description | ext_ref_description | biotype | url |
|---|---|---|---|---|---|
| ENSG00000034713 | GABARAPL2 | GABA type A receptor associated protein like 2 [Source:HGNC Symbol;Acc:HGNC:13291] | GABA type A receptor associated protein like 2 | protein_coding | https://uswest.ensembl.org/homo_sapiens/Gene/Summary?g=ENSG00000034713 |
| . . . | . . . | . . . | . . . | . . . | . . . |
++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Obtenga la(s) secuencia(s) nucleótidos o aminoácidos de un gen (y todas sus isoformas) con su ID de Ensembl.
+Regresa: Archivo de tipo FASTA.
Parámetro posicional
+ens_ids
+One or more Ensembl IDs.
Parámetros optionales
+-o --out
+Ruta al archivo en el que se guardarán los resultados, p. ruta/al/directorio/resultados.fa. Por defecto: salida estándar (STDOUT).
+Para Python, usa save=True para guardar los resultados en el directorio de trabajo actual.
Banderas
+-t --translate
+Regresa secuencias de aminoácidos (en lugar de nucleótidos).
+Las secuencias de nucleótidos se obtienen de Ensembl.
+Las secuencias de aminoácidos se obtienen de UniProt.
-iso --isoforms
+Regresa las secuencias de todas las transcripciones conocidas.
+(Solo para IDs de genes).
-q --quiet
+Solo para la Terminal. Impide la informacion de progreso de ser exhibida durante la corrida.
+Para Python, usa verbose=False para imipidir la informacion de progreso de ser exhibida durante la corrida.
gget seq ENSG00000034713 ENSG00000104853 ENSG00000170296
+# Python
+gget.seq(["ENSG00000034713", "ENSG00000104853", "ENSG00000170296"])
+→ Regresa las secuencias de nucleótidos de ENSG00000034713, ENSG00000104853, y ENSG00000170296 en formato FASTA.
+gget seq -t -iso ENSG00000034713
+# Python
+gget.seq("ENSG00000034713", translate=True, isoforms=True)
+→ Regresa las secuencias de aminoácidos de todas las transcripciones conocidas de ENSG00000034713 en formato FASTA.
+++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Función para instalar/descargar dependencias de terceros para un módulo de gget.
+Parámetro posicional
+module
+Módulo gget para el que se deben instalar las dependencias.
gget setup alphafold
+# Python
+gget.setup("alphafold")
+→ Instala todas las dependencias de terceros (modificadas) y descarga los parámetros del algoritmo (~4 GB) necesarios para ejecutar gget alphafold.
Versión ≥ 0.28.6 (2 de junio de 2024):
+gget mutategget cosmic: Ahora puedes descargar bases de datos completas de COSMIC utilizando el argumento download_cosmicgget ref: Ahora puede obtener la ensambladura del genoma GRCh27 usando species='human_grch37'gget search: Ajusta el acceso a los datos humanos a la estructura de la versión 112 de Ensembl (corrige issue 129)Version ≥ 0.28.5 (May 29, 2024):
gget.setup("alphafold") + mutaciones de inversión en gget mutate solo invierten la cadena en lugar de también calcular la hebra complementariaVersión ≥ 0.28.4 (31 de enero de 2024):
+gget setup: soluciona el error con la ruta del archivo al ejecutar gget.setup("elm") en el sistema operativo Windows.Versión ≥ 0.28.3 (22 de enero de 2024):
+gget search y gget ref ahora también admiten hongos 🍄, protistas 🌝 y metazoos de invertebrados 🐝 🐜 🐌 🐙 (además de vertebrados y plantas)gget cosmicgget enrichr: corrige puntos de dispersión duplicados en el gráfico cuando los nombres de las rutas están duplicadosgget elm:
+gget.gget setup: utilice el argumento out para especificar un directorio en el que se descargará la base de datos ELM. Completa esta solicitud de función.gget Diamond: El comando DIAMOND ahora se ejecuta con el indicador --ignore-warnings, lo que permite secuencias de nicho, como secuencias de aminoácidos que solo contienen caracteres de nucleótidos y secuencias repetidas. Esto también es válido para las alineaciones DIAMOND realizadas dentro de gget elm.gget ref y gget search: la versión actual de Ensembl se obtiene del nuevo archivo de versión en el sitio FTP de Ensembl para evitar errores durante la carga de nuevos lanzamientos.gget search:
+--ftp) se guardan en formato de archivo txt en lugar de json.gget ref:
+list_iv_species para enumerar todas las especies de invertebrados disponibles (se puede combinar con el argumento release para obtener todas las especies disponibles de una liberación específica de Ensembl)Versión ≥ 0.28.2 (15 de noviembre de 2023):
+gget info: devuelve un mensaje de error cuando el servidor NCBI falla por un motivo distinto a un error de recuperación (esto es un error en el lado del servidor en lugar de un error con gget)BeautifulSoupgget elm: Elimina instancias de falsos positivos y verdaderos negativos de los resultados devueltos.gget elm: agrega el argumento expandVersión ≥ 0.28.0 (5 de noviembre de 2023):
+gget muscle para agregar un tutorial sobre cómo visualizar secuencias con diferentes longitudes de nombres de secuencia + ligero cambio en la visualización devuelta para que sea un poco más sólida ante diferentes nombres de secuenciagget muscle ahora también permite una lista de secuencias como entrada (como alternativa a proporcionar la ruta a un archivo FASTA)gget cellxgene (corrige error)gget seq: permite nombres de genes faltantes (correccione [https://github.com/pachterlab/gget/issues/107](https://github.com/pachterlab/gget /números/107))gget enrichr: use el argumento kegg_out y kegg_rank para crear una imagen de la vía KEGG con los genes del análisis de enriquecimiento resaltados (gracias a [este PR](https ://github.com/pachterlab/gget/pull/106) por Noriaki Sato)gget elm y gget DiamondVersión ≥ 0.27.9 (7 de agosto de 2023):
+gget enrichr: use el argumento background_list para proporcionar una lista de genes 'background'gget search ahora también busca sinónimos Ensembl (además de nombres y descripciones de genes) para obtener resultados de búsqueda más completos (gracias a Samuel Klein por la sugerencia)Versión ≥ 0.27.8 (12 de julio de 2023):
+gget search: especifique la versión de Ensembl desde la cual se obtiene la información con -r --releasegget pdb (este error se introdujo en la versión 0.27.5)Versión ≥ 0.27.7 (15 de mayo de 2023):
+gget gpt y gget cellxgene de los requisitos instalados automáticamente a gget setupgget alphafold actualizadas para compatibilidad con Python >= 3.10census_version a gget cellxgeneVersión ≥ 0.27.6 (1 de mayo de 2023) (TIRO debido a problemas con las dependencias -> reemplazada por la versión 0.27.7):
+gget search ahora también puede consultar los ID de plantas 🌱 Ensemblgget cellxgeneVersión ≥ 0.27.5 (6 de abril de 2023):
+gget search para que funcione correctamente con la nueva versión de Pandas 2.0.0 (lanzado el 3 de abril de 2023), además de versiones anteriores de Pandasgget info con nuevos banderas uniprot y ncbi que permiten desactivar los resultados de estas bases de datos de forma independiente para ahorrar tiempo de ejecución (nota: el indicador ensembl_only quedó obsoleto)-q / --quiet (para Python: verbose=False) para desactivar la información de progresoVersión ≥ 0.27.4 (19 de marzo de 2023):
+gget gpt Versión ≥ 0.27.3 (11 de marzo de 2023):
+gget info excluye los ID de PDB de forma predeterminada para aumentar la velocidad (los resultados de PDB se pueden incluir usando la marca --pdb / pdb=True).Versión ≥ 0.27.2 (1 de enero de 2023):
+gget alphafold a DeepMind's AlphaFold v2.3.0 (incluidos los nuevos argumentos multimer_for_monomer y multimer_recycles)Versión ≥ 0.27.0 (10 de diciembre de 2022):
+gget alphafold para que coincida con los cambios recientes de DeepMindVersión ≥ 0.3.13 (11 de noviembre de 2022):
+gget enrichr y gget archs4 cuando se usa con ID de EnsemblVersión ≥ 0.3.12 (10 de noviembre de 2022):
+gget info ahora también devuelve datos de localización subcelular de UniProtgget info ensembl_only devuelve solo los resultados de Ensemblgget info y gget seqVersión ≥ 0.3.11 (7 de septiembre de 2022):
+gget pdbVersión ≥ 0.3.10 (2 de septiembre de 2022):
+gget alphafold ahora también devuelve valores pLDDT para generar gráficos sin volver a ejecutar el programa (consulte también las preguntas frecuentes de gget alphafold)Versión ≥ 0.3.9 (25 de agosto de 2022):
+gget alphafoldVersión ≥ 0.3.8 (12 de agosto de 2022):
+Versión ≥ 0.3.7 (9 de agosto de 2022):
+gget ≥ 0.3.7 si usa obtener refVersión ≥ 0.3.5 (6 de agosto de 2022):
+gget alphafoldVersión ≥ 0.2.6 (7 de julio de 2022):
+gget ref ahora admite genomas de plantas! 🌱Versión ≥ 0.2.5 (30 de junio de 2022):
+gget ≥ 0.2.5 si usa alguno de los módulos que consultan datos de UniProt (gget info y gget seq).Versión ≥ 0.2.3: (26 de junio de 2022):
+[-csv][--csv]). El formato data frame/CSV sigue siendo el formato de regreso predeterminada para Python (Jupyter Lab/Google Colab) (y se puede convertir a JSON con json=True).gget ref -s human → gget ref human.gget info: [--expand] está en desuso. El módulo ahora siempre devolverá toda la información disponible.gget info, incluida la devolución de los ID de Ensembl versionados.gget info y gget seq ahora son compatibles con las IDs de WormBase y FlyBase.gget archs4 y gget enrichr con la bandera [-e][--ensembl] (ensembl=True para Python (Jupyter Lab / Google Colab)).seqtype de gget seq fue reemplazado por la bandera [-t][--translate] (translate=True/False para Python (Jupyter Lab / Google Colab)) que devolverá secuencias de nucleótidos (False) o aminoácidos (True).seqtype de gget search se renombró a id_type (aún tomando los mismos parámetros 'gene' o 'transcript').gget is a free, open-source command-line tool and Python package that enables efficient querying of genomic databases.
+
+gget consists of a collection of separate but interoperable modules, each designed to facilitate one type of database querying in a single line of code.
+
+NOTE: The databases queried by gget are continuously being updated which sometimes changes their structure. gget modules are tested automatically on a biweekly basis and updated to match new database structures when necessary. If you encounter a problem, please upgrade to the latest gget version using pip install --upgrade gget. If the problem persists, please report the issue.
+
+
Request a new feature
+
+
+++
gget infoandgget seqare currently unable to fetch information for WormBase and FlyBase IDs (all other IDs are functioning normally). This issue arose due to a bug in Ensembl release 112. We appreciate Ensembl's efforts in addressing this issue and expect a fix soon. Thank you for your patience.
These are the gget core modules. Click on any module to access detailed documentation.
| gget alphafold Predict 3D protein structure from an amino acid sequence. |
+ gget archs4 What is the expression of my gene in tissue X? |
+ gget bgee Find all orthologs of a gene. |
+ gget blast BLAST a nucleotide or amino acid sequence. |
+
| gget blat Find the genomic location of a nucleotide or amino acid sequence. |
+ gget cbio Explore a gene's expression in the specified cancers. |
+ gget cellxgene Get ready-to-use single-cell RNA seq count matrices from certain tissues/ diseases/ etc. |
+ gget cosmic Search for genes, mutations, and other factors associated with certain cancers. |
+
| gget diamond Align amino acid sequences to a reference. |
+ gget elm Find protein interaction domains and functions in an amino acid sequence. |
+ gget enrichr Check if a list of genes is associated with a specific celltype/ pathway/ disease/ etc. |
+ gget info Fetch all of the information associated with an Ensembl ID. |
+
| gget muscle Align multiple nucleotide or amino acid sequences to each other. |
+ gget mutate Mutate nucleotide sequences based on specified mutations. |
+ gget opentargets Explore which diseases and drugs a gene is associated with. |
+ gget pdb Fetch data from the Protein Data Bank (PDB) based on a PDB ID. |
+
| gget ref Get reference genomes from Ensembl. |
+ gget search Find Ensembl IDs associated with the specified search word. |
+ gget seq Fetch the nucleotide or amino acid sequence of a gene. |
+
If you use gget in a publication, please cite*:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Read the article here: https://doi.org/10.1093/bioinformatics/btac836
+gget is a free, open-source command-line tool and Python package that enables efficient querying of genomic databases.
+
+gget consists of a collection of separate but interoperable modules, each designed to facilitate one type of database querying in a single line of code.
+
+NOTE: The databases queried by gget are continuously being updated which sometimes changes their structure. gget modules are tested automatically on a biweekly basis and updated to match new database structures when necessary. If you encounter a problem, please upgrade to the latest gget version using pip install --upgrade gget. If the problem persists, please report the issue.
+
+
Request a new feature
+
+
+++
gget infoandgget seqare currently unable to fetch information for WormBase and FlyBase IDs (all other IDs are functioning normally). This issue arose due to a bug in Ensembl release 112. We appreciate Ensembl's efforts in addressing this issue and expect a fix soon. Thank you for your patience.
These are the gget core modules. Click on any module to access detailed documentation.
| gget alphafold Predict 3D protein structure from an amino acid sequence. |
+ gget archs4 What is the expression of my gene in tissue X? |
+ gget bgee Find all orthologs of a gene. |
+ gget blast BLAST a nucleotide or amino acid sequence. |
+
| gget blat Find the genomic location of a nucleotide or amino acid sequence. |
+ gget cbio Explore a gene's expression in the specified cancers. |
+ gget cellxgene Get ready-to-use single-cell RNA seq count matrices from certain tissues/ diseases/ etc. |
+ gget cosmic Search for genes, mutations, and other factors associated with certain cancers. |
+
| gget diamond Align amino acid sequences to a reference. |
+ gget elm Find protein interaction domains and functions in an amino acid sequence. |
+ gget enrichr Check if a list of genes is associated with a specific celltype/ pathway/ disease/ etc. |
+ gget info Fetch all of the information associated with an Ensembl ID. |
+
| gget muscle Align multiple nucleotide or amino acid sequences to each other. |
+ gget mutate Mutate nucleotide sequences based on specified mutations. |
+ gget opentargets Explore which diseases and drugs a gene is associated with. |
+ gget pdb Fetch data from the Protein Data Bank (PDB) based on a PDB ID. |
+
| gget ref Get reference genomes from Ensembl. |
+ gget search Find Ensembl IDs associated with the specified search word. |
+ gget seq Fetch the nucleotide or amino acid sequence of a gene. |
+
If you use gget in a publication, please cite*:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Read the article here: https://doi.org/10.1093/bioinformatics/btac836
+Version ≥ 0.29.0 (Sep 25, 2024):
+gget enrichr now also supports species other than human and mouse (fly, yeast, worm, and fish) via modEnrichRgget mutate:gget mutate will now merge identical sequences in the final file by default. Mutation creation was vectorized to decrease runtime. Improved flanking sequence check for non-substitution mutations to make sure no wildtype kmer is retained in the mutation-containing sequence. Addition of several new arguments to customize sequence generation and output.gget cosmic:gget ref:gget info and gget seq:Version ≥ 0.28.6 (Jun 2, 2024):
+gget mutategget cosmic: You can now download entire COSMIC databases using the argument download_cosmic argumentgget ref: Can now fetch the GRCh37 genome assembly using species='human_grch37'gget search: Adjust access of human data to the structure of Ensembl release 112 (fixes issue 129)Version ≥ 0.28.5 (May 29, 2024):
gget.setup("alphafold") + inversion mutations in gget mutate only reverse the string instead of also computing the complementary strandVersion ≥ 0.28.4 (January 31, 2024):
+gget setup: Fix bug with filepath when running gget.setup("elm") on Windows OS.Version ≥ 0.28.3 (January 22, 2024):
+gget search and gget ref now also support fungi 🍄, protists 🌝, and invertebrate metazoa 🐝 🐜 🐌 🐙 (in addition to vertebrates and plants)gget cosmicgget enrichr: Fix duplicate scatter dots in plot when pathway names are duplicatedgget elm:
+gget modules.gget setup: Use the out argument to specify a directory the ELM database will be downloaded into. Completes this feature request.gget diamond: The DIAMOND command is now run with --ignore-warnings flag, allowing niche sequences such as amino acid sequences that only contain nucleotide characters and repeated sequences. This is also true for DIAMOND alignments performed within gget elm.gget ref and gget search back-end change: the current Ensembl release is fetched from the new release file on the Ensembl FTP site to avoid errors during uploads of new releases.gget search:
+--ftp) are saved in txt file format instead of json.gget ref:
+list_iv_species to list all available invertebrate species (can be combined with the release argument to fetch all species available from a specific Ensembl release)Version ≥ 0.28.2 (November 15, 2023):
+gget info: Return a logging error message when the NCBI server fails for a reason other than a fetch fail (this is an error on the server side rather than an error with gget)BeautifulSoupgget elm: Remove false positive and true negative instances from returned resultsgget elm: Add expand argumentVersion ≥ 0.28.0 (November 5, 2023):
+gget muscle to add a tutorial on how to visualize sequences with varying sequence name lengths + slight change to returned visualization so it's a bit more robust to varying sequence namesgget muscle now also allows a list of sequences as input (as an alternative to providing the path to a FASTA file)gget cellxgene (fixes bug)gget seq: Allow missing gene names (fixes https://github.com/pachterlab/gget/issues/107)gget enrichr: Use new arguments kegg_out and kegg_rank to create an image of the KEGG pathway with the genes from the enrichment analysis highlighted (thanks to this PR by Noriaki Sato)gget elm and gget diamondVersion ≥ 0.27.9 (August 7, 2023):
+gget enrichr: Use new argument background_list to provide a list of background genesgget search now also searches Ensembl synonyms (in addition to gene descriptions and names) to return more comprehensive search results (thanks to Samuel Klein for the suggestion)Version ≥ 0.27.8 (July 12, 2023):
+gget search: Specify the Ensembl release from which information is fetched with new argument -r --releasegget pdb (this bug was introduced in version 0.27.5)Version ≥ 0.27.7 (May 15, 2023):
+gget gpt and gget cellxgene from automatically installed requirements to gget setup.gget alphafold dependencies for compatibility with Python >= 3.10.census_version argument to gget cellxgene.Version ≥ 0.27.6 (May 1, 2023) (YANKED due to problems with dependencies -> replaced with version 0.27.7):
+gget search can now also query plant 🌱 Ensembl IDs.gget cellxgeneVersion ≥ 0.27.5 (April 6, 2023):
+gget search to function correctly with new Pandas version 2.0.0 (released on April 3rd, 2023) as well as older versions of Pandasgget info with new flags uniprot and ncbi which allow turning off results from these databases independently to save runtime (note: flag ensembl_only was deprecated)-q / --quiet (Python: verbose=False) flag to turn off progress informationVersion ≥ 0.27.4 (March 19, 2023):
+gget gptVersion ≥ 0.27.3 (March 11, 2023):
+gget info excludes PDB IDs by default to increase speed (PDB results can be included using flag --pdb / pdb=True).Version ≥ 0.27.2 (January 1, 2023):
+gget alphafold to DeepMind's AlphaFold v2.3.0 (including new arguments multimer_for_monomer and multimer_recycles)Version ≥ 0.27.0 (December 10, 2022):
+gget alphafold to match recent changes by DeepMindVersion ≥ 0.3.13 (November 11, 2022):
+gget enrichr and gget archs4 when used with Ensembl IDsVersion ≥ 0.3.12 (November 10, 2022):
+gget info now also returns subcellular localisation data from UniProtgget info flag ensembl_only returns only Ensembl resultsgget info and gget seqVersion ≥ 0.3.11 (September 7, 2022):
+gget pdbVersion ≥ 0.3.10 (September 2, 2022):
+gget alphafold now also returns pLDDT values for generating plots from output without rerunning the program (also see the gget alphafold FAQ)Version ≥ 0.3.9 (August 25, 2022):
+gget alphafoldVersion ≥ 0.3.8 (August 12, 2022):
+Version ≥ 0.3.7 (August 9, 2022):
+gget version ≥ 0.3.7 if you use gget refVersion ≥ 0.3.5 (August 6, 2022):
+gget alphafoldVersion ≥ 0.2.6 (July 7, 2022):
+gget ref now supports plant genomes! 🌱Version ≥ 0.2.5 (June 30, 2022):
+gget version ≥ 0.2.5 if you use any of the modules querying data from UniProt (gget info and gget seq).Version ≥ 0.2.3: (June 26, 2022):
+[-csv][--csv]). Data frame/CSV remains the default output for Jupyter Lab / Google Colab (and can be converted to JSON with json=True).gget ref -s human → gget ref human.gget info: [--expand] is deprecated. The module will now always return all of the available information.gget info, including the return of versioned Ensembl IDs.gget info and gget seq now support 🪱 WormBase and 🪰 FlyBase IDs.gget archs4 and gget enrichr now also take Ensembl IDs as input with added flag [-e][--ensembl] (ensembl=True in Jupyter Lab / Google Colab).gget seq argument seqtype was replaced by flag [-t][--translate] (translate=True/False in Jupyter Lab / Google Colab) which will return either nucleotide (False) or amino acid (True) sequences.gget search argument seqtype was renamed to id_type for clarity (still taking the same arguments 'gene' or 'transcript').pip install --upgrade gget
+Alternative:
+conda install -c bioconda gget
+For use in Jupyter Lab / Google Colab:
+import gget
+Command line:
+# Fetch all Homo sapiens reference and annotation FTPs from the latest Ensembl release
+$ gget ref homo_sapiens
+
+# Get Ensembl IDs of human genes with "ace2" or "angiotensin converting enzyme 2" in their name/description
+$ gget search -s homo_sapiens 'ace2' 'angiotensin converting enzyme 2'
+
+# Look up gene ENSG00000130234 (ACE2) and its transcript ENST00000252519
+$ gget info ENSG00000130234 ENST00000252519
+
+# Fetch the amino acid sequence of the canonical transcript of gene ENSG00000130234
+$ gget seq --translate ENSG00000130234
+
+# Quickly find the genomic location of (the start of) that amino acid sequence
+$ gget blat MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS
+
+# BLAST (the start of) that amino acid sequence
+$ gget blast MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS
+
+# Align multiple nucleotide or amino acid sequences against each other (also accepts path to FASTA file)
+$ gget muscle MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS
+
+# Align one or more amino acid sequences against a reference (containing one or more sequences) (local BLAST) (also accepts paths to FASTA files)
+$ gget diamond MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS -ref MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS
+
+# Use Enrichr for an ontology analysis of a list of genes
+$ gget enrichr -db ontology ACE2 AGT AGTR1 ACE AGTRAP AGTR2 ACE3P
+
+# Get the human tissue expression of gene ACE2
+$ gget archs4 -w tissue ACE2
+
+# Get the protein structure (in PDB format) of ACE2 as stored in the Protein Data Bank (PDB ID returned by gget info)
+$ gget pdb 1R42 -o 1R42.pdb
+
+# Find Eukaryotic Linear Motifs (ELMs) in a protein sequence
+$ gget setup elm # setup only needs to be run once
+$ gget elm -o results MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS
+
+# Fetch a scRNAseq count matrix (AnnData format) based on specified gene(s), tissue(s), and cell type(s) (default species: human)
+$ gget setup cellxgene # setup only needs to be run once
+$ gget cellxgene --gene ACE2 SLC5A1 --tissue lung --cell_type 'mucus secreting cell' -o example_adata.h5ad
+
+# Predict the protein structure of GFP from its amino acid sequence
+$ gget setup alphafold # setup only needs to be run once
+$ gget alphafold MSKGEELFTGVVPILVELDGDVNGHKFSVSGEGEGDATYGKLTLKFICTTGKLPVPWPTLVTTFSYGVQCFSRYPDHMKQHDFFKSAMPEGYVQERTIFFKDDGNYKTRAEVKFEGDTLVNRIELKGIDFKEDGNILGHKLEYNYNSHNVYIMADKQKNGIKVNFKIRHNIEDGSVQLADHYQQNTPIGDGPVLLPDNHYLSTQSALSKDPNEKRDHMVLLEFVTAAGITHGMDELYK
+Python (Jupyter Lab / Google Colab):
+import gget
+gget.ref("homo_sapiens")
+gget.search(["ace2", "angiotensin converting enzyme 2"], "homo_sapiens")
+gget.info(["ENSG00000130234", "ENST00000252519"])
+gget.seq("ENSG00000130234", translate=True)
+gget.blat("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS")
+gget.blast("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS")
+gget.muscle(["MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS", "MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS"])
+gget.diamond("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS", reference="MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS")
+gget.enrichr(["ACE2", "AGT", "AGTR1", "ACE", "AGTRAP", "AGTR2", "ACE3P"], database="ontology", plot=True)
+gget.archs4("ACE2", which="tissue")
+gget.pdb("1R42", save=True)
+
+gget.setup("elm") # setup only needs to be run once
+ortho_df, regex_df = gget.elm("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS")
+
+gget.setup("cellxgene") # setup only needs to be run once
+gget.cellxgene(gene = ["ACE2", "SLC5A1"], tissue = "lung", cell_type = "mucus secreting cell")
+
+gget.setup("alphafold") # setup only needs to be run once
+gget.alphafold("MSKGEELFTGVVPILVELDGDVNGHKFSVSGEGEGDATYGKLTLKFICTTGKLPVPWPTLVTTFSYGVQCFSRYPDHMKQHDFFKSAMPEGYVQERTIFFKDDGNYKTRAEVKFEGDTLVNRIELKGIDFKEDGNILGHKLEYNYNSHNVYIMADKQKNGIKVNFKIRHNIEDGSVQLADHYQQNTPIGDGPVLLPDNHYLSTQSALSKDPNEKRDHMVLLEFVTAAGITHGMDELYK")
+Call gget from R using reticulate:
system("pip install gget")
+install.packages("reticulate")
+library(reticulate)
+gget <- import("gget")
+
+gget$ref("homo_sapiens")
+gget$search(list("ace2", "angiotensin converting enzyme 2"), "homo_sapiens")
+gget$info(list("ENSG00000130234", "ENST00000252519"))
+gget$seq("ENSG00000130234", translate=TRUE)
+gget$blat("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS")
+gget$blast("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS")
+gget$muscle(list("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS", "MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS"), out="out.afa")
+gget$diamond("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS", reference="MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS")
+gget$enrichr(list("ACE2", "AGT", "AGTR1", "ACE", "AGTRAP", "AGTR2", "ACE3P"), database="ontology")
+gget$archs4("ACE2", which="tissue")
+gget$pdb("1R42", save=TRUE)
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Predict the 3D structure of a protein from its amino acid sequence using a simplified version of DeepMind’s AlphaFold2 originally released and benchmarked for AlphaFold Colab.
+Returns: Predicted structure (PDB) and alignment error (json).
Before using gget alphafold for the first time:
conda install -qy conda==4.13.0 && conda install -qy -c conda-forge openmm=7.5.1openmm=7.7.0 for Python >= 3.10)conda update -qy conda to update conda to the latest version afterwards.gget setup alphafold / gget.setup("alphafold") once (also see gget setup). Running gget setup alphafold / gget.setup("alphafold") will download and install the latest version of AlphaFold2 hosted on the AlphaFold GitHub Repo. You can rerun this command any time to update the software after a new AlphaFold release.Positional argument
+sequence
+Amino acid sequence (str), or list of sequences (gget alphafold will automatically use the multimer model if multiple sequences are passed), or path to FASTA file.
Optional arguments
+-mr --multimer_recycles
+The multimer model will continue recycling until the predictions stop changing, up to the limit set here. Default: 3.
+For higher accuracy, at the potential cost of longer inference times, set this to 20.
-o --out
+Path to folder to save prediction results in (str). Default: "./[date_time]_gget_alphafold_prediction".
Flags
+-mfm --multimer_for_monomer
+Use multimer model for a monomer.
-r --relax
+AMBER relax the best model.
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
plot
+Python only. plot=True provides an interactive, 3D graphical overview of the predicted structure and alignment quality using py3Dmol and matplotlib (default: True).
show_sidechains
+Python only. show_sidechains=True includes side chains in the plot (default: True).
# Generate new prediction from amino acid sequence
+gget alphafold MAAHKGAEHHHKAAEHHEQAAKHHHAAAEHHEKGEHEQAAHHADTAYAHHKHAEEHAAQAAKHDAEHHAPKPH
+
+# Find similar sequences deposited on the PDB for comparative analysis
+gget blast --database pdbaa MAAHKGAEHHHKAAEHHEQAAKHHHAAAEHHEKGEHEQAAHHADTAYAHHKHAEEHAAQAAKHDAEHHAPKPH
+
+# Fetch the PDB files of similar structures returned by gget blast for comparison, to get a measure for model quality
+gget pdb 3UQ3 -o 3UQ3.pdb
+gget pdb 2K42 -o 2K42.pdb
+# Python
+gget.alphafold("MAAHKGAEHHHKAAEHHEQAAKHHHAAAEHHEKGEHEQAAHHADTAYAHHKHAEEHAAQAAKHDAEHHAPKPH")
+
+# Find similar sequences deposited on the PDB for comparative analysis
+gget.blast("MAAHKGAEHHHKAAEHHEQAAKHHHAAAEHHEKGEHEQAAHHADTAYAHHKHAEEHAAQAAKHDAEHHAPKPH", database="pdbaa")
+
+# Fetch the PDB files of similar structures returned by gget blast for comparison, to get a measure for model quality
+gget.pdb("3UQ3", save=True)
+gget.pdb("2K42", save=True)
+→ gget alphafold returns the predicted structure (PDB) and predicted alignment error (.json) in a new folder ("./[date_time]_gget_alphafold_prediction"). The use case above exemplifies how to use gget blast and gget pdb for a comparative analysis of the new prediction. PDB files can be viewed interactively in 3D online, or using programs like PyMOL or Blender. To compare two PDB files, you can use this website. The Python interface also returns interactive plots, which can be generated from the PDB and JSON as described in the gget alphafold FAQ Q4.
If you use gget alphafold in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Jumper, J., Evans, R., Pritzel, A. et al. Highly accurate protein structure prediction with AlphaFold. Nature 596, 583–589 (2021). https://doi.org/10.1038/s41586-021-03819-2
+And, if applicable:
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Find the most correlated genes to a gene of interest or find the gene's tissue expression atlas using ARCHS4.
+Return format: JSON (command-line) or data frame/CSV (Python).
Positional argument
+gene
+Short name (gene symbol) of gene of interest, e.g. STAT4.
+Alternatively: use flag --ensembl to input an Ensembl gene IDs, e.g. ENSG00000138378.
Optional arguments
+-w --which
+'correlation' (default) or 'tissue'.
+'correlation' returns a gene correlation table that contains the 100 most correlated genes to the gene of interest. The Pearson correlation is calculated over all samples and tissues in ARCHS4.
+'tissue' returns a tissue expression atlas calculated from human or mouse samples (as defined by 'species') in ARCHS4.
-s --species
+'human' (default) or 'mouse'.
+Defines whether to use human or mouse samples from ARCHS4.
+(Only for tissue expression atlas.)
-o --out
+Path to the file the results will be saved in, e.g. path/to/directory/results.csv (or .json). Default: Standard out.
+Python: save=True will save the output in the current working directory.
Flags
+-e --ensembl
+Add this flag if gene is given as an Ensembl gene ID.
-csv --csv
+Command-line only. Returns results in CSV format.
+Python: Use json=True to return output in JSON format.
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
gget archs4 ACE2
+# Python
+gget.archs4("ACE2")
+→ Returns the 100 most correlated genes to ACE2:
+| gene_symbol | pearson_correlation |
|---|---|
| SLC5A1 | 0.579634 |
| CYP2C18 | 0.576577 |
| . . . | . . . |
gget archs4 -w tissue ACE2
+# Python
+gget.archs4("ACE2", which="tissue")
+→ Returns the tissue expression of ACE2 (by default, human data is used):
+| id | min | q1 | median | q3 | max |
|---|---|---|---|---|---|
| System.Urogenital/Reproductive System.Kidney.RENAL CORTEX | 0.113644 | 8.274060 | 9.695840 | 10.51670 | 11.21970 |
| System.Digestive System.Intestine.INTESTINAL EPITHELIAL CELL | 0.113644 | 5.905560 | 9.570450 | 13.26470 | 13.83590 |
| . . . | . . . | . . . | . . . | . . . | . . . |
+Check out this tutorial by Dave Tang who wrote an R script to create this figure from the gget archs4 JSON output:
If you use gget archs4 in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Lachmann A, Torre D, Keenan AB, Jagodnik KM, Lee HJ, Wang L, Silverstein MC, Ma’ayan A. Massive mining of publicly available RNA-seq data from human and mouse. Nature Communications 9. Article number: 1366 (2018), doi:10.1038/s41467-018-03751-6
+Bray NL, Pimentel H, Melsted P and Pachter L, Near optimal probabilistic RNA-seq quantification, Nature Biotechnology 34, p 525--527 (2016). https://doi.org/10.1038/nbt.3519
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Fetch orthology and gene expression data from Bgee using Ensembl IDs.
+Return format: JSON/CSV (command-line) or data frame (Python).
++If you are specifically interested in human gene expression data, consider using gget opentargets or gget archs4 instead. +gget bgee has less data, but supports more species.
+
This module was written by Sam Wagenaar.
+Positional argument
+ens_id
+Ensembl gene ID, e.g. ENSG00000169194 or ENSSSCG00000014725.
Required arguments
+-t --type
+Type of data to fetch. Options: orthologs, expression.
Optional arguments
+-o --out
+Path to the JSON file the results will be saved in, e.g. path/to/directory/results.json. Default: Standard out.
Flags
+-csv --csv
+Command-line only. Returns the output in CSV format, instead of JSON format.
+Python: Use json=True to return output in JSON format.
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
Get orthologs for a gene
+gget bgee ENSSSCG00000014725 -t orthologs
+import gget
+gget.bgee("ENSSSCG00000014725", type="orthologs")
+→ Returns orthologs for the gene with Ensembl ID ENSSSCG00000014725.
+| gene_id | gene_name | species_id | genus | species |
|---|---|---|---|---|
| 734881 | hbb1 | 8355 | Xenopus | laevis |
| ENSFCAG00000038029 | LOC101098159 | 9685 | Felis | catus |
| ENSBTAG00000047356 | LOC107131172 | 9913 | Bos | taurus |
| ENSOARG00000019163 | LOC101105437 | 9940 | Ovis | aries |
| ENSXETG00000025667 | hbg1 | 8364 | Xenopus | tropicalis |
| ... | ... | ... | ... | ... |
Get gene expression data for a gene
+gget bgee ENSSSCG00000014725 -t expression
+import gget
+gget.bgee("ENSSSCG00000014725", type="expression")
+→ Returns gene expression data for the gene with Ensembl ID ENSSSCG00000014725.
+| anat_entity_id | anat_entity_name | score | score_confidence | expression_state |
|---|---|---|---|---|
| UBERON:0000178 | blood | 99.98 | high | expressed |
| UBERON:0002106 | spleen | 99.96 | high | expressed |
| UBERON:0002190 | subcutaneous adipose tissue | 99.70 | high | expressed |
| UBERON:0005316 | endocardial endothelium | 99.61 | high | expressed |
| UBERON:0002107 | liver | 99.27 | high | expressed |
| ... | ... | ... | ... | ... |
If you use gget bgee in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Frederic B Bastian, Julien Roux, Anne Niknejad, Aurélie Comte, Sara S Fonseca Costa, Tarcisio Mendes de Farias, Sébastien Moretti, Gilles Parmentier, Valentine Rech de Laval, Marta Rosikiewicz, Julien Wollbrett, Amina Echchiki, Angélique Escoriza, Walid H Gharib, Mar Gonzales-Porta, Yohan Jarosz, Balazs Laurenczy, Philippe Moret, Emilie Person, Patrick Roelli, Komal Sanjeev, Mathieu Seppey, Marc Robinson-Rechavi (2021). The Bgee suite: integrated curated expression atlas and comparative transcriptomics in animals. Nucleic Acids Research, Volume 49, Issue D1, 8 January 2021, Pages D831–D847, https://doi.org/10.1093/nar/gkaa793
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
BLAST a nucleotide or amino acid sequence to any BLAST database.
+Return format: JSON (command-line) or data frame/CSV (Python).
Positional argument
+sequence
+Nucleotide or amino acid sequence, or path to FASTA or .txt file.
Optional arguments
+-p --program
+'blastn', 'blastp', 'blastx', 'tblastn', or 'tblastx'.
+Default: 'blastn' for nucleotide sequences; 'blastp' for amino acid sequences.
-db --database
+'nt', 'nr', 'refseq_rna', 'refseq_protein', 'swissprot', 'pdbaa', or 'pdbnt'.
+Default: 'nt' for nucleotide sequences; 'nr' for amino acid sequences.
+More info on BLAST databases
-l --limit
+Limits number of hits to return. Default: 50.
-e --expect
+Defines the expect value cutoff. Default: 10.0.
-o --out
+Path to the file the results will be saved in, e.g. path/to/directory/results.csv (or .json). Default: Standard out.
+Python: save=True will save the output in the current working directory.
Flags
+-lcf --low_comp_filt
+Turns on low complexity filter.
-mbo --megablast_off
+Turns off MegaBLAST algorithm. Default: MegaBLAST on (blastn only).
-csv --csv
+Command-line only. Returns results in CSV format.
+Python: Use json=True to return output in JSON format.
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
wrap_text
+Python only. wrap_text=True displays data frame with wrapped text for easy reading (default: False).
gget blast MKWMFKEDHSLEHRCVESAKIRAKYPDRVPVIVEKVSGSQIVDIDKRKYLVPSDITVAQFMWIIRKRIQLPSEKAIFLFVDKTVPQSR
+# Python
+gget.blast("MKWMFKEDHSLEHRCVESAKIRAKYPDRVPVIVEKVSGSQIVDIDKRKYLVPSDITVAQFMWIIRKRIQLPSEKAIFLFVDKTVPQSR")
+→ Returns the BLAST result of the sequence of interest. gget blast automatically detects this sequence as an amino acid sequence and therefore sets the BLAST program to blastp with database nr.
| Description | Scientific Name | Common Name | Taxid | Max Score | Total Score | Query Cover | ... |
|---|---|---|---|---|---|---|---|
| PREDICTED: gamma-aminobutyric acid receptor-as... | Colobus angolensis palliatus | NaN | 336983 | 180 | 180 | 100% | ... |
| . . . | . . . | . . . | . . . | . . . | . . . | . . . | ... |
+BLAST from .fa or .txt file:
gget blast fasta.fa
+# Python
+gget.blast("fasta.fa")
+→ Returns the BLAST results of the first sequence contained in the fasta.fa file.
+If you use gget blast in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ. Basic local alignment search tool. J Mol Biol. 1990 Oct 5;215(3):403-10. doi: 10.1016/S0022-2836(05)80360-2. PMID: 2231712.
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Find the genomic location of a nucleotide or amino acid sequence using BLAT.
+Return format: JSON (command-line) or data frame/CSV (Python).
Positional argument
+sequence
+Nucleotide or amino acid sequence, or path to FASTA or .txt file.
Optional arguments
+-st --seqtype
+'DNA', 'protein', 'translated%20RNA', or 'translated%20DNA'.
+Default: 'DNA' for nucleotide sequences; 'protein' for amino acid sequences.
-a --assembly
+'human' (hg38) (default), 'mouse' (mm39), 'zebrafinch' (taeGut2),
+or any of the species assemblies available here (use short assembly name).
-o --out
+Path to the file the results will be saved in, e.g. path/to/directory/results.csv (or .json). Default: Standard out.
+Python: save=True will save the output in the current working directory.
Flags
+-csv --csv
+Command-line only. Returns results in CSV format.
+Python: Use json=True to return output in JSON format.
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
gget blat -a taeGut2 MKWMFKEDHSLEHRCVESAKIRAKYPDRVPVIVEKVSGSQIVDIDKRKYLVPSDITVAQFMWIIRKRIQLPSEKAIFLFVDKTVPQSR
+# Python
+gget.blat("MKWMFKEDHSLEHRCVESAKIRAKYPDRVPVIVEKVSGSQIVDIDKRKYLVPSDITVAQFMWIIRKRIQLPSEKAIFLFVDKTVPQSR", assembly="taeGut2")
+→ Returns BLAT results for assembly taeGut2 (zebra finch). In the above example, gget blat automatically detects this sequence as an amino acid sequence and therefore sets the BLAT seqtype to protein.
| genome | query_size | aligned_start | aligned_end | matches | mismatches | %_aligned | ... |
|---|---|---|---|---|---|---|---|
| taeGut2 | 88 | 12 | 88 | 77 | 0 | 87.5 | ... |
If you use gget blat in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Kent WJ. BLAT--the BLAST-like alignment tool. Genome Res. 2002 Apr;12(4):656-64. doi: 10.1101/gr.229202. PMID: 11932250; PMCID: PMC187518.
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Plot cancer genomics heatmaps using data from cBioPortal using Ensembl IDs or gene names.
+This module was written by Sam Wagenaar.
+Positional argument
+subcommand
+Either search or plot
search subcommand (Python: gget.cbio_search)Find cBioPortal study IDs by keyword.
+Return format: JSON (command-line) or string list (Python).
+Note: This does not return studies with mixed cancer types.
Positional argument
+keywords
+Space-separated list of keywords to search for, e.g. breast lung.
+Python: Pass keywords as a list of strings.
plot subcommand (Python: gget.cbio_plot)Plot cancer genomics heatmaps using data from cBioPortal. +Return format: PNG (command-line and Python)
+Required arguments
+-s --study_ids
+Space-separated list of cBioPortal study IDs, e.g. msk_impact_2017 egc_msk_2023.
-g --genes
+Space-separated list of gene names or Ensembl IDs, e.g. NOTCH3 ENSG00000108375.
Optional arguments
+-st --stratification
+Column to stratify the data by. Default: tissue.
+Options:
-vt --variation_type
+Type of variation to plot. Default: mutation_occurrences.
+Options:
stratification must be 'sample' for this option)stratification must be 'sample' for this option)-f --filter
+Filter the data by a specific value in a specific column, e.g. study_id:msk_impact_2017
+Python: filter=(column, value)
-dd --data_dir
+Directory to store data files. Default: ./gget_cbio_cache.
-fd --figure_dir
+Directory to output figures. Default: ./gget_cbio_figures.
-fn --filename
+Filename for the output figure, relative to figure_dir. Default: auto-generated
+Python: figure_filename
-t --title
+Title for the output figure. Default: auto-generated
+Python: figure_title
-dpi --dpi
+DPI of the output figure. Default: 100.
Flags
+-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
-nc --no_confirm
+Command-line only. Skip download confirmation prompts.
+Python: Use confirm_download=True to enable download confirmation prompts.
-sh --show
+Show the plot in a window (automatic in Jupyter notebooks).
Find all cBioPortal studies with cancer types matching specific keywords:
+gget cbio search esophag ovary ovarian
+# Python
+import gget
+gget.cbio_search(['esophag', 'ovary', 'ovarian'])
+→ Returns a list of studies with cancer types matching the keywords esophag, ovary, or ovarian.
['egc_tmucih_2015', 'egc_msk_2017', ..., 'msk_spectrum_tme_2022']
+Plot a heatmap of mutation occurrences for specific genes in a specific study:
+gget cbio plot \
+ -s msk_impact_2017 \
+ -g AKT1 ALK FLT4 MAP3K1 MLL2 MLL3 NOTCH3 NOTCH4 PDCD1 RNF43 \
+ -st tissue \
+ -vt mutation_occurrences \
+ -dpi 200 \
+ -y
+# Python
+import gget
+gget.cbio_plot(
+ ['msk_impact_2017'],
+ ['AKT1', 'ALK', 'FLT4', 'MAP3K1', 'MLL2', 'MLL3', 'NOTCH3', 'NOTCH4', 'PDCD1', 'RNF43'],
+ stratification='tissue',
+ variation_type='mutation_occurrences',
+ dpi=200
+)
+→ Saves a heatmap of mutation occurrences for the specified genes in the specified study to ./gget_cbio_figures/Heatmap_tissue.png.

Plot a heatmap of mutation types for specific genes in a specific study:
+gget cbio plot \
+ -s msk_impact_2017 \
+ -g AKT1 ALK FLT4 MAP3K1 MLL2 MLL3 NOTCH3 NOTCH4 PDCD1 RNF43 \
+ -st sample \
+ -vt Consequence \
+ -dpi 200 \
+ -y
+# Python
+import gget
+gget.cbio_plot(
+ ['msk_impact_2017'],
+ ['AKT1', 'ALK', 'FLT4', 'MAP3K1', 'MLL2', 'MLL3', 'NOTCH3', 'NOTCH4', 'PDCD1', 'RNF43'],
+ stratification='sample',
+ variation_type='Consequence',
+ dpi=200,
+)
+→ Saves a heatmap of mutation types for the specified genes in the specified study to ./gget_cbio_figures/Heatmap_sample.png.

Plot a heatmap of mutation types for specific genes in a specific study, filtering by tissue:
+gget cbio plot \
+ -s msk_impact_2017 \
+ -g AKT1 ALK FLT4 MAP3K1 MLL2 MLL3 NOTCH3 NOTCH4 PDCD1 RNF43 \
+ -st sample \
+ -vt Consequence \
+ -f tissue:intestine \
+ -dpi 200 \
+ -y
+# Python
+import gget
+gget.cbio_plot(
+ ['msk_impact_2017'],
+ ['AKT1', 'ALK', 'FLT4', 'MAP3K1', 'MLL2', 'MLL3', 'NOTCH3', 'NOTCH4', 'PDCD1', 'RNF43'],
+ stratification='sample',
+ variation_type='Consequence',
+ filter=('tissue', 'intestine'),
+ dpi=200,
+)
+→ Saves a heatmap of mutation types for the specified genes in the specified study, filtered by tissue, to ./gget_cbio_figures/Heatmap_sample_intestine.png.

Plot a heatmap with a custom title and filename:
+gget cbio plot \
+ -s msk_impact_2017 \
+ -g AKT1 ALK FLT4 MAP3K1 MLL2 MLL3 NOTCH3 NOTCH4 PDCD1 RNF43 \
+ -st sample \
+ -vt Consequence \
+ -f tissue:intestine \
+ -dpi 200 \
+ -y \
+ -t "Intestinal Mutations" \
+ -fn intestinal_mutations.png
+# Python
+import gget
+gget.cbio_plot(
+ ['msk_impact_2017'],
+ ['AKT1', 'ALK', 'FLT4', 'MAP3K1', 'MLL2', 'MLL3', 'NOTCH3', 'NOTCH4', 'PDCD1', 'RNF43'],
+ stratification='sample',
+ variation_type='Consequence',
+ filter=('tissue', 'intestine'),
+ dpi=200,
+ figure_title='Intestinal Mutations',
+ figure_filename='intestinal_mutations.png'
+)
+→ Saves a heatmap of mutation types for the specified genes in the specified study, filtered by tissue, with the title "Intestinal Mutations" to ./gget_cbio_figures/intestinal_mutations.png.

If you use gget cbio in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Cerami E, Gao J, Dogrusoz U, Gross BE, Sumer SO, Aksoy BA, Jacobsen A, Byrne CJ, Heuer ML, Larsson E, Antipin Y, Reva B, Goldberg AP, Sander C, Schultz N. The cBio cancer genomics portal: an open platform for exploring multidimensional cancer genomics data. Cancer Discov. 2012 May;2(5):401-4. doi: 10.1158/2159-8290.CD-12-0095. Erratum in: Cancer Discov. 2012 Oct;2(10):960. PMID: 22588877; PMCID: PMC3956037.
+Gao J, Aksoy BA, Dogrusoz U, Dresdner G, Gross B, Sumer SO, Sun Y, Jacobsen A, Sinha R, Larsson E, Cerami E, Sander C, Schultz N. Integrative analysis of complex cancer genomics and clinical profiles using the cBioPortal. Sci Signal. 2013 Apr 2;6(269):pl1. doi: 10.1126/scisignal.2004088. PMID: 23550210; PMCID: PMC4160307.
+de Bruijn I, Kundra R, Mastrogiacomo B, Tran TN, Sikina L, Mazor T, Li X, Ochoa A, Zhao G, Lai B, Abeshouse A, Baiceanu D, Ciftci E, Dogrusoz U, Dufilie A, Erkoc Z, Garcia Lara E, Fu Z, Gross B, Haynes C, Heath A, Higgins D, Jagannathan P, Kalletla K, Kumari P, Lindsay J, Lisman A, Leenknegt B, Lukasse P, Madela D, Madupuri R, van Nierop P, Plantalech O, Quach J, Resnick AC, Rodenburg SYA, Satravada BA, Schaeffer F, Sheridan R, Singh J, Sirohi R, Sumer SO, van Hagen S, Wang A, Wilson M, Zhang H, Zhu K, Rusk N, Brown S, Lavery JA, Panageas KS, Rudolph JE, LeNoue-Newton ML, Warner JL, Guo X, Hunter-Zinck H, Yu TV, Pilai S, Nichols C, Gardos SM, Philip J; AACR Project GENIE BPC Core Team, AACR Project GENIE Consortium; Kehl KL, Riely GJ, Schrag D, Lee J, Fiandalo MV, Sweeney SM, Pugh TJ, Sander C, Cerami E, Gao J, Schultz N. Analysis and Visualization of Longitudinal Genomic and Clinical Data from the AACR Project GENIE Biopharma Collaborative in cBioPortal. Cancer Res. 2023 Dec 1;83(23):3861-3867. doi: 10.1158/0008-5472.CAN-23-0816. PMID: 37668528; PMCID: PMC10690089.
+Please also cite the source of the data if you are using a publicly available dataset.
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Query data from CZ CELLxGENE Discover using the CZ CELLxGENE Discover Census. CZ CELLxGENE Discover provides ready-to-use single-cell RNA sequencing count matrices for certain tissues/diseases/genes/etc.
+Returns: An AnnData object containing the count matrix and metadata of single-cell RNA sequencing data from the defined tissues/genes/etc.
+Before using gget cellxgene for the first time, run gget setup cellxgene / gget.setup("cellxgene") once (also see gget setup).
Optional arguments
+-s --species
+Choice of 'homo_sapiens' or 'mus_musculus'. Default: 'homo_sapiens'.
-g --gene
+Str or list of gene name(s) or Ensembl ID(s). Default: None.
+NOTE: Use -e / --ensembl (Python: ensembl=True) when providing Ensembl ID(s) instead of gene name(s).
+See https://cellxgene.cziscience.com/gene-expression for examples of available genes.
-cv --census_version
+Str defining version of Census, e.g. "2023-05-15", or "latest" or "stable". Default: "stable".
-cn --column_names
+List of metadata columns to return (stored in AnnData.obs).
+Default: ['dataset_id', 'assay', 'suspension_type', 'sex', 'tissue_general', 'tissue', 'cell_type']
+For more options, see: https://api.cellxgene.cziscience.com/curation/ui/#/ -> Schemas -> dataset
-o --out
+Path to file to save generated AnnData .h5ad file (or .csv with -mo / --meta_only).
+Required when using from command line!
Flags
+-e --ensembl
+Use when genes are provided as Ensembl IDs instead of gene names.
-mo --meta_only
+Only returns metadata data frame (corresponds to AnnData.obs).
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
Optional arguments corresponding to CZ CELLxGENE Discover metadata attributes
+--tissue
+Str or list of tissue(s), e.g. ['lung', 'blood']. Default: None.
+See https://cellxgene.cziscience.com/gene-expression for examples of available tissues.
--cell_type
+Str or list of cell type (s), e.g. ['mucus secreting cell', 'neuroendocrine cell']. Default: None.
+See https://cellxgene.cziscience.com/gene-expression and select a tissue to see examples of available cell types.
--development_stage
+Str or list of development stage(s). Default: None.
--disease
+Str or list of disease(s). Default: None.
--sex
+Str or list of sex(es), e.g. 'female'. Default: None.
--dataset_id
+Str or list of CELLxGENE dataset ID(s). Default: None.
--tissue_general_ontology_term_id
+Str or list of high-level tissue UBERON ID(s). Default: None.
+Tissue labels and their corresponding UBERON IDs are listed here.
--tissue_general
+Str or list of high-level tissue label(s). Default: None.
+Tissue labels and their corresponding UBERON IDs are listed here.
--tissue_ontology_term_id
+Str or list of tissue ontology term ID(s) as defined in the CELLxGENE dataset schema. Default: None.
--assay_ontology_term_id
+Str or list of assay ontology term ID(s) as defined in the CELLxGENE dataset schema. Default: None.
--assay
+Str or list of assay(s) as defined in the CELLxGENE dataset schema. Default: None.
--cell_type_ontology_term_id
+Str or list of cell type ontology term ID(s) as defined in the CELLxGENE dataset schema. Default: None.
--development_stage_ontology_term_id
+Str or list of development stage ontology term ID(s) as defined in the CELLxGENE dataset schema. Default: None.
--disease_ontology_term_id
+Str or list of disease ontology term ID(s) as defined in the CELLxGENE dataset schema. Default: None.
--donor_id
+Str or list of donor ID(s) as defined in the CELLxGENE dataset schema. Default: None.
--self_reported_ethnicity_ontology_term_id
+Str or list of self-reported ethnicity ontology ID(s) as defined in the CELLxGENE dataset schema. Default: None.
--self_reported_ethnicity
+Str or list of self-reported ethnicity as defined in the CELLxGENE dataset schema. Default: None.
--sex_ontology_term_id
+Str or list of sex ontology ID(s) as defined in the CELLxGENE dataset schema. Default: None.
--suspension_type
+Str or list of suspension type(s) as defined in the CELLxGENE dataset schema. Default: None.
gget cellxgene --gene ACE2 ABCA1 SLC5A1 --tissue lung --cell_type 'mucus secreting cell' 'neuroendocrine cell' -o example_adata.h5ad
+# Python
+adata = gget.cellxgene(
+ gene = ["ACE2", "ABCA1", "SLC5A1"],
+ tissue = "lung",
+ cell_type = ["mucus secreting cell", "neuroendocrine cell"]
+)
+adata
+→ Returns an AnnData object containing the scRNAseq ACE2, ABCA1, and SLC5A1 count matrix of 3322 human lung mucus secreting and neuroendocrine cells from CZ CELLxGENE Discover and their corresponding metadata.
+Fetch metadata (corresponds to AnnData.obs) only:
+gget cellxgene --meta_only --gene ENSMUSG00000015405 --ensembl --tissue lung --species mus_musculus -o example_meta.csv
+# Python
+df = gget.cellxgene(
+ meta_only = True,
+ gene = "ENSMUSG00000015405",
+ ensembl = True,
+ tissue = "lung",
+ species = "mus_musculus"
+)
+df
+→ Returns only the metadata from ENSMUSG00000015405 (ACE2) expression datasets corresponding to mouse lung cells.
+Also see: https://chanzuckerberg.github.io/cellxgene-census/notebooks/api_demo/census_gget_demo.html
+If you use gget cellxgene in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Chanzuckerberg Initiative. (n.d.). CZ CELLxGENE Discover. Retrieved [insert date here], from https://cellxgene.cziscience.com/
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Search for genes, mutations, and other factors associated with cancer using the COSMIC (Catalogue Of Somatic Mutations In Cancer) database.
+Return format: JSON (command-line) or data frame/CSV (Python) when download_cosmic=False. When download_cosmic=True, downloads the requested database into the specified folder.
This module was written in part by @AubakirovArman (information querying) and @josephrich98 (database download).
+NOTE: License fees apply for the commercial use of COSMIC. You can read more about licensing COSMIC data here.
+Positional argument (for querying information)
+searchterm
+Search term, which can be a mutation, or gene name (or Ensembl ID), or sample, etc.
+Examples for the searchterm and entitity arguments:
| searchterm | entitity | |
|---|---|---|
| EGFR | mutations | -> Find mutations in the EGFR gene that are associated with cancer |
| v600e | mutations | -> Find genes for which a v600e mutation is associated with cancer |
| COSV57014428 | mutations | -> Find mutations associated with this COSMIC mutations ID |
| EGFR | genes | -> Get the number of samples, coding/simple mutations, and fusions observed in COSMIC for EGFR |
| prostate | cancer | -> Get number of tested samples and mutations for prostate cancer |
| prostate | tumour_site | -> Get number of tested samples, genes, mutations, fusions, etc. with 'prostate' as primary tissue site |
| ICGC | studies | -> Get project code and descriptions for all studies from the ICGC (International Cancer Genome Consortium) |
| EGFR | pubmed | -> Find PubMed publications on EGFR and cancer |
| ICGC | samples | -> Get metadata on all samples from the ICGC (International Cancer Genome Consortium) |
| COSS2907494 | samples | -> Get metadata on this COSMIC sample ID (cancer type, tissue, # analyzed genes, # mutations, etc.) |
NOTE: (Python only) Set to None when downloading COSMIC databases with download_cosmic=True.
Optional arguments (for querying information)
+-e --entity
+'mutations' (default), 'genes', 'cancer', 'tumour site', 'studies', 'pubmed', or 'samples'.
+Defines the type of the results to return.
-l --limit
+Limits number of hits to return. Default: 100.
Flags (for downloading COSMIC databases)
+-d --download_cosmic
+Switches into database download mode.
-gm --gget_mutate
+TURNS OFF creation of a modified version of the database for use with gget mutate.
+Python: gget_mutate is True by default. Set gget_mutate=False to disable.
Optional arguments (for downloading COSMIC databases)
+-mc --mutation_class
+'cancer' (default), 'cell_line', 'census', 'resistance', 'genome_screen', 'targeted_screen', or 'cancer_example'
+Type of COSMIC database to download.
-cv --cosmic_version
+Version of the COSMIC database. Default: None -> Defaults to latest version.
-gv --grch_version
+Version of the human GRCh reference genome the COSMIC database was based on (37 or 38). Default: 37
--keep_genome_info
+Whether to keep genome information in the modified database for use with gget mutate. Default: False
--remove_duplicates
+Whether to remove duplicate rows from the modified database for use with gget mutate. Default: False
Optional arguments (general)
+-o --out
+Path to the file (or folder when downloading databases with the download_cosmic flag) the results will be saved in, e.g. 'path/to/results.json'.
+Default: None
+-> When download_cosmic=False: Results will be returned to standard out
+-> When download_cosmic=True: Database will be downloaded into current working directory
Flags (general)
+-csv --csv
+Command-line only. Returns results in CSV format.
+Python: Use json=True to return output in JSON format.
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
gget cosmic EGFR
+# Python
+gget.cosmic("EGFR")
+→ Returns mutations in the EGFR gene that are associated with cancer in the format:
+| Gene | Syntax | Alternate IDs | Canonical |
|---|---|---|---|
| EGFR | c.*2446A>G | EGFR c.*2446A>G, EGFR p.?, ... | y |
| EGFR | c.(2185_2283)ins(18) | EGFR c.(2185_2283)ins(18), EGFR p.?, ... | y |
| . . . | . . . | . . . | . . . |
gget cosmic --download_cosmic
+# Python
+gget.cosmic(searchterm=None, download_cosmic=True)
+→ Downloads the COSMIC cancer database of the latest COSMIC release into the current working directory.
+If you use gget cosmic in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Tate JG, Bamford S, Jubb HC, Sondka Z, Beare DM, Bindal N, Boutselakis H, Cole CG, Creatore C, Dawson E, Fish P, Harsha B, Hathaway C, Jupe SC, Kok CY, Noble K, Ponting L, Ramshaw CC, Rye CE, Speedy HE, Stefancsik R, Thompson SL, Wang S, Ward S, Campbell PJ, Forbes SA. COSMIC: the Catalogue Of Somatic Mutations In Cancer. Nucleic Acids Res. 2019 Jan 8;47(D1):D941-D947. doi: 10.1093/nar/gky1015. PMID: 30371878; PMCID: PMC6323903.
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Align multiple protein or translated DNA sequences using DIAMOND (DIAMOND is similar to BLAST, but this is a local computation).
+Return format: JSON (command-line) or data frame/CSV (Python).
Positional argument
+query
+Sequences (str or list) or path to FASTA file containing sequences to be aligned against the reference.
Required arguments
+-ref --reference
+Reference sequences (str or list) or path to FASTA file containing reference sequences.
Optional arguments
+-db --diamond_db
+Path to save DIAMOND database created from reference (str).
+Default: None -> Temporary db file will be deleted after alignment or saved in out if out is provided.
-s --sensitivity
+Sensitivity of alignment (str). Default: "very-sensitive".
+One of the following: fast, mid-sensitive, sensitive, more-sensitive, very-sensitive, or ultra-sensitive.
-t --threads
+Number of threads used (int). Default: 1.
-db --diamond_binary
+Path to DIAMOND binary (str). Default: None -> Uses DIAMOND binary installed with gget.
-o --out
+Path to the folder to save results in (str), e.g. "path/to/directory". Default: Standard out; temporary files are deleted.
Flags
+-csv --csv
+Command-line only. Returns results in CSV format.
+Python: Use json=True to return output in JSON format.
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
# !!! Make sure to list the positional argument first here so it is not added as a reference sequence
+gget diamond GGETISAWESQME ELVISISALIVE LQVEFRANKLIN PACHTERLABRQCKS -ref GGETISAWESQMEELVISISALIVELQVEFRANKLIN PACHTERLABRQCKS
+# Python
+gget.diamond(["GGETISAWESQME", "ELVISISALIVE", "LQVEFRANKLIN", "PACHTERLABRQCKS"], reference=["GGETISAWESQMEELVISISALIVELQVEFRANKLIN", "PACHTERLABRQCKS"])
+→ Returns results in JSON (command-line) or data frame/CSV (Python) format:
+| query_accession | subject_accession | identity_percentage | query_seq_length | subject_seq_length | length | mismatches | gap_openings | query_start | query_end | subject_start | subject_end | e-value | bit_score |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Seq0 | Seq0 | 100 | 13 | 37 | 13 | 0 | 0 | 1 | 13 | 1 | 13 | 2.82e-09 | 30.8 |
| Seq2 | Seq0 | 100 | 12 | 37 | 12 | 0 | 0 | 1 | 12 | 26 | 37 | 4.35e-08 | 27.7 |
| Seq3 | Seq1 | 100 | 15 | 15 | 15 | 0 | 0 | 1 | 15 | 1 | 15 | 2.01e-11 | 36.2 |
If you use gget diamond in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Buchfink, B., Xie, C. & Huson, D. Fast and sensitive protein alignment using DIAMOND. Nat Methods 12, 59–60 (2015). https://doi.org/10.1038/nmeth.3176
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Locally predict Eukaryotic Linear Motifs from an amino acid sequence or UniProt Acc using data from the ELM database.
+Return format: JSON (command-line) or data frame/CSV (Python). This module returns two data frames (or JSON formatted files) (see examples).
ELM data can be downloaded & distributed for non-commercial use according to the ELM Software License Agreement.
+Before using gget elm for the first time, run gget setup elm (bash) / gget.setup("elm") (Python) once (also see gget setup).
Positional argument
+sequence
+Amino acid sequence or Uniprot Acc (str).
+When providing a Uniprot Acc, use flag --uniprot (Python: uniprot=True).
Optional arguments
+-s --sensitivity
+Sensitivity of DIAMOND alignment (str). Default: "very-sensitive".
+One of the following: fast, mid-sensitive, sensitive, more-sensitive, very-sensitive, or ultra-sensitive.
-t --threads
+Number of threads used in DIAMOND alignment (int). Default: 1.
-bin --diamond_binary
+Path to DIAMOND binary (str). Default: None -> Uses DIAMOND binary installed with gget.
-o --out
+Path to the folder to save results in (str), e.g. "path/to/directory". Default: Standard out; temporary files are deleted.
Flags
+-u --uniprot
+Set to True if sequence is a Uniprot Acc instead of an amino acid sequence.
-e --expand
+Expand the information returned in the regex data frame to include the protein names, organisms, and references that the motif was orignally validated on.
-csv --csv
+Command-line only. Returns results in CSV format.
+Python: Use json=True to return output in JSON format.
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
Find ELMs in an amino acid sequence:
+gget setup elm # Downloads/updates local ELM database
+gget elm -o gget_elm_results LIAQSIGQASFV
+# Python
+gget.setup(“elm”) # Downloads/updates local ELM database
+ortholog_df, regex_df = gget.elm("LIAQSIGQASFV")
+Find ELMs giving a UniProt Acc as input:
+gget setup elm # Downloads/updates local ELM database
+gget elm -o gget_elm_results --uniprot Q02410 -e
+# Python
+gget.setup(“elm”) # Downloads/updates local ELM database
+ortholog_df, regex_df = gget.elm("Q02410", uniprot=True, expand=True)
+→ Returns two data frames (or JSON formatted dictionaries for command line) containing extensive information about linear motifs associated with orthologous proteins and motifs found in the input sequence directly based on their regex expressions:
+ortholog_df:
+| Ortholog_UniProt_Acc | ProteinName | class_accession | ELMIdentifier | FunctionalSiteName | Description | Organism | … |
|---|---|---|---|---|---|---|---|
| Q02410 | APBA1_HUMAN | ELME000357 | LIG_CaMK_CASK_1 | CASK CaMK domain binding ligand motif | Motif that mediates binding to the calmodulin-dependent protein kinase (CaMK) domain of the peripheral plasma membrane protein CASK/Lin2. | Homo sapiens | … |
| Q02410 | APBA1_HUMAN | ELME000091 | LIG_PDZ_Class_2 | PDZ domain ligands | The C-terminal class 2 PDZ-binding motif is classically represented by a pattern such as | Homo sapiens | … |
regex_df:
+| Instance_accession | ELMIdentifier | FunctionalSiteName | ELMType | Description | Instances (Matched Sequence) | Organism | … |
|---|---|---|---|---|---|---|---|
| ELME000321 | CLV_C14_Caspase3-7 | Caspase cleavage motif | CLV | Caspase-3 and Caspase-7 cleavage site. | ERSDG | Mus musculus | … |
| ELME000102 | CLV_NRD_NRD_1 | NRD cleavage site | CLV | N-Arg dibasic convertase (NRD/Nardilysin) cleavage site. | RRA | Rattus norvegicus | … |
| ELME000100 | CLV_PCSK_PC1ET2_1 | PCSK cleavage site | CLV | NEC1/NEC2 cleavage site. | KRD | Mus musculus | … |
| ELME000146 | CLV_PCSK_SKI1_1 | PCSK cleavage site | CLV | Subtilisin/kexin isozyme-1 (SKI1) cleavage site. | RLLTA | Homo sapiens | … |
| ELME000231 | DEG_APCC_DBOX_1 | APCC-binding Destruction motifs | DEG | An RxxL-based motif that binds to the Cdh1 and Cdc20 components of APC/C thereby targeting the protein for destruction in a cell cycle dependent manner | SRVKLNIVR | Saccharomyces cerevisiae S288c | … |
| … | … | … | … | … | … | … | … |
gget elm demogget elm results based on disordered protein regionsIf you use gget elm in a publication, please cite the following articles:
Laura Luebbert, Chi Hoang, Manjeet Kumar, Lior Pachter, Fast and scalable querying of eukaryotic linear motifs with gget elm, Bioinformatics, 2024, btae095, https://doi.org/10.1093/bioinformatics/btae095
+Manjeet Kumar, Sushama Michael, Jesús Alvarado-Valverde, Bálint Mészáros, Hugo Sámano‐Sánchez, András Zeke, Laszlo Dobson, Tamas Lazar, Mihkel Örd, Anurag Nagpal, Nazanin Farahi, Melanie Käser, Ramya Kraleti, Norman E Davey, Rita Pancsa, Lucía B Chemes, Toby J Gibson, The Eukaryotic Linear Motif resource: 2022 release, Nucleic Acids Research, Volume 50, Issue D1, 7 January 2022, Pages D497–D508, https://doi.org/10.1093/nar/gkab975
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Perform an enrichment analysis on a list of genes using Enrichr or modEnrichr.
+Return format: JSON (command-line) or data frame/CSV (Python).
Positional argument
+genes
+Short names (gene symbols) of genes to perform enrichment analysis on, e.g. PHF14 RBM3 MSL1 PHF21A.
+Alternatively: use flag --ensembl to input a list of Ensembl gene IDs, e.g. ENSG00000106443 ENSG00000102317 ENSG00000188895.
Other required arguments
+-db --database
+Database database to use as reference for the enrichment analysis.
+Supports any database listed here under 'Gene-set Library' or one of the following shortcuts:
+'pathway' (KEGG_2021_Human)
+'transcription' (ChEA_2016)
+'ontology' (GO_Biological_Process_2021)
+'diseases_drugs' (GWAS_Catalog_2019)
+'celltypes' (PanglaoDB_Augmented_2021)
+'kinase_interactions' (KEA_2015)
NOTE: database shortcuts are not supported for species other than 'human' or 'mouse'. Click on the species databases listed below under species to view a list of databases available for each species.
Optional arguments
+-s --species
+Species to use as reference for the enrichment analysis. (Default: human)
+Options:
| Species | Database list |
|---|---|
human | Enrichr |
mouse | Equivalent to human |
fly | FlyEnrichr |
yeast | YeastEnrichr |
worm | WormEnrichr |
fish | FishEnrichr |
-bkg_l --background_list
+Short names (gene symbols) of background genes to perform enrichment analysis on, e.g. NSUN3 POLRMT NLRX1.
+Alternatively: use flag --ensembl_background to input a list of Ensembl gene IDs.
+See this Tweetorial to learn why you should use a background gene list when performing an enrichment analysis.
-o --out
+Path to the file the results will be saved in, e.g. path/to/directory/results.csv (or .json). (Default: Standard out.)
+Python: save=True will save the output in the current working directory.
-ko --kegg_out
+Path to the png file the marked KEGG pathway images will be saved in, e.g. path/to/directory/pathway.png. (Default: None)
-kr --kegg_rank
+Rank of the KEGG pathway to be plotted. (Default: 1)
figsize
+Python only. (width, height) of plot in inches. (Default: (10,10))
ax
+Python only. Pass a matplotlib axes object for plot customization. (Default: None)
Flags
+-e --ensembl
+Add this flag if genes are given as Ensembl gene IDs.
-e_b --ensembl_background
+Add this flag if background_list are given as Ensembl gene IDs.
-bkg --background
+If True, use set of > 20,000 default background genes listed here.
-csv --csv
+Command-line only. Returns results in CSV format.
+Python: Use json=True to return output in JSON format.
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
plot
+Python only. plot=True provides a graphical overview of the first 15 results (default: False).
gget enrichr -db ontology ACE2 AGT AGTR1
+# Python
+gget.enrichr(["ACE2", "AGT", "AGTR1"], database="ontology", plot=True)
+→ Returns pathways/functions involving genes ACE2, AGT, and AGTR1 from the GO Biological Process 2021 database. In Python, plot=True returns a graphical overview of the results:

Use gget enrichr with a background gene list:
+See this Tweetorial to learn why you should use a background gene list when performing an enrichment analysis.
# Here, we are passing the input genes first (positional argument 'genes'), so they are not added to the background gene list behind the '-bkgr_l' argument
+gget enrichr \
+ PHF14 RBM3 MSL1 PHF21A ARL10 INSR JADE2 P2RX7 LINC00662 CCDC101 PPM1B KANSL1L CRYZL1 ANAPC16 TMCC1 CDH8 RBM11 CNPY2 HSPA1L CUL2 PLBD2 LARP7 TECPR2 ZNF302 CUX1 MOB2 CYTH2 SEC22C EIF4E3 ROBO2 ADAMTS9-AS2 CXXC1 LINC01314 ATF7 ATP5F1 \
+ -db ChEA_2022 \
+ -bkg_l NSUN3 POLRMT NLRX1 SFXN5 ZC3H12C SLC25A39 ARSG DEFB29 PCMTD2 ACAA1A LRRC1 2810432D09RIK SEPHS2 SAC3D1 TMLHE LOC623451 TSR2 PLEKHA7 GYS2 ARHGEF12 HIBCH LYRM2 ZBTB44 ENTPD5 RAB11FIP2 LIPT1 INTU ANXA13 KLF12 SAT2 GAL3ST2 VAMP8 FKBPL AQP11 TRAP1 PMPCB TM7SF3 RBM39 BRI3 KDR ZFP748 NAP1L1 DHRS1 LRRC56 WDR20A STXBP2 KLF1 UFC1 CCDC16 9230114K14RIK RWDD3 2610528K11RIK ACO1 CABLES1 LOC100047214 YARS2 LYPLA1 KALRN GYK ZFP787 ZFP655 RABEPK ZFP650 4732466D17RIK EXOSC4 WDR42A GPHN 2610528J11RIK 1110003E01RIK MDH1 1200014M14RIK AW209491 MUT 1700123L14RIK 2610036D13RIK PHF14 RBM3 MSL1 PHF21A ARL10 INSR JADE2 P2RX7 LINC00662 CCDC101 PPM1B KANSL1L CRYZL1 ANAPC16 TMCC1 CDH8 RBM11 CNPY2 HSPA1L CUL2 PLBD2 LARP7 TECPR2 ZNF302 CUX1 MOB2 CYTH2 SEC22C EIF4E3 ROBO2 ADAMTS9-AS2 CXXC1 LINC01314 ATF7 ATP5F1COX15 TMEM30A NSMCE4A TM2D2 RHBDD3 ATXN2 NFS1 3110001I20RIK BC038156 C330002I19RIK ZFYVE20 POLI TOMM70A LOC100047782 2410012H22RIK RILP A230062G08RIK PTTG1IP RAB1 AFAP1L1 LYRM5 2310026E23RIK SLC7A6OS MAT2B 4932438A13RIK LRRC8A SMO NUPL2
+# Python
+gget.enrichr(
+ genes = [
+ "PHF14", "RBM3", "MSL1", "PHF21A", "ARL10", "INSR", "JADE2", "P2RX7",
+ "LINC00662", "CCDC101", "PPM1B", "KANSL1L", "CRYZL1", "ANAPC16", "TMCC1",
+ "CDH8", "RBM11", "CNPY2", "HSPA1L", "CUL2", "PLBD2", "LARP7", "TECPR2",
+ "ZNF302", "CUX1", "MOB2", "CYTH2", "SEC22C", "EIF4E3", "ROBO2",
+ "ADAMTS9-AS2", "CXXC1", "LINC01314", "ATF7", "ATP5F1"
+ ],
+ database = "ChEA_2022",
+ background_list = [
+ "NSUN3","POLRMT","NLRX1","SFXN5","ZC3H12C","SLC25A39","ARSG",
+ "DEFB29","PCMTD2","ACAA1A","LRRC1","2810432D09RIK","SEPHS2",
+ "SAC3D1","TMLHE","LOC623451","TSR2","PLEKHA7","GYS2","ARHGEF12",
+ "HIBCH","LYRM2","ZBTB44","ENTPD5","RAB11FIP2","LIPT1",
+ "INTU","ANXA13","KLF12","SAT2","GAL3ST2","VAMP8","FKBPL",
+ "AQP11","TRAP1","PMPCB","TM7SF3","RBM39","BRI3","KDR","ZFP748",
+ "NAP1L1","DHRS1","LRRC56","WDR20A","STXBP2","KLF1","UFC1",
+ "CCDC16","9230114K14RIK","RWDD3","2610528K11RIK","ACO1",
+ "CABLES1", "LOC100047214","YARS2","LYPLA1","KALRN","GYK",
+ "ZFP787","ZFP655","RABEPK","ZFP650","4732466D17RIK","EXOSC4",
+ "WDR42A","GPHN","2610528J11RIK","1110003E01RIK","MDH1","1200014M14RIK",
+ "AW209491","MUT","1700123L14RIK","2610036D13RIK",
+ "PHF14", "RBM3", "MSL1", "PHF21A", "ARL10", "INSR", "JADE2",
+ "P2RX7", "LINC00662", "CCDC101", "PPM1B", "KANSL1L", "CRYZL1",
+ "ANAPC16", "TMCC1","CDH8", "RBM11", "CNPY2", "HSPA1L", "CUL2",
+ "PLBD2", "LARP7", "TECPR2", "ZNF302", "CUX1", "MOB2", "CYTH2",
+ "SEC22C", "EIF4E3", "ROBO2", "ADAMTS9-AS2", "CXXC1", "LINC01314", "ATF7",
+ "ATP5F1""COX15","TMEM30A","NSMCE4A","TM2D2","RHBDD3","ATXN2","NFS1",
+ "3110001I20RIK","BC038156","C330002I19RIK","ZFYVE20","POLI","TOMM70A",
+ "LOC100047782","2410012H22RIK","RILP","A230062G08RIK",
+ "PTTG1IP","RAB1","AFAP1L1", "LYRM5","2310026E23RIK",
+ "SLC7A6OS","MAT2B","4932438A13RIK","LRRC8A","SMO","NUPL2"
+ ],
+ plot=True
+)
+→ Returns hits of the input gene list given the background gene list from the transcription factor/target library ChEA 2022. In Python, plot=True returns a graphical overview of the results:

Generate a KEGG pathway image with the genes from the enrichment analysis highlighted:
+This feature is available thanks to a PR by Noriaki Sato.
gget enrichr -db pathway --kegg_out kegg.png --kegg_rank 1 ZBP1 IRF3 RIPK1
+# Python
+gget.enrichr(["ZBP1", "IRF3", "RIPK1"], database="pathway", kegg_out="kegg.png", kegg_rank=1)
+→ In addition to the standard gget enrichr output, the kegg_out argument saves an image with the genes from the enrichment analysis highlighted in the KEGG pathway:
The following example was submitted by Dylan Lawless via PR:
+Use gget enrichr in R and create a similar plot using ggplot.
+NOTE the switch of axes compared to the Python plot.
system("pip install gget")
+install.packages("reticulate")
+library(reticulate)
+gget <- import("gget")
+
+# Perform enrichment analysis on a list of genes
+df <- gget$enrichr(list("ACE2", "AGT", "AGTR1"), database = "ontology")
+
+# Count number of overlapping genes
+df$overlapping_genes_count <- lapply(df$overlapping_genes, length) |> as.numeric()
+
+# Only keep the top 15 results
+df <- df[1:15, ]
+
+# Plot
+library(ggplot2)
+
+df |>
+ ggplot() +
+ geom_bar(aes(
+ x = -log10(adj_p_val),
+ y = reorder(path_name, -adj_p_val)
+ ),
+ stat = "identity",
+ fill = "lightgrey",
+ width = 0.5,
+ color = "black") +
+ geom_text(
+ aes(
+ y = path_name,
+ x = (-log10(adj_p_val)),
+ label = overlapping_genes_count
+ ),
+ nudge_x = 0.75,
+ show.legend = NA,
+ color = "red"
+ ) +
+ geom_text(
+ aes(
+ y = Inf,
+ x = Inf,
+ hjust = 1,
+ vjust = 1,
+ label = "# of overlapping genes"
+ ),
+ show.legend = NA,
+ color = "red"
+ ) +
+ geom_vline(linetype = "dotted", linewidth = 1, xintercept = -log10(0.05)) +
+ ylab("Pathway name") +
+ xlab("-log10(adjusted P value)")
+Using gget enrichr with background genes
If you use gget enrichr in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Chen EY, Tan CM, Kou Y, Duan Q, Wang Z, Meirelles GV, Clark NR, Ma'ayan A. Enrichr: interactive and collaborative HTML5 gene list enrichment analysis tool. BMC Bioinformatics. 2013; 128(14). https://doi.org/10.1186/1471-2105-14-128
+Kuleshov MV, Jones MR, Rouillard AD, Fernandez NF, Duan Q, Wang Z, Koplev S, Jenkins SL, Jagodnik KM, Lachmann A, McDermott MG, Monteiro CD, Gundersen GW, Ma'ayan A. Enrichr: a comprehensive gene set enrichment analysis web server 2016 update. Nucleic Acids Research. 2016; gkw377. doi: 10.1093/nar/gkw377
+Xie Z, Bailey A, Kuleshov MV, Clarke DJB., Evangelista JE, Jenkins SL, Lachmann A, Wojciechowicz ML, Kropiwnicki E, Jagodnik KM, Jeon M, & Ma’ayan A. Gene set knowledge discovery with Enrichr. Current Protocols, 1, e90. 2021. doi: 10.1002/cpz1.90.
+If working with non-human/mouse datasets, please also cite:
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Generates natural language text based on a given prompt using the OpenAI API's 'openai.ChatCompletion.create' endpoint. +This module, including its source code, documentation and unit tests, were partly written by OpenAI's Chat-GTP3.
+NOTE:
+OpenAI API calls are only 'free' for the first three months after generating your OpenAI Account (OpenAI provides a $5 credit that expires).
+You can define a hard monthly billing limit (e.g. $1) here.
+See their pricing and FAQ here.
+Get your OpenAI API key here.
Returns: A string containing the generated text.
+Before using gget gpt for the first time, run gget setup gpt / gget.setup("gpt") once (also see gget setup).
Positional argument
+prompt
+The input prompt to generate text from (str).
api_key
+Your OpenAI API key (str) (get your API key).
Optional arguments
+-m --model
+The name of the GPT model to use for generating the text (str). Default is "gpt-3.5-turbo".
+See https://platform.openai.com/docs/models/gpt-4 for more information on the available models.
-temp --temperature
+Value between 0 and 2 that controls the level of randomness and creativity in the generated text (float).
+Higher values result in more creative and varied text. Default is 1.
-tp --top_p
+Controls the diversity of the generated text as an alternative to sampling with temperature (float).
+Higher values result in more diverse and unexpected text. Default is 1.
+Note: OpenAI recommends altering this or temperature but not both.
-s --stop
+A sequence of tokens to mark the end of the generated text (str). Default is None.
-mt --max_tokens
+Controls the maximum length of the generated text, in tokens (int). Default is 200.
-pp --presence_penalty
+Number between -2.0 and 2.0. Higher values result increase the model's likelihood to talk about new topics (float). Default is 0.
-fp --frequency_penalty
+Number between -2.0 and 2.0. Higher values decrease the model's likelihood to repeat the same line verbatim (float). Default is 0.
-lb --logit_bias
+A dictionary that specifies a bias towards certain tokens in the generated text (dict). Default is None.
-o --out
+If provided, saves the generated text to a file with the specified path (str). Default: Standard out.
gget gpt "How are you today GPT?" your_api_token
+# Python
+print(gget.gpt("How are you today GPT?", "your_api_token"))
+ +
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Fetch extensive gene and transcript metadata from Ensembl, UniProt, and NCBI using Ensembl IDs.
+Return format: JSON (command-line) or data frame/CSV (Python).
Positional argument
+ens_ids
+One or more Ensembl IDs (WormBase and Flybase IDs are also supported).
Optional arguments
+-o --out
+Path to the file the results will be saved in, e.g. path/to/directory/results.csv (or .json). Default: Standard out.
+Python: save=True will save the output in the current working directory.
Flags
+-n --ncbi
+TURN OFF results from NCBI.
+Python: ncbi=False prevents data retrieval from NCBI (default: True).
-u --uniprot
+TURN OFF results from UniProt.
+Python: uniprot=False prevents data retrieval from UniProt (default: True).
-pdb --pdb
+INCLUDE PDB IDs in output (might increase runtime).
+Python: pdb=True includes PDB IDs in the results (default: False).
-csv --csv
+Command-line only. Returns results in CSV format.
+Python: Use json=True to return output in JSON format.
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
wrap_text
+Python only. wrap_text=True displays data frame with wrapped text for easy reading (default: False).
gget info ENSG00000034713 ENSG00000104853 ENSG00000170296
+# Python
+gget.info(["ENSG00000034713", "ENSG00000104853", "ENSG00000170296"])
+→ Returns extensive information about each requested Ensembl ID:
+| uniprot_id | ncbi_gene_id | primary_gene_name | synonyms | protein_names | ensembl_description | uniprot_description | ncbi_description | biotype | canonical_transcript | ... | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ENSG00000034713 | P60520 | 11345 | GABARAPL2 | [ATG8, ATG8C, FLC3A, GABARAPL2, GATE-16, GATE16, GEF-2, GEF2] | Gamma-aminobutyric acid receptor-associated protein like 2 (GABA(A) receptor-associated protein-like 2)... | GABA type A receptor associated protein like 2 [Source:HGNC Symbol;Acc:HGNC:13291] | FUNCTION: Ubiquitin-like modifier involved in intra- Golgi traffic (By similarity). Modulates intra-Golgi transport through coupling between NSF activity and ... | Enables ubiquitin protein ligase binding activity. Involved in negative regulation of proteasomal protein catabolic process and protein... | protein_coding | ENST00000037243.7 | ... |
| . . . | . . . | . . . | . . . | . . . | . . . | . . . | . . . | . . . | . . . | . . . | ... |
If you use gget info in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Martin FJ, Amode MR, Aneja A, Austine-Orimoloye O, Azov AG, Barnes I, Becker A, Bennett R, Berry A, Bhai J, Bhurji SK, Bignell A, Boddu S, Branco Lins PR, Brooks L, Ramaraju SB, Charkhchi M, Cockburn A, Da Rin Fiorretto L, Davidson C, Dodiya K, Donaldson S, El Houdaigui B, El Naboulsi T, Fatima R, Giron CG, Genez T, Ghattaoraya GS, Martinez JG, Guijarro C, Hardy M, Hollis Z, Hourlier T, Hunt T, Kay M, Kaykala V, Le T, Lemos D, Marques-Coelho D, Marugán JC, Merino GA, Mirabueno LP, Mushtaq A, Hossain SN, Ogeh DN, Sakthivel MP, Parker A, Perry M, Piližota I, Prosovetskaia I, Pérez-Silva JG, Salam AIA, Saraiva-Agostinho N, Schuilenburg H, Sheppard D, Sinha S, Sipos B, Stark W, Steed E, Sukumaran R, Sumathipala D, Suner MM, Surapaneni L, Sutinen K, Szpak M, Tricomi FF, Urbina-Gómez D, Veidenberg A, Walsh TA, Walts B, Wass E, Willhoft N, Allen J, Alvarez-Jarreta J, Chakiachvili M, Flint B, Giorgetti S, Haggerty L, Ilsley GR, Loveland JE, Moore B, Mudge JM, Tate J, Thybert D, Trevanion SJ, Winterbottom A, Frankish A, Hunt SE, Ruffier M, Cunningham F, Dyer S, Finn RD, Howe KL, Harrison PW, Yates AD, Flicek P. Ensembl 2023. Nucleic Acids Res. 2023 Jan 6;51(D1):D933-D941. doi: 10.1093/nar/gkac958. PMID: 36318249; PMCID: PMC9825606.
+Sayers EW, Beck J, Bolton EE, Brister JR, Chan J, Comeau DC, Connor R, DiCuccio M, Farrell CM, Feldgarden M, Fine AM, Funk K, Hatcher E, Hoeppner M, Kane M, Kannan S, Katz KS, Kelly C, Klimke W, Kim S, Kimchi A, Landrum M, Lathrop S, Lu Z, Malheiro A, Marchler-Bauer A, Murphy TD, Phan L, Prasad AB, Pujar S, Sawyer A, Schmieder E, Schneider VA, Schoch CL, Sharma S, Thibaud-Nissen F, Trawick BW, Venkatapathi T, Wang J, Pruitt KD, Sherry ST. Database resources of the National Center for Biotechnology Information. Nucleic Acids Res. 2024 Jan 5;52(D1):D33-D43. doi: 10.1093/nar/gkad1044. PMID: 37994677; PMCID: PMC10767890.
+The UniProt Consortium , UniProt: the Universal Protein Knowledgebase in 2023, Nucleic Acids Research, Volume 51, Issue D1, 6 January 2023, Pages D523–D531, https://doi.org/10.1093/nar/gkac1052
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Align multiple nucleotide or amino acid sequences to each other using Muscle5.
+Return format: ClustalW formatted standard out or aligned FASTA (.afa).
Positional argument
+fasta
+List of sequences or path to FASTA or .txt file containing the nucleotide or amino acid sequences to be aligned.
Optional arguments
+-o --out
+Path to the aligned FASTA file the results will be saved in, e.g. path/to/directory/results.afa. Default: Standard out.
+Python: save=True will save the output in the current working directory.
Flags
+-s5 --super5
+Aligns input using the Super5 algorithm instead of the Parallel Perturbed Probcons (PPP) algorithm to decrease time and memory.
+Use for large inputs (a few hundred sequences).
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
gget muscle MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS
+# Python
+gget.muscle(["MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS", "MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS"])
+gget muscle fasta.fa
+# Python
+gget.muscle("fasta.fa")
+→ Returns an overview of the aligned sequences with ClustalW coloring. (To return an aligned FASTA (.afa) file, use --out argument (or save=True in Jupyter Lab/Google Colab).) In the above example, the 'fasta.fa' includes several sequences to be aligned (e.g. isoforms returned from gget seq).

You can also view aligned fasta files returned by gget.muscle using programs like alv, as shown below:
# Python
+!pip install biopython
+!pip install alv
+from Bio import AlignIO
+import alv
+
+gget.muscle("fasta.fa", out="fasta_aligned.afa")
+msa = AlignIO.read("fasta_aligned.afa", "fasta")
+alv.view(msa)
+If you use gget muscle in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Edgar RC (2021), MUSCLE v5 enables improved estimates of phylogenetic tree confidence by ensemble bootstrapping, bioRxiv 2021.06.20.449169. https://doi.org/10.1101/2021.06.20.449169
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Takes in nucleotide sequences and mutations (in standard mutation annotation and returns mutated versions of the input sequences according to the provided mutations.
+Return format: Saves mutated sequences in FASTA format (or returns a list containing the mutated sequences if out=None).
This module was written by Joseph Rich.
+Positional argument
+sequences
+Path to the FASTA file containing the sequences to be mutated, e.g., 'path/to/seqs.fa'.
+Sequence identifiers following the '>' character must correspond to the identifiers in the seq_ID column of mutations.
Example format of the FASTA file:
+>seq1 (or ENSG00000106443)
+ACTGCGATAGACT
+>seq2
+AGATCGCTAG
+Alternatively: Input sequence(s) as a string or list, e.g. 'AGCTAGCT'.
+NOTE: Only the letters until the first space or dot will be used as sequence identifiers - Version numbers of Ensembl IDs will be ignored.
+NOTE: When the sequences input is a genome fasta file, also see the gtf argument below.
Required arguments
+-m --mutations
+Path to the csv or tsv file (e.g., 'path/to/mutations.csv') or data frame (DataFrame object) containing information about the mutations in the following format (the 'notes' and 'mut_ID' columns are optional):
| mutation | mut_ID | seq_ID | notes |
|---|---|---|---|
| c.2C>T | mut1 | seq1 | -> Apply mutation 1 to sequence 1 |
| c.9_13inv | mut2 | seq2 | -> Apply mutation 2 to sequence 2 |
| c.9_13inv | mut2 | seq4 | -> Apply mutation 2 to sequence 4 |
| c.9_13delinsAAT | mut3 | seq4 | -> Apply mutation 3 to sequence 4 |
| ... | ... | ... |
'mutation' = Column containing the mutations to be performed written in standard mutation annotation
+'mut_ID' = Column containing the identifier for each mutation
+'seq_ID' = Column containing the identifiers of the sequences to be mutated (must correspond to the string following the '>' character in the 'sequences' FASTA file; do NOT include spaces or dots)
Alternatively: Input mutation(s) as a string or list, e.g., 'c.2C>T'.
+If a list is provided, the number of mutations must equal the number of input sequences.
For use from the terminal (bash): Enclose individual mutation annotations in quotation marks to prevent parsing errors.
+Optional input-related arguments
+-mc --mut_column
+Name of the column containing the mutations to be performed in mutations. Default: 'mutation'.
-sic --seq_id_column
+Name of the column containing the IDs of the sequences to be mutated in mutations. Default: 'seq_ID'.
-mic --mut_id_column
+Name of the column containing the IDs of each mutation in mutations. Default: Same as mut_column.
-gtf --gtf
+Path to a .gtf file. When providing a genome fasta file as input for 'sequences', you can provide a .gtf file here and the input sequences will be defined according to the transcript boundaries, e.g. 'path/to/genome_annotation.gtf'. Default: None
-gtic --gtf_transcript_id_column
+Column name in the input mutations file containing the transcript ID. In this case, column seq_id_column should contain the chromosome number.
+Required when gtf is provided. Default: None
Optional mutant sequence generation/filtering arguments
+-k --k
+Length of sequences flanking the mutation. Default: 30.
+If k > total length of the sequence, the entire sequence will be kept.
-msl --min_seq_len
+Minimum length of the mutant output sequence, e.g. 100. Mutant sequences smaller than this will be dropped. Default: None
-ma --max_ambiguous
+Maximum number of 'N' (or 'n') characters allowed in the output sequence, e.g. 10. Default: None (no ambiguous character filter will be applied)
Optional mutant sequence generation/filtering flags
+-ofr --optimize_flanking_regions
+Removes nucleotides from either end of the mutant sequence to ensure (when possible) that the mutant sequence does not contain any k-mers also found in the wildtype/input sequence.
-rswk --remove_seqs_with_wt_kmers
+Removes output sequences where at least one k-mer is also present in the wildtype/input sequence in the same region.
+When used with --optimize_flanking_regions, only sequences for which a wildtype k-mer is still present after optimization will be removed.
-mio --merge_identical_off
+Do not merge identical mutant sequences in the output (by default, identical sequences will be merged by concatenating the sequence headers for all identical sequences).
Optional arguments to generate additional output
+This output is activated using the --update_df flag and will be stored in a copy of the mutations DataFrame.
-udf_o --update_df_out
+Path to output csv file containing the updated DataFrame, e.g. 'path/to/mutations_updated.csv'. Only valid when used with --update_df.
+Default: None -> the new csv file will be saved in the same directory as the mutations DataFrame with appendix '_updated'
-ts --translate_start
+(int or str) The position in the input nucleotide sequence to start translating, e.g. 5. If a string is provided, it should correspond to a column name in mutations containing the open reading frame start positions for each sequence/mutation. Only valid when used with --translate.
+Default: translates from the beginning of each sequence
-te --translate_end
+(int or str) The position in the input nucleotide sequence to end translating, e.g. 35. If a string is provided, it should correspond to a column name in mutations containing the open reading frame end positions for each sequence/mutation. Only valid when used with --translate.
+Default: translates until the end of each sequence
Optional flags to modify additional output
+-udf --update_df
+Updates the input mutations DataFrame to include additional columns with the mutation type, wildtype nucleotide sequence, and mutant nucleotide sequence (only valid if mutations is a .csv or .tsv file).
-sfs --store_full_sequences
+Includes the complete wildtype and mutant sequences in the updated mutations DataFrame (not just the sub-sequence with k-length flanks). Only valid when used with --update_df.
-tr --translate
+Adds additional columns to the updated mutations DataFrame containing the wildtype and mutant amino acid sequences. Only valid when used with --store_full_sequences.
Optional general arguments
+-o --out
+Path to output FASTA file containing the mutated sequences, e.g., 'path/to/output_fasta.fa'.
+Default: None -> returns a list of the mutated sequences to standard out.
+The identifiers (following the '>') of the mutated sequences in the output FASTA will be '>[seq_ID]_[mut_ID]'.
Optional general flags
+-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
gget mutate ATCGCTAAGCT -m 'c.4G>T'
+# Python
+gget.mutate("ATCGCTAAGCT", "c.4G>T")
+→ Returns ATCTCTAAGCT.
+List of sequences with a mutation for each sequence provided in a list:
+gget mutate ATCGCTAAGCT TAGCTA -m 'c.4G>T' 'c.1_3inv' -o mut_fasta.fa
+# Python
+gget.mutate(["ATCGCTAAGCT", "TAGCTA"], ["c.4G>T", "c.1_3inv"], out="mut_fasta.fa")
+→ Saves 'mut_fasta.fa' file containing:
+>seq1_mut1
+ATCTCTAAGCT
+>seq2_mut2
+GATCTA
+One mutation applied to several sequences with adjusted k:
gget mutate ATCGCTAAGCT TAGCTA -m 'c.1_3inv' -k 3
+# Python
+gget.mutate(["ATCGCTAAGCT", "TAGCTA"], "c.1_3inv", k=3)
+→ Returns ['CTAGCT', 'GATCTA'].
+Add mutations to an entire genome with extended output
+Main input:
mutations CSV (by having seq_id_column contain chromosome information, and mut_column contain mutation information with respect to genome coordinates)sequences fileSince we are passing the path to a gtf file to the gtf argument, transcript boundaries will be respected (the genome will be split into transcripts). gtf_transcript_id_column specifies the name of the column in mutations containing the transcript IDs corresponding to the transcript IDs in the gtf file.
The optimize_flanking_regions argument maximizes the length of the resulting mutation-containing sequences while maintaining specificity (no wildtype k-mer will be retained).
update_df activates the creation of a new CSV file with updated information about each input and output sequence. This new CSV file will be saved as update_df_out. Since store_full_sequences is activated, this new CSV file will not only contain the output sequences (restricted in size by flanking regiong of size k), but also the complete input and output sequences. This allows us to observe the mutation in the context of the entire sequence. Lastly, we are also adding the translated versions of the complete sequences by adding the with the translate flag, so we can observe how the resulting amino acid sequence is changed. The translate_start and translate_end arguments specify the names of the columns in mutations that contain the start and end positions of the open reading frame (start and end positions for translating the nucleotide sequence to an amino acid sequence), respectively.
gget mutate \
+ -m mutations_input.csv \
+ -o mut_fasta.fa \
+ -k 4 \
+ -sic Chromosome \
+ -mic Mutation \
+ -gtf genome_annotation.gtf \
+ -gtic Ensembl_Transcript_ID \
+ -ofr \
+ -update_df \
+ -udf_o mutations_updated.csv \
+ -sfs \
+ -tr \
+ -ts Translate_Start \
+ -te Translate_End \
+ genome_reference.fa
+# Python
+gget.mutate(sequences="genome_reference.fa", mutations="mutations_input.csv", out="mut_fasta.fa", k=4, seq_id_column="Chromosome", mut_column="Mutation", gtf="genome_annotation.gtf", gtf_transcript_id_column="Ensembl_Transcript_ID", optimize_flanking_regions=True, update_df=True, update_df_out="mutations_updated.csv", store_full_sequences=True, translate=True, translate_start="Translate_Start", translate_end="Translate_End")
+→ Takes as input 'mutations_input.csv' file containing:
+| Chromosome | Mutation | Ensembl_Transcript_ID | Translate_Start | Translate_End |
+|------------|-------------------|------------------------|-----------------|---------------|
+| 1 | g.224411A>C | ENST00000193812 | 0 | 100 |
+| 8 | g.25111del | ENST00000174411 | 0 | 294 |
+| X | g.1011_1012insAA | ENST00000421914 | 9 | 1211 |
+→ Saves 'mut_fasta.fa' file containing:
+>1:g.224411A>C
+TGCTCTGCT
+>8:g.25111del
+GAGTCGAT
+>X:g.1011_1012insAA
+TTAGAACTT
+→ Saves 'mutations_updated.csv' file containing:
+
+| Chromosome | Mutation | Ensembl_Transcript_ID | mutation_type | wt_sequence | mutant_sequence | wt_sequence_full | mutant_sequence_full | wt_sequence_aa_full | mutant_sequence_aa_full |
+|------------|-------------------|------------------------|---------------|-------------|-----------------|-------------------|----------------------|---------------------|-------------------------|
+| 1 | g.224411A>C | ENSMUST00000193812 | Substitution | TGCTATGCT | TGCTCTGCT | ...TGCTATGCT... | ...TGCTCTGCT... | ...CYA... | ...CSA... |
+| 8 | g.25111del | ENST00000174411 | Deletion | GAGTCCGAT | GAGTCGAT | ...GAGTCCGAT... | ...GAGTCGAT... | ...ESD... | ...ES... |
+| X | g.1011_1012insAA | ENST00000421914 | Insertion | TTAGCTT | TTAGAACTT | ...TTAGCTT... | ...TTAGAACTT... | ...A... | ...EL... |
+
+If you use gget mutate in a publication, please cite the following articles:
++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Fetch associated diseases or drugs from OpenTargets using Ensembl IDs.
+Return format: JSON/CSV (command-line) or data frame (Python).
This module was written by Sam Wagenaar.
+Positional argument
+ens_id
+Ensembl gene ID, e.g ENSG00000169194.
Optional arguments
+-r --resource
+Defines the type of information to return in the output. Default: 'diseases'.
+Possible resources are:
| Resource | Return Value | Valid Filters | Sources |
|---|---|---|---|
diseases | Associated diseases | None | Various:etc. |
drugs | Associated drugs | disease_id | ChEMBL |
tractability | Tractability data | None | Open Targets |
pharmacogenetics | Pharmacogenetic responses | drug_id | PharmGKB |
expression | Gene expression data (by tissues, organs, and anatomical systems) | tissue_idanatomical_systemorgan | |
depmap | DepMap gene→disease-effect data. | tissue_id | DepMap Portal |
interactions | Protein⇄protein interactions | protein_a_idprotein_b_idgene_b_id |
-l --limit
+Limit the number of results, e.g 10. Default: No limit.
+Note: Not compatible with the tractability and depmap resources.
-o --out
+Path to the JSON file the results will be saved in, e.g. path/to/directory/results.json. Default: Standard out.
+Python: save=True will save the output in the current working directory.
Optional filter arguments
+-fd --filter_disease disease_id
+Filter by disease ID, e.g. 'EFO_0000274'. Only valid for the drugs resource.
-fc --filter_drug drug_id
+Filter by drug ID, e.g. 'CHEMBL1743081'. Only valid for the pharmacogenetics resource.
-ft --filter_tissue tissue_id
+Filter by tissue ID, e.g. 'UBERON_0000473'. Only valid for the expression and depmap resources.
-fa --filter_anat_sys
+Filter by anatomical system, e.g. 'nervous system'. Only valid for the expression resource.
-fo --filter_organ anatomical_system
+Filter by organ, e.g. 'brain'. Only valid for the expression resource.
-fpa --filter_protein_a protein_a_id
+Filter by the protein ID of the first protein in the interaction, e.g. 'ENSP00000304915'. Only valid for the interactions resource.
-fpb --filter_protein_b protein_b_id
+Filter by the protein ID of the second protein in the interaction, e.g. 'ENSP00000379111'. Only valid for the interactions resource.
-fgb --filter_gene_b gene_b_id
+Filter by the gene ID of the second protein in the interaction, e.g. 'ENSG00000077238'. Only valid for the interactions resource.
filters
+Python only. A dictionary of filters, e.g.
{'disease_id': ['EFO_0000274', 'HP_0000964']}
+filter_mode
+Python only. filter_mode='or' combines filters of different IDs with OR logic.
+filter_mode='and' combines filters of different IDs with AND logic (default).
Flags
+-csv --csv
+Command-line only. Returns the output in CSV format, instead of JSON format.
+Python: Use json=True to return output in JSON format.
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
-or --or
+Command-line only. Filters are combined with OR logic. Default: AND logic.
wrap_text
+Python only. wrap_text=True displays data frame with wrapped text for easy reading (default: False).
Get associated diseases for a specific gene:
+gget opentargets ENSG00000169194 -r diseases -l 1
+# Python
+import gget
+gget.opentargets('ENSG00000169194', resource='diseases', limit=1)
+→ Returns the top disease associated with the gene ENSG00000169194.
+| id | name | description | score |
|---|---|---|---|
| EFO_0000274 | atopic eczema | A chronic inflammatory genetically determined disease of the skin ... | 0.66364347241831 |
Get associated drugs for a specific gene:
+gget opentargets ENSG00000169194 -r drugs -l 2
+# Python
+import gget
+gget.opentargets('ENSG00000169194', resource='drugs', limit=2)
+→ Returns the top 2 drugs associated with the gene ENSG00000169194.
+| id | name | type | action_mechanism | description | synonyms | trade_names | disease_id | disease_name | trial_phase | trial_status | trial_ids | approved |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CHEMBL1743081 | TRALOKINUMAB | Antibody | Interleukin‑13 inhibitor | Antibody drug with a maximum clinical trial phase of IV ... | ['CAT-354', 'Tralokinumab'] | ['Adbry', 'Adtralza'] | EFO_0000274 | atopic eczema | 4 | [] | True | |
| CHEMBL4297864 | CENDAKIMAB | Antibody | Interleukin‑13 inhibitor | Antibody drug with a maximum clinical trial phase of III ... | [ABT-308, Abt-308, CC-93538, Cendakimab, RPC-4046] | [] | EFO_0004232 | eosinophilic esophagitis | 3 | Recruiting | [NCT04991935] | False |
Note: Returned trial_ids are ClinicalTrials.gov identifiers
Get tractability data for a specific gene:
+gget opentargets ENSG00000169194 -r tractability
+# Python
+import gget
+gget.opentargets('ENSG00000169194', resource='tractability')
+→ Returns tractability data for the gene ENSG00000169194.
+| label | modality |
|---|---|
| High-Quality Pocket | Small molecule |
| Approved Drug | Antibody |
| GO CC high conf | Antibody |
| UniProt loc med conf | Antibody |
| UniProt SigP or TMHMM | Antibody |
Get pharmacogenetic responses for a specific gene:
+gget opentargets ENSG00000169194 -r pharmacogenetics -l 1
+# Python
+import gget
+gget.opentargets('ENSG00000169194', resource='pharmacogenetics', limit=1)
+→ Returns pharmacogenetic responses for the gene ENSG00000169194.
+| rs_id | genotype_id | genotype | variant_consequence_id | variant_consequence_label | drugs | phenotype | genotype_annotation | response_category | direct_target | evidence_level | source | literature |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| rs1295686 | 5_132660151_T_T,T | TT | SO:0002073 | no_sequence_alteration | id name 0 None hepatitis vaccines | increased risk for non‑immune response to the hepatitis B vaccine | Patients with the TT genotype may be at increased risk for non-immune response to the hepatitis B vaccine... | efficacy | False | 3 | pharmgkb | [21111021] |
Note: Returned literature ids are Europe PMC identifiers
Get tissues where a gene is most expressed:
+gget opentargets ENSG00000169194 -r expression -l 2
+# Python
+import gget
+gget.opentargets('ENSG00000169194', resource='expression', limit=2)
+→ Returns the top 2 tissues where the gene ENSG00000169194 is most expressed.
+| tissue_id | tissue_name | rna_zscore | rna_value | rna_unit | rna_level | anatomical_systems | organs |
|---|---|---|---|---|---|---|---|
| UBERON_0000473 | testis | 5 | 1026 | 3 | [reproductive system] | [reproductive organ, reproductive structure] | |
| CL_0000542 | EBV‑transformed lymphocyte | 1 | 54 | 2 | [hemolymphoid system, immune system, lymphoid system] | [immune organ] |
Get DepMap gene-disease effect data for a specific gene:
+gget opentargets ENSG00000169194 -r depmap
+# Python
+import gget
+gget.opentargets('ENSG00000169194', resource='depmap')
+→ Returns DepMap gene-disease effect data for the gene ENSG00000169194.
+| depmap_id | expression | effect | tissue_id | tissue_name | cell_line_name | disease_cell_line_id | disease_name | mutation |
|---|---|---|---|---|---|---|---|---|
| ACH‑001532 | 0.176323 | 0.054950 | UBERON_0002113 | kidney | JMU-RTK-2 | None | Rhabdoid Cancer | None |
Get protein-protein interactions for a specific gene:
+gget opentargets ENSG00000169194 -r interactions -l 2
+# Python
+import gget
+gget.opentargets('ENSG00000169194', resource='interactions', limit=2)
+→ Returns the top 2 protein-protein interactions for the gene ENSG00000169194.
+| evidence_score | evidence_count | source_db | protein_a_id | gene_a_id | gene_a_symbol | role_a | taxon_a | protein_b_id | gene_b_id | gene_b_symbol | role_b | taxon_b |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0.999 | 3 | string | ENSP00000304915 | ENSG00000169194 | IL13 | unspecified role | 9606 | ENSP00000379111 | ENSG00000077238 | IL4R | unspecified role | 9606 |
| 0.999 | 3 | string | ENSP00000304915 | ENSG00000169194 | IL13 | unspecified role | 9606 | ENSP00000360730 | ENSG00000131724 | IL13RA1 | unspecified role | 9606 |
Get protein-protein interactions for a specific gene, filtering by protein and gene IDs:
+gget opentargets ENSG00000169194 -r interactions -fpa P35225 --filter_gene_b ENSG00000077238
+# Python
+import gget
+gget.opentargets('ENSG00000169194', resource='interactions', filters={'protein_a_id': 'P35225', 'gene_b_id': 'ENSG00000077238'})
+→ Returns protein-protein interactions for the gene ENSG00000169194, where the first protein is P35225 and the second gene is ENSG00000077238.
+| evidence_score | evidence_count | source_db | protein_a_id | gene_a_id | gene_a_symbol | role_a | taxon_a | protein_b_id | gene_b_id | gene_b_symbol | role_b | taxon_b |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| None | 3 | reactome | P35225 | ENSG00000169194 | IL13 | unspecified role | 9606 | P24394 | ENSG00000077238 | IL4R | unspecified role | 9606 |
| None | 2 | signor | P35225 | ENSG00000169194 | IL13 | regulator | 9606 | P24394 | ENSG00000077238 | IL4R | regulator target | 9606 |
Get protein-protein interactions for a specific gene, filtering by protein or gene IDs:
+gget opentargets ENSG00000169194 -r interactions -fpa P35225 --filter_gene_b ENSG00000077238 ENSG00000111537 --or -l 5
+# Python
+import gget
+gget.opentargets(
+ 'ENSG00000169194',
+ resource='interactions',
+ filters={'protein_a_id': 'P35225', 'gene_b_id': ['ENSG00000077238', 'ENSG00000111537']},
+ filter_mode='or',
+ limit=5
+)
+→ Returns protein-protein interactions for the gene ENSG00000169194, where the first protein is P35225 or the second gene is either ENSG00000077238 or ENSG00000111537.
+| evidence_score | evidence_count | source_db | protein_a_id | gene_a_id | gene_a_symbol | role_a | taxon_a | protein_b_id | gene_b_id | gene_b_symbol | role_b | taxon_b |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0.999 | 3 | string | ENSP00000304915 | ENSG00000169194 | IL13 | unspecified role | 9606 | ENSP00000379111 | ENSG00000077238 | IL4R | unspecified role | 9606 |
| 0.961 | 2 | string | ENSP00000304915 | ENSG00000169194 | IL13 | unspecified role | 9606 | ENSP00000229135 | ENSG00000111537 | IFNG | unspecified role | 9606 |
| 0.800 | 9 | intact | P35225 | ENSG00000169194 | IL13 | unspecified role | 9606 | Q14627 | ENSG00000123496 | IL13RA2 | unspecified role | 9606 |
| 0.740 | 6 | intact | P35225 | ENSG00000169194 | IL13 | unspecified role | 9606 | P78552 | ENSG00000131724 | IL13RA1 | unspecified role | 9606 |
| 0.400 | 1 | intact | P35225 | ENSG00000169194 | IL13 | unspecified role | 9606 | Q86XT9 | ENSG00000149932 | TMEM219 | stimulator | 9606 |
If you use gget opentargets in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Ochoa D, Hercules A, Carmona M, Suveges D, Baker J, Malangone C, Lopez I, Miranda A, Cruz-Castillo C, Fumis L, Bernal-Llinares M, Tsukanov K, Cornu H, Tsirigos K, Razuvayevskaya O, Buniello A, Schwartzentruber J, Karim M, Ariano B, Martinez Osorio RE, Ferrer J, Ge X, Machlitt-Northen S, Gonzalez-Uriarte A, Saha S, Tirunagari S, Mehta C, Roldán-Romero JM, Horswell S, Young S, Ghoussaini M, Hulcoop DG, Dunham I, McDonagh EM. The next-generation Open Targets Platform: reimagined, redesigned, rebuilt. Nucleic Acids Res. 2023 Jan 6;51(D1):D1353-D1359. doi: 10.1093/nar/gkac1046. PMID: 36399499; PMCID: PMC9825572.
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Query RCSB Protein Data Bank (PDB) for the protein structure/metadata of a given PDB ID.
+Return format: Resource 'pdb' is returned in PDB format. All other resources are returned in JSON format.
Positional argument
+pdb_id
+PDB ID to be queried, e.g. '7S7U'.
Optional arguments
+-r --resource
+Defines type of information to be returned. One of the following:
+'pdb': Returns the protein structure in PDB format (default).
+'entry': Information about PDB structures at the top level of PDB structure hierarchical data organization.
+'pubmed': Get PubMed annotations (data integrated from PubMed) for a given entry's primary citation.
+'assembly': Information about PDB structures at the quaternary structure level.
+'branched_entity': Get branched entity description (define entity ID as 'identifier').
+'nonpolymer_entity': Get non-polymer entity data (define entity ID as 'identifier').
+'polymer_entity': Get polymer entity data (define entity ID as 'identifier').
+'uniprot': Get UniProt annotations for a given macromolecular entity (define entity ID as 'identifier').
+'branched_entity_instance': Get branched entity instance description (define chain ID as 'identifier').
+'polymer_entity_instance': Get polymer entity instance (a.k.a chain) data (define chain ID as 'identifier').
+'nonpolymer_entity_instance': Get non-polymer entity instance description (define chain ID as 'identifier').
-i --identifier
+Can be used to define assembly, entity or chain ID (default: None). Assembly/entity IDs are numbers (e.g. 1), and chain IDs are letters (e.g. 'A').
-o --out
+Path to the file the results will be saved in, e.g. path/to/directory/7S7U.pdb or path/to/directory/7S7U_entry.json. Default: Standard out.
+Python: save=True will save the output in the current working directory.
gget pdb 7S7U -o 7S7U.pdb
+# Python
+gget.pdb("7S7U", save=True)
+→ Saves the structure of 7S7U in PDB format as '7S7U.pdb' in the current working directory.
+Find PDB crystal structures for a comparative analysis of protein structure:
+# Find PDB IDs associated with an Ensembl ID
+gget info ENSG00000130234
+
+# Alternatively: Since many entries in the PDB do not have linked Ensembl IDs,
+# you will likely find more PDB entries by BLASTing the sequence agains the PDB.
+
+# Get the amino acid sequence of a transcript from an Ensembl ID
+gget seq --translate ENSG00000130234 -o gget_seq_results.fa
+
+# BLAST an amino acid sequence to find similar structures in the PDB
+gget blast --database pdbaa gget_seq_results.fa
+
+# Get PDB files from the PDB IDs returned by gget blast for comparative analysis
+gget pdb 7DQA -o 7DQA.pdb
+gget pdb 7CT5 -o 7CT5.pdb
+# Find PDB IDs associated with an Ensembl ID
+gget.info("ENSG00000130234")
+
+# Alternatively: Since many entries in the PDB do not have linked Ensembl IDs,
+# you will likely find more PDB entries by BLASTing the sequence agains the PDB.
+
+# Get the amino acid sequence of a transcript from an Ensembl ID
+gget.seq("ENSG00000130234", translate=True, save=True)
+
+# BLAST an amino acid sequence to find similar structures in the PDB
+gget.blast("gget_seq_results.fa", database="pdbaa")
+
+# Get PDB files from the PDB IDs returned by gget blast for comparative analysis
+gget.pdb("7DQA", save=True)
+gget.pdb("7CT5", save=True)
+→ The use case above exemplifies how to find PDB files for comparative analysis of protein structure starting with Ensembl IDs or amino acid sequences. The fetched PDB files can also be compared to predicted structures generated by gget alphafold. PDB files can be viewed interactively in 3D online, or using programs like PyMOL or Blender. To compare two PDB files, you can use this website.
If you use gget pdb in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Berman HM, Westbrook J, Feng Z, Gilliland G, Bhat TN, Weissig H, Shindyalov IN, Bourne PE. The Protein Data Bank. Nucleic Acids Res. 2000 Jan 1;28(1):235-42. doi: 10.1093/nar/28.1.235. PMID: 10592235; PMCID: PMC102472.
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Fetch FTPs and their respective metadata (or use flag ftp to only return the links) for reference genomes and annotations from Ensembl by species.
+Return format: dictionary/JSON.
Positional argument
+species
+Species for which the FTPs will be fetched in the format genus_species, e.g. homo_sapiens.
+Supports all available vertebrate and invertebrate (plants, fungi, protists, and invertebrate metazoa) genomes from Ensembl, except bacteria.
+Note: Not required when using flags --list_species or --list_iv_species.
+Supported shortcuts: 'human', 'mouse', 'human_grch37' (accesses the GRCh37 genome assembly)
Optional arguments
+-w --which
+Defines which results to return. Default: 'all' -> Returns all available results.
+Possible entries are one or a combination (as comma-separated list) of the following:
+'gtf' - Returns the annotation (GTF).
+'cdna' - Returns the trancriptome (cDNA).
+'dna' - Returns the genome (DNA).
+'cds' - Returns the coding sequences corresponding to Ensembl genes. (Does not contain UTR or intronic sequence.)
+'cdrna' - Returns transcript sequences corresponding to non-coding RNA genes (ncRNA).
+'pep' - Returns the protein translations of Ensembl genes.
-r --release
+Defines the Ensembl release number from which the files are fetched, e.g. 104. Default: latest Ensembl release.
-od --out_dir
+Path to the directory where the FTPs will be saved, e.g. path/to/directory/. Default: Current working directory.
-o --out
+Path to the JSON file the results will be saved in, e.g. path/to/directory/results.json. Default: Standard out.
+Python: save=True will save the output in the current working directory.
Flags
+-l --list_species
+Lists all available vertebrate species. (Python: combine with species=None.)
-liv --list_iv_species
+Lists all available invertebrate species. (Python: combine with species=None.)
-ftp --ftp
+Returns only the requested FTP links.
-d --download
+Command-line only. Downloads the requested FTPs to the directory specified by out_dir (requires curl to be installed).
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
Use gget ref in combination with kallisto | bustools to build a reference index:
kb ref -i INDEX -g T2G -f1 FASTA $(gget ref --ftp -w dna,gtf homo_sapiens)
+→ kb ref builds a reference index using the latest DNA and GTF files of species Homo sapiens passed to it by gget ref.
List all available genomes from Ensembl release 103:
+gget ref --list_species -r 103
+# Python
+gget.ref(species=None, list_species=True, release=103)
+→ Returns a list with all available genomes (checks if GTF and FASTAs are available) from Ensembl release 103.
+(If no release is specified, gget ref will always return information from the latest Ensembl release.)
Get the genome reference for a specific species:
+gget ref -w gtf,dna homo_sapiens
+# Python
+gget.ref("homo_sapiens", which=["gtf", "dna"])
+→ Returns a JSON with the latest human GTF and FASTA FTPs, and their respective metadata, in the format:
+{
+ "homo_sapiens": {
+ "annotation_gtf": {
+ "ftp": "http://ftp.ensembl.org/pub/release-106/gtf/homo_sapiens/Homo_sapiens.GRCh38.106.gtf.gz",
+ "ensembl_release": 106,
+ "release_date": "28-Feb-2022",
+ "release_time": "23:27",
+ "bytes": "51379459"
+ },
+ "genome_dna": {
+ "ftp": "http://ftp.ensembl.org/pub/release-106/fasta/homo_sapiens/dna/Homo_sapiens.GRCh38.dna.primary_assembly.fa.gz",
+ "ensembl_release": 106,
+ "release_date": "21-Feb-2022",
+ "release_time": "09:35",
+ "bytes": "881211416"
+ }
+ }
+}
+If you use gget ref in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Martin FJ, Amode MR, Aneja A, Austine-Orimoloye O, Azov AG, Barnes I, Becker A, Bennett R, Berry A, Bhai J, Bhurji SK, Bignell A, Boddu S, Branco Lins PR, Brooks L, Ramaraju SB, Charkhchi M, Cockburn A, Da Rin Fiorretto L, Davidson C, Dodiya K, Donaldson S, El Houdaigui B, El Naboulsi T, Fatima R, Giron CG, Genez T, Ghattaoraya GS, Martinez JG, Guijarro C, Hardy M, Hollis Z, Hourlier T, Hunt T, Kay M, Kaykala V, Le T, Lemos D, Marques-Coelho D, Marugán JC, Merino GA, Mirabueno LP, Mushtaq A, Hossain SN, Ogeh DN, Sakthivel MP, Parker A, Perry M, Piližota I, Prosovetskaia I, Pérez-Silva JG, Salam AIA, Saraiva-Agostinho N, Schuilenburg H, Sheppard D, Sinha S, Sipos B, Stark W, Steed E, Sukumaran R, Sumathipala D, Suner MM, Surapaneni L, Sutinen K, Szpak M, Tricomi FF, Urbina-Gómez D, Veidenberg A, Walsh TA, Walts B, Wass E, Willhoft N, Allen J, Alvarez-Jarreta J, Chakiachvili M, Flint B, Giorgetti S, Haggerty L, Ilsley GR, Loveland JE, Moore B, Mudge JM, Tate J, Thybert D, Trevanion SJ, Winterbottom A, Frankish A, Hunt SE, Ruffier M, Cunningham F, Dyer S, Finn RD, Howe KL, Harrison PW, Yates AD, Flicek P. Ensembl 2023. Nucleic Acids Res. 2023 Jan 6;51(D1):D933-D941. doi: 10.1093/nar/gkac958. PMID: 36318249; PMCID: PMC9825606.
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Fetch genes and transcripts from Ensembl using free-form search terms.
+Results are matched based on the "gene name" and "description" sections in the Ensembl database. gget version >= 0.27.9 also includes results that match the Ensembl "synonym" section.
+Return format: JSON (command-line) or data frame/CSV (Python).
Positional argument
+searchwords
+One or more free form search words, e.g. gaba nmda. (Note: Search is not case-sensitive.)
Other required arguments
+-s --species
+Species or database to be searched.
+A species can be passed in the format 'genus_species', e.g. 'homo_sapiens' or 'arabidopsis_thaliana'.
+To pass a specific database, pass the name of the CORE database, e.g. 'mus_musculus_dba2j_core_105_1'.
All available core databases can be found here:
+Vertebrates: http://ftp.ensembl.org/pub/current/mysql/
+Invertebrates: http://ftp.ensemblgenomes.org/pub/current/ + select kingdom + go to mysql/
Supported shortcuts: 'human', 'mouse'
+Optional arguments
+-r --release
+Defines the Ensembl release number from which the files are fetched, e.g. 104. Default: None -> latest Ensembl release is used.
Note: The release argument does not apply to invertebrate species (you can pass a specific core database (which includes a release number) to the species argument instead). For invertebrate species, Ensembl only stores databases from 10 releases prior to the current release.
This argument is overwritten if a specific database (which includes a release number) is passed to the species argument.
+-t --id_type
+'gene' (default) or 'transcript'
+Returns genes or transcripts, respectively.
-ao --andor
+'or' (default) or 'and'
+'or': Returns all genes that INCLUDE AT LEAST ONE of the searchwords in their name/description.
+'and': Returns only genes that INCLUDE ALL of the searchwords in their name/description.
-l --limit
+Limits the number of search results, e.g. 10. Default: None.
-o --out
+Path to the csv the results will be saved in, e.g. path/to/directory/results.csv (or .json). Default: Standard out.
+Python: save=True will save the output in the current working directory.
Flags
+-csv --csv
+Command-line only. Returns results in CSV format.
+Python: Use json=True to return output in JSON format.
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
wrap_text
+Python only. wrap_text=True displays data frame with wrapped text for easy reading (default: False).
gget search -s human gaba gamma-aminobutyric
+# Python
+gget.search(["gaba", "gamma-aminobutyric"], "homo_sapiens")
+→ Returns all genes that contain at least one of the search words in their name or Ensembl/external reference description:
+| ensembl_id | gene_name | ensembl_description | ext_ref_description | biotype | url |
|---|---|---|---|---|---|
| ENSG00000034713 | GABARAPL2 | GABA type A receptor associated protein like 2 [Source:HGNC Symbol;Acc:HGNC:13291] | GABA type A receptor associated protein like 2 | protein_coding | https://uswest.ensembl.org/homo_sapiens/Gene/Summary?g=ENSG00000034713 |
| . . . | . . . | . . . | . . . | . . . | . . . |
If you use gget search in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Martin FJ, Amode MR, Aneja A, Austine-Orimoloye O, Azov AG, Barnes I, Becker A, Bennett R, Berry A, Bhai J, Bhurji SK, Bignell A, Boddu S, Branco Lins PR, Brooks L, Ramaraju SB, Charkhchi M, Cockburn A, Da Rin Fiorretto L, Davidson C, Dodiya K, Donaldson S, El Houdaigui B, El Naboulsi T, Fatima R, Giron CG, Genez T, Ghattaoraya GS, Martinez JG, Guijarro C, Hardy M, Hollis Z, Hourlier T, Hunt T, Kay M, Kaykala V, Le T, Lemos D, Marques-Coelho D, Marugán JC, Merino GA, Mirabueno LP, Mushtaq A, Hossain SN, Ogeh DN, Sakthivel MP, Parker A, Perry M, Piližota I, Prosovetskaia I, Pérez-Silva JG, Salam AIA, Saraiva-Agostinho N, Schuilenburg H, Sheppard D, Sinha S, Sipos B, Stark W, Steed E, Sukumaran R, Sumathipala D, Suner MM, Surapaneni L, Sutinen K, Szpak M, Tricomi FF, Urbina-Gómez D, Veidenberg A, Walsh TA, Walts B, Wass E, Willhoft N, Allen J, Alvarez-Jarreta J, Chakiachvili M, Flint B, Giorgetti S, Haggerty L, Ilsley GR, Loveland JE, Moore B, Mudge JM, Tate J, Thybert D, Trevanion SJ, Winterbottom A, Frankish A, Hunt SE, Ruffier M, Cunningham F, Dyer S, Finn RD, Howe KL, Harrison PW, Yates AD, Flicek P. Ensembl 2023. Nucleic Acids Res. 2023 Jan 6;51(D1):D933-D941. doi: 10.1093/nar/gkac958. PMID: 36318249; PMCID: PMC9825606.
+++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Function to install/download third-party dependencies for a specified gget module.
+Positional argument
+module
+gget module for which dependencies should be installed.
Optional arguments
+-o --out
+Path to the folder downloaded files will be saved in (currently only applies to module = 'elm').
+NOTE: Do NOT use this argument when downloading the files for use with gget.elm.
+Default: None (downloaded files are saved inside the gget package installation folder).
Flags
+-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
gget setup alphafold
+# Python
+gget.setup("alphafold")
+→ Installs all (modified) third-party dependencies and downloads model parameters (~4GB) required to run gget alphafold.
++Python arguments are equivalent to long-option arguments (
+--arg), unless otherwise specified. Flags are True/False arguments in Python. The manual for any gget tool can be called from the command-line using the-h--helpflag.
Fetch nucleotide or amino acid sequence(s) of a gene (and all its isoforms) or a transcript by Ensembl ID.
+Return format: FASTA.
Positional argument
+ens_ids
+One or more Ensembl IDs.
Optional arguments
+-o --out
+Path to the file the results will be saved in, e.g. path/to/directory/results.fa. Default: Standard out.
+Python: save=True will save the output in the current working directory.
Flags
+-t --translate
+Returns amino acid (instead of nucleotide) sequences.
+Nucleotide sequences are fetched from Ensembl.
+Amino acid sequences are fetched from UniProt.
-iso --isoforms
+Returns the sequences of all known transcripts.
+(Only for gene IDs.)
-q --quiet
+Command-line only. Prevents progress information from being displayed.
+Python: Use verbose=False to prevent progress information from being displayed.
gget seq ENSG00000034713 ENSG00000104853 ENSG00000170296
+# Python
+gget.seq(["ENSG00000034713", "ENSG00000104853", "ENSG00000170296"])
+→ Returns the nucleotide sequences of ENSG00000034713, ENSG00000104853, and ENSG00000170296 in FASTA format.
+gget seq -t -iso ENSG00000034713
+# Python
+gget.seq("ENSG00000034713", translate=True, isoforms=True)
+→ Returns the amino acid sequences of all known transcripts of ENSG00000034713 in FASTA format.
+If you use gget seq in a publication, please cite the following articles:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Martin FJ, Amode MR, Aneja A, Austine-Orimoloye O, Azov AG, Barnes I, Becker A, Bennett R, Berry A, Bhai J, Bhurji SK, Bignell A, Boddu S, Branco Lins PR, Brooks L, Ramaraju SB, Charkhchi M, Cockburn A, Da Rin Fiorretto L, Davidson C, Dodiya K, Donaldson S, El Houdaigui B, El Naboulsi T, Fatima R, Giron CG, Genez T, Ghattaoraya GS, Martinez JG, Guijarro C, Hardy M, Hollis Z, Hourlier T, Hunt T, Kay M, Kaykala V, Le T, Lemos D, Marques-Coelho D, Marugán JC, Merino GA, Mirabueno LP, Mushtaq A, Hossain SN, Ogeh DN, Sakthivel MP, Parker A, Perry M, Piližota I, Prosovetskaia I, Pérez-Silva JG, Salam AIA, Saraiva-Agostinho N, Schuilenburg H, Sheppard D, Sinha S, Sipos B, Stark W, Steed E, Sukumaran R, Sumathipala D, Suner MM, Surapaneni L, Sutinen K, Szpak M, Tricomi FF, Urbina-Gómez D, Veidenberg A, Walsh TA, Walts B, Wass E, Willhoft N, Allen J, Alvarez-Jarreta J, Chakiachvili M, Flint B, Giorgetti S, Haggerty L, Ilsley GR, Loveland JE, Moore B, Mudge JM, Tate J, Thybert D, Trevanion SJ, Winterbottom A, Frankish A, Hunt SE, Ruffier M, Cunningham F, Dyer S, Finn RD, Howe KL, Harrison PW, Yates AD, Flicek P. Ensembl 2023. Nucleic Acids Res. 2023 Jan 6;51(D1):D933-D941. doi: 10.1093/nar/gkac958. PMID: 36318249; PMCID: PMC9825606
+The UniProt Consortium , UniProt: the Universal Protein Knowledgebase in 2023, Nucleic Acids Research, Volume 51, Issue D1, 6 January 2023, Pages D523–D531, https://doi.org/10.1093/nar/gkac1052
+Thank you for investing your time in contributing to our project! Any contribution you make will be reflected on the gget repo. ✨
+Read our Code of Conduct to keep our community approachable and respectable.
+In this guide you will get an overview of the contribution workflow from opening an issue or creating a pull request (PR) to reviewing and merging a PR.
+If you spot a problem with gget or you have an idea for a new feature, check if an issue already exists. If a related issue doesn't exist, you can open a new issue using the relevant issue form.
+Scan through our existing issues to find one that interests you. You can narrow down the search using labels as filters. If you find an issue to work on, you are welcome to open a PR with a fix.
Using GitHub Desktop:
+Using the command line:
+Commit the changes once you are happy with them.
+pip install -r dev-requirements.txtcoverage run -m pytest -ra -v tests && coverage report --omit=main.py,tests*If you have any questions, feel free to start a discussion or create an issue as described above.
+When you're finished with the changes, create a pull request, also known as a PR.
+‼️ Please make all PRs against the dev branch of the gget repository.
Once you submit your PR, a gget team member will review your proposal. We may ask questions or request additional information.
+Congratulations! 🎉 The gget team thanks you. ✨
+Once your PR is merged, your contributions will be publicly visible on the gget repo.
+We as members, contributors, and leaders pledge to make participation in our +community a harassment-free experience for everyone, regardless of age, body +size, visible or invisible disability, ethnicity, sex characteristics, gender +identity and expression, level of experience, education, socio-economic status, +nationality, personal appearance, race, religion, or sexual identity +and orientation.
+We pledge to act and interact in ways that contribute to an open, welcoming, +diverse, inclusive, and healthy community.
+Examples of behavior that contributes to a positive environment for our +community include:
+Examples of unacceptable behavior include:
+Community leaders are responsible for clarifying and enforcing our standards of +acceptable behavior and will take appropriate and fair corrective action in +response to any behavior that they deem inappropriate, threatening, offensive, +or harmful.
+Community leaders have the right and responsibility to remove, edit, or reject +comments, commits, code, wiki edits, issues, and other contributions that are +not aligned to this Code of Conduct, and will communicate reasons for moderation +decisions when appropriate.
+This Code of Conduct applies within all community spaces, and also applies when +an individual is officially representing the community in public spaces. +Examples of representing our community include using an official e-mail address, +posting via an official social media account, or acting as an appointed +representative at an online or offline event.
+Instances of abusive, harassing, or otherwise unacceptable behavior may be +reported to the community leaders responsible for enforcement at +lpachter@caltech.edu. +All complaints will be reviewed and investigated promptly and fairly.
+All community leaders are obligated to respect the privacy and security of the +reporter of any incident.
+Community leaders will follow these Community Impact Guidelines in determining +the consequences for any action they deem in violation of this Code of Conduct:
+Community Impact: Use of inappropriate language or other behavior deemed +unprofessional or unwelcome in the community.
+Consequence: A private, written warning from community leaders, providing +clarity around the nature of the violation and an explanation of why the +behavior was inappropriate. A public apology may be requested.
+Community Impact: A violation through a single incident or series +of actions.
+Consequence: A warning with consequences for continued behavior. No +interaction with the people involved, including unsolicited interaction with +those enforcing the Code of Conduct, for a specified period of time. This +includes avoiding interactions in community spaces as well as external channels +like social media. Violating these terms may lead to a temporary or +permanent ban.
+Community Impact: A serious violation of community standards, including +sustained inappropriate behavior.
+Consequence: A temporary ban from any sort of interaction or public +communication with the community for a specified period of time. No public or +private interaction with the people involved, including unsolicited interaction +with those enforcing the Code of Conduct, is allowed during this period. +Violating these terms may lead to a permanent ban.
+Community Impact: Demonstrating a pattern of violation of community +standards, including sustained inappropriate behavior, harassment of an +individual, or aggression toward or disparagement of classes of individuals.
+Consequence: A permanent ban from any sort of public interaction within +the community.
+This Code of Conduct is adapted from the Contributor Covenant, +version 2.0, available at https://www.contributor-covenant.org/version/2/0/code_of_conduct.html.
+Community Impact Guidelines were inspired by Mozilla's code of conduct +enforcement ladder.
+For answers to common questions about this code of conduct, see the FAQ at +https://www.contributor-covenant.org/faq. Translations are available at +https://www.contributor-covenant.org/translations.
+ +If you use gget in a publication, please cite:
+Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
If using gget alphafold, please also cite:
And, if applicable:
+If using gget archs4, please also cite:
Lachmann A, Torre D, Keenan AB, Jagodnik KM, Lee HJ, Wang L, Silverstein MC, Ma’ayan A. Massive mining of publicly available RNA-seq data from human and mouse. Nature Communications 9. Article number: 1366 (2018), doi:10.1038/s41467-018-03751-6
+Bray NL, Pimentel H, Melsted P and Pachter L, Near optimal probabilistic RNA-seq quantification, Nature Biotechnology 34, p 525--527 (2016). https://doi.org/10.1038/nbt.3519
+If using gget bgee, please also cite:
If using gget blast, please also cite:
If using gget blat, please also cite:
If using gget cbio, please also cite:
Cerami E, Gao J, Dogrusoz U, Gross BE, Sumer SO, Aksoy BA, Jacobsen A, Byrne CJ, Heuer ML, Larsson E, Antipin Y, Reva B, Goldberg AP, Sander C, Schultz N. The cBio cancer genomics portal: an open platform for exploring multidimensional cancer genomics data. Cancer Discov. 2012 May;2(5):401-4. doi: 10.1158/2159-8290.CD-12-0095. Erratum in: Cancer Discov. 2012 Oct;2(10):960. PMID: 22588877; PMCID: PMC3956037.
+Gao J, Aksoy BA, Dogrusoz U, Dresdner G, Gross B, Sumer SO, Sun Y, Jacobsen A, Sinha R, Larsson E, Cerami E, Sander C, Schultz N. Integrative analysis of complex cancer genomics and clinical profiles using the cBioPortal. Sci Signal. 2013 Apr 2;6(269):pl1. doi: 10.1126/scisignal.2004088. PMID: 23550210; PMCID: PMC4160307.
+de Bruijn I, Kundra R, Mastrogiacomo B, Tran TN, Sikina L, Mazor T, Li X, Ochoa A, Zhao G, Lai B, Abeshouse A, Baiceanu D, Ciftci E, Dogrusoz U, Dufilie A, Erkoc Z, Garcia Lara E, Fu Z, Gross B, Haynes C, Heath A, Higgins D, Jagannathan P, Kalletla K, Kumari P, Lindsay J, Lisman A, Leenknegt B, Lukasse P, Madela D, Madupuri R, van Nierop P, Plantalech O, Quach J, Resnick AC, Rodenburg SYA, Satravada BA, Schaeffer F, Sheridan R, Singh J, Sirohi R, Sumer SO, van Hagen S, Wang A, Wilson M, Zhang H, Zhu K, Rusk N, Brown S, Lavery JA, Panageas KS, Rudolph JE, LeNoue-Newton ML, Warner JL, Guo X, Hunter-Zinck H, Yu TV, Pilai S, Nichols C, Gardos SM, Philip J; AACR Project GENIE BPC Core Team, AACR Project GENIE Consortium; Kehl KL, Riely GJ, Schrag D, Lee J, Fiandalo MV, Sweeney SM, Pugh TJ, Sander C, Cerami E, Gao J, Schultz N. Analysis and Visualization of Longitudinal Genomic and Clinical Data from the AACR Project GENIE Biopharma Collaborative in cBioPortal. Cancer Res. 2023 Dec 1;83(23):3861-3867. doi: 10.1158/0008-5472.CAN-23-0816. PMID: 37668528; PMCID: PMC10690089.
+Please also cite the source of the data if you are using a publicly available dataset.
+If using gget cellxgene, please also cite:
If using gget cosmic, please also cite:
If using gget diamond, please also cite:
If using gget elm, please also cite:
Laura Luebbert, Chi Hoang, Manjeet Kumar, Lior Pachter, Fast and scalable querying of eukaryotic linear motifs with gget elm, Bioinformatics, 2024, btae095, https://doi.org/10.1093/bioinformatics/btae095
+Manjeet Kumar, Sushama Michael, Jesús Alvarado-Valverde, Bálint Mészáros, Hugo Sámano‐Sánchez, András Zeke, Laszlo Dobson, Tamas Lazar, Mihkel Örd, Anurag Nagpal, Nazanin Farahi, Melanie Käser, Ramya Kraleti, Norman E Davey, Rita Pancsa, Lucía B Chemes, Toby J Gibson, The Eukaryotic Linear Motif resource: 2022 release, Nucleic Acids Research, Volume 50, Issue D1, 7 January 2022, Pages D497–D508, https://doi.org/10.1093/nar/gkab975
+If using gget enrichr, please also cite:
Chen EY, Tan CM, Kou Y, Duan Q, Wang Z, Meirelles GV, Clark NR, Ma'ayan A. Enrichr: interactive and collaborative HTML5 gene list enrichment analysis tool. BMC Bioinformatics. 2013; 128(14). https://doi.org/10.1186/1471-2105-14-128
+Kuleshov MV, Jones MR, Rouillard AD, Fernandez NF, Duan Q, Wang Z, Koplev S, Jenkins SL, Jagodnik KM, Lachmann A, McDermott MG, Monteiro CD, Gundersen GW, Ma'ayan A. Enrichr: a comprehensive gene set enrichment analysis web server 2016 update. Nucleic Acids Research. 2016; gkw377. doi: 10.1093/nar/gkw377
+Xie Z, Bailey A, Kuleshov MV, Clarke DJB., Evangelista JE, Jenkins SL, Lachmann A, Wojciechowicz ML, Kropiwnicki E, Jagodnik KM, Jeon M, & Ma’ayan A. Gene set knowledge discovery with Enrichr. Current Protocols, 1, e90. 2021. doi: 10.1002/cpz1.90.
+If working with non-human/mouse datasets, please also cite:
+If using gget info, please also cite:
Martin FJ, Amode MR, Aneja A, Austine-Orimoloye O, Azov AG, Barnes I, Becker A, Bennett R, Berry A, Bhai J, Bhurji SK, Bignell A, Boddu S, Branco Lins PR, Brooks L, Ramaraju SB, Charkhchi M, Cockburn A, Da Rin Fiorretto L, Davidson C, Dodiya K, Donaldson S, El Houdaigui B, El Naboulsi T, Fatima R, Giron CG, Genez T, Ghattaoraya GS, Martinez JG, Guijarro C, Hardy M, Hollis Z, Hourlier T, Hunt T, Kay M, Kaykala V, Le T, Lemos D, Marques-Coelho D, Marugán JC, Merino GA, Mirabueno LP, Mushtaq A, Hossain SN, Ogeh DN, Sakthivel MP, Parker A, Perry M, Piližota I, Prosovetskaia I, Pérez-Silva JG, Salam AIA, Saraiva-Agostinho N, Schuilenburg H, Sheppard D, Sinha S, Sipos B, Stark W, Steed E, Sukumaran R, Sumathipala D, Suner MM, Surapaneni L, Sutinen K, Szpak M, Tricomi FF, Urbina-Gómez D, Veidenberg A, Walsh TA, Walts B, Wass E, Willhoft N, Allen J, Alvarez-Jarreta J, Chakiachvili M, Flint B, Giorgetti S, Haggerty L, Ilsley GR, Loveland JE, Moore B, Mudge JM, Tate J, Thybert D, Trevanion SJ, Winterbottom A, Frankish A, Hunt SE, Ruffier M, Cunningham F, Dyer S, Finn RD, Howe KL, Harrison PW, Yates AD, Flicek P. Ensembl 2023. Nucleic Acids Res. 2023 Jan 6;51(D1):D933-D941. doi: 10.1093/nar/gkac958. PMID: 36318249; PMCID: PMC9825606.
+Sayers EW, Beck J, Bolton EE, Brister JR, Chan J, Comeau DC, Connor R, DiCuccio M, Farrell CM, Feldgarden M, Fine AM, Funk K, Hatcher E, Hoeppner M, Kane M, Kannan S, Katz KS, Kelly C, Klimke W, Kim S, Kimchi A, Landrum M, Lathrop S, Lu Z, Malheiro A, Marchler-Bauer A, Murphy TD, Phan L, Prasad AB, Pujar S, Sawyer A, Schmieder E, Schneider VA, Schoch CL, Sharma S, Thibaud-Nissen F, Trawick BW, Venkatapathi T, Wang J, Pruitt KD, Sherry ST. Database resources of the National Center for Biotechnology Information. Nucleic Acids Res. 2024 Jan 5;52(D1):D33-D43. doi: 10.1093/nar/gkad1044. PMID: 37994677; PMCID: PMC10767890.
+The UniProt Consortium , UniProt: the Universal Protein Knowledgebase in 2023, Nucleic Acids Research, Volume 51, Issue D1, 6 January 2023, Pages D523–D531, https://doi.org/10.1093/nar/gkac1052
+If using gget muscle, please also cite:
If using gget opentargets, please also cite:
If using gget pdb, please also cite:
If using gget ref or gget search, please also cite:
If using gget seq, please also cite:
Martin FJ, Amode MR, Aneja A, Austine-Orimoloye O, Azov AG, Barnes I, Becker A, Bennett R, Berry A, Bhai J, Bhurji SK, Bignell A, Boddu S, Branco Lins PR, Brooks L, Ramaraju SB, Charkhchi M, Cockburn A, Da Rin Fiorretto L, Davidson C, Dodiya K, Donaldson S, El Houdaigui B, El Naboulsi T, Fatima R, Giron CG, Genez T, Ghattaoraya GS, Martinez JG, Guijarro C, Hardy M, Hollis Z, Hourlier T, Hunt T, Kay M, Kaykala V, Le T, Lemos D, Marques-Coelho D, Marugán JC, Merino GA, Mirabueno LP, Mushtaq A, Hossain SN, Ogeh DN, Sakthivel MP, Parker A, Perry M, Piližota I, Prosovetskaia I, Pérez-Silva JG, Salam AIA, Saraiva-Agostinho N, Schuilenburg H, Sheppard D, Sinha S, Sipos B, Stark W, Steed E, Sukumaran R, Sumathipala D, Suner MM, Surapaneni L, Sutinen K, Szpak M, Tricomi FF, Urbina-Gómez D, Veidenberg A, Walsh TA, Walts B, Wass E, Willhoft N, Allen J, Alvarez-Jarreta J, Chakiachvili M, Flint B, Giorgetti S, Haggerty L, Ilsley GR, Loveland JE, Moore B, Mudge JM, Tate J, Thybert D, Trevanion SJ, Winterbottom A, Frankish A, Hunt SE, Ruffier M, Cunningham F, Dyer S, Finn RD, Howe KL, Harrison PW, Yates AD, Flicek P. Ensembl 2023. Nucleic Acids Res. 2023 Jan 6;51(D1):D933-D941. doi: 10.1093/nar/gkac958. PMID: 36318249; PMCID: PMC9825606.
+The UniProt Consortium , UniProt: the Universal Protein Knowledgebase in 2023, Nucleic Acids Research, Volume 51, Issue D1, 6 January 2023, Pages D523–D531, https://doi.org/10.1093/nar/gkac1052
+gget is only as accurate as the databases/servers/APIs it queries from. The accuracy or reliability of the data is not guaranteed or warranted in any way and the providers disclaim liability of any kind whatsoever, including, without limitation, liability for quality, performance, merchantability and fitness for a particular purpose arising out of the use, or inability to use the data.
gget es un programa gratuito de código fuente abierta de Terminal y Python que permite la consulta eficiente de bases de datos genómicas.
+
+gget consiste en un conjunto de módulos separados pero interoperables, cada uno diseñado para facilitar un tipo de consulta de base de datos en una sola línea de código.
+
+Las bases de datos consultadas por gget se actualizan continuamente, lo que a veces cambia su estructura. Los módulos gget se prueban automáticamente cada dos semanas y se actualizan para que coincidan con las nuevas estructuras de la base de datos cuando es necesario. Si encuentra algún problema, actualice a la última versión de gget usando pip install --upgrade gget. Si el problema persiste, informa el problema.
+
+
Solicitar una nueva función
+
Estos son los módulos principales de gget. Haga clic en cualquier módulo para acceder a la documentación detallada.
| gget alphafold Predecir la estructura 3D de una proteína a partir de una secuencia de aminoácidos. |
+ gget archs4 ¿Cuál es la expresión de mi gen en el tejido X? |
+ gget bgee Encontrar todos los ortólogos de un gen. |
+ gget blast Realizar un BLAST de una secuencia de nucleótidos o aminoácidos. |
+
| gget blat Encontrar la ubicación genómica de una secuencia de nucleótidos o aminoácidos. |
+ gget cbio Explorar la expresión de un gen en los cánceres especificados. |
+ gget cellxgene Obtener matrices de conteo de ARN de células individuales listas para usar para ciertos tejidos/enfermedades/etc. |
+ gget cosmic Buscar genes, mutaciones y otros factores asociados con ciertos cánceres. |
+
| gget diamond Alinear secuencias de aminoácidos a una referencia. |
+ gget elm Encontrar dominios y funciones de interacción de proteínas en una secuencia de aminoácidos. |
+ gget enrichr Verificar si una lista de genes está asociada con un tipo celular específico/ vía/ enfermedad/ etc. |
+ gget info Recuperar toda la información asociada con un ID de Ensembl. |
+
| gget muscle Alinear múltiples secuencias de nucleótidos o aminoácidos entre sí. |
+ gget mutate Mutar secuencias de nucleótidos según mutaciones específicas. |
+ gget opentargets Explorar qué enfermedades y medicamentos están asociados con un gen. |
+ gget pdb Recuperar datos de la Base de Datos de Proteínas (PDB) según un ID de PDB. |
+
| gget ref Obtener genomas de referencia de Ensembl. |
+ gget search Encontrar IDs de Ensembl asociados con la palabra de búsqueda especificada. |
+ gget seq Recuperar la secuencia de nucleótidos o aminoácidos de un gen. |
+
Si usa gget en una publicación, por favor cite*:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Lea el artículo aquí: https://doi.org/10.1093/bioinformatics/btac836
+Gracias a Victor Garcia-Ruiz y Anna Karen Orta por su ayuda con la traduccion del sitio web.
+Versión ≥ 0.28.6 (2 de junio de 2024):
+gget mutategget cosmic: Ahora puedes descargar bases de datos completas de COSMIC utilizando el argumento download_cosmicgget ref: Ahora puede obtener la ensambladura del genoma GRCh27 usando species='human_grch37'gget search: Ajusta el acceso a los datos humanos a la estructura de la versión 112 de Ensembl (corrige issue 129)Version ≥ 0.28.5 (May 29, 2024):
gget.setup("alphafold") + mutaciones de inversión en gget mutate solo invierten la cadena en lugar de también calcular la hebra complementariaVersión ≥ 0.28.4 (31 de enero de 2024):
+gget setup: soluciona el error con la ruta del archivo al ejecutar gget.setup("elm") en el sistema operativo Windows.Versión ≥ 0.28.3 (22 de enero de 2024):
+gget search y gget ref ahora también admiten hongos 🍄, protistas 🌝 y metazoos de invertebrados 🐝 🐜 🐌 🐙 (además de vertebrados y plantas)gget cosmicgget enrichr: corrige puntos de dispersión duplicados en el gráfico cuando los nombres de las rutas están duplicadosgget elm:
+gget.gget setup: utilice el argumento out para especificar un directorio en el que se descargará la base de datos ELM. Completa esta solicitud de función.gget Diamond: El comando DIAMOND ahora se ejecuta con el indicador --ignore-warnings, lo que permite secuencias de nicho, como secuencias de aminoácidos que solo contienen caracteres de nucleótidos y secuencias repetidas. Esto también es válido para las alineaciones DIAMOND realizadas dentro de gget elm.gget ref y gget search: la versión actual de Ensembl se obtiene del nuevo archivo de versión en el sitio FTP de Ensembl para evitar errores durante la carga de nuevos lanzamientos.gget search:
+--ftp) se guardan en formato de archivo txt en lugar de json.gget ref:
+list_iv_species para enumerar todas las especies de invertebrados disponibles (se puede combinar con el argumento release para obtener todas las especies disponibles de una liberación específica de Ensembl)Versión ≥ 0.28.2 (15 de noviembre de 2023):
+gget info: devuelve un mensaje de error cuando el servidor NCBI falla por un motivo distinto a un error de recuperación (esto es un error en el lado del servidor en lugar de un error con gget)BeautifulSoupgget elm: Elimina instancias de falsos positivos y verdaderos negativos de los resultados devueltos.gget elm: agrega el argumento expandVersión ≥ 0.28.0 (5 de noviembre de 2023):
+gget muscle para agregar un tutorial sobre cómo visualizar secuencias con diferentes longitudes de nombres de secuencia + ligero cambio en la visualización devuelta para que sea un poco más sólida ante diferentes nombres de secuenciagget muscle ahora también permite una lista de secuencias como entrada (como alternativa a proporcionar la ruta a un archivo FASTA)gget cellxgene (corrige error)gget seq: permite nombres de genes faltantes (correccione [https://github.com/pachterlab/gget/issues/107](https://github.com/pachterlab/gget /números/107))gget enrichr: use el argumento kegg_out y kegg_rank para crear una imagen de la vía KEGG con los genes del análisis de enriquecimiento resaltados (gracias a [este PR](https ://github.com/pachterlab/gget/pull/106) por Noriaki Sato)gget elm y gget DiamondVersión ≥ 0.27.9 (7 de agosto de 2023):
+gget enrichr: use el argumento background_list para proporcionar una lista de genes 'background'gget search ahora también busca sinónimos Ensembl (además de nombres y descripciones de genes) para obtener resultados de búsqueda más completos (gracias a Samuel Klein por la sugerencia)Versión ≥ 0.27.8 (12 de julio de 2023):
+gget search: especifique la versión de Ensembl desde la cual se obtiene la información con -r --releasegget pdb (este error se introdujo en la versión 0.27.5)Versión ≥ 0.27.7 (15 de mayo de 2023):
+gget gpt y gget cellxgene de los requisitos instalados automáticamente a gget setupgget alphafold actualizadas para compatibilidad con Python >= 3.10census_version a gget cellxgeneVersión ≥ 0.27.6 (1 de mayo de 2023) (TIRO debido a problemas con las dependencias -> reemplazada por la versión 0.27.7):
+gget search ahora también puede consultar los ID de plantas 🌱 Ensemblgget cellxgeneVersión ≥ 0.27.5 (6 de abril de 2023):
+gget search para que funcione correctamente con la nueva versión de Pandas 2.0.0 (lanzado el 3 de abril de 2023), además de versiones anteriores de Pandasgget info con nuevos banderas uniprot y ncbi que permiten desactivar los resultados de estas bases de datos de forma independiente para ahorrar tiempo de ejecución (nota: el indicador ensembl_only quedó obsoleto)-q / --quiet (para Python: verbose=False) para desactivar la información de progresoVersión ≥ 0.27.4 (19 de marzo de 2023):
+gget gpt Versión ≥ 0.27.3 (11 de marzo de 2023):
+gget info excluye los ID de PDB de forma predeterminada para aumentar la velocidad (los resultados de PDB se pueden incluir usando la marca --pdb / pdb=True).Versión ≥ 0.27.2 (1 de enero de 2023):
+gget alphafold a DeepMind's AlphaFold v2.3.0 (incluidos los nuevos argumentos multimer_for_monomer y multimer_recycles)Versión ≥ 0.27.0 (10 de diciembre de 2022):
+gget alphafold para que coincida con los cambios recientes de DeepMindVersión ≥ 0.3.13 (11 de noviembre de 2022):
+gget enrichr y gget archs4 cuando se usa con ID de EnsemblVersión ≥ 0.3.12 (10 de noviembre de 2022):
+gget info ahora también devuelve datos de localización subcelular de UniProtgget info ensembl_only devuelve solo los resultados de Ensemblgget info y gget seqVersión ≥ 0.3.11 (7 de septiembre de 2022):
+gget pdbVersión ≥ 0.3.10 (2 de septiembre de 2022):
+gget alphafold ahora también devuelve valores pLDDT para generar gráficos sin volver a ejecutar el programa (consulte también las preguntas frecuentes de gget alphafold)Versión ≥ 0.3.9 (25 de agosto de 2022):
+gget alphafoldVersión ≥ 0.3.8 (12 de agosto de 2022):
+Versión ≥ 0.3.7 (9 de agosto de 2022):
+gget ≥ 0.3.7 si usa obtener refVersión ≥ 0.3.5 (6 de agosto de 2022):
+gget alphafoldVersión ≥ 0.2.6 (7 de julio de 2022):
+gget ref ahora admite genomas de plantas! 🌱Versión ≥ 0.2.5 (30 de junio de 2022):
+gget ≥ 0.2.5 si usa alguno de los módulos que consultan datos de UniProt (gget info y gget seq).Versión ≥ 0.2.3: (26 de junio de 2022):
+[-csv][--csv]). El formato data frame/CSV sigue siendo el formato de regreso predeterminada para Python (Jupyter Lab/Google Colab) (y se puede convertir a JSON con json=True).gget ref -s human → gget ref human.gget info: [--expand] está en desuso. El módulo ahora siempre devolverá toda la información disponible.gget info, incluida la devolución de los ID de Ensembl versionados.gget info y gget seq ahora son compatibles con las IDs de WormBase y FlyBase.gget archs4 y gget enrichr con la bandera [-e][--ensembl] (ensembl=True para Python (Jupyter Lab / Google Colab)).seqtype de gget seq fue reemplazado por la bandera [-t][--translate] (translate=True/False para Python (Jupyter Lab / Google Colab)) que devolverá secuencias de nucleótidos (False) o aminoácidos (True).seqtype de gget search se renombró a id_type (aún tomando los mismos parámetros 'gene' o 'transcript').pip install --upgrade gget
+Alternativa:
+conda install -c bioconda gget
+Para uso en Jupyter Lab / Google Colab:
+import gget
+Terminal:
+# Obtenga todos los FTP de anotaciones y referencias de Homo sapiens de la última versión de Ensembl
+$ gget ref homo_sapiens
+
+# Obtenga IDs de Ensembl de genes humanos con "ace2" o "angiotensin converting enzyme 2" en su nombre/descripción
+$ gget search -s homo_sapiens 'ace2' 'angiotensin converting enzyme 2'
+
+# Busque el gen ENSG00000130234 (ACE2) y su transcripción ENST00000252519
+$ gget info ENSG00000130234 ENST00000252519
+
+# Obtenga la secuencia de aminoácidos de la transcripción canónica del gen ENSG00000130234
+$ gget seq --translate ENSG00000130234
+
+# Rápidamente encuentra la ubicación genómica de la secuencia de aminoácidos
+$ gget blat MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS
+
+# BLAST la secuencia de aminoácidos
+$ gget blast MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS
+
+# Alinee múltiples secuencias de nucleótidos o aminoácidos entre sí (también acepta la ruta al archivo FASTA)
+$ gget muscle MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS
+
+# Alinee una o más secuencias de aminoácidos con una referencia (que contiene una o más secuencias) (BLAST local) (también acepta rutas a archivos FASTA)
+$ gget diamond MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS -ref MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS
+
+# Alinea secuencias de nucleótidos o aminoácidos en un archivo FASTA
+$ gget muscle path/to/file.fa
+
+# Use Enrichr para un análisis de ontología de una lista de genes
+$ gget enrichr -db ontology ACE2 AGT AGTR1 ACE AGTRAP AGTR2 ACE3P
+
+# Obtene la expresión en tejido humano del gen ACE2
+$ gget archs4 -w tissue ACE2
+
+# Obtenga la estructura de la proteína (en formato PDB) de ACE2 (ID de PDB devuelta por gget info)
+$ gget pdb 1R42 -o 1R42.pdb
+
+# Encuentre motivos lineales eucarióticos (ELM) en una secuencia de aminoácidos
+$ gget setup elm # solo debe ejecutarse una vez
+$ gget elm -o results MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS
+
+# Obtene una matriz de recuento de scRNAseq (formato AnnData) basada en genes, tejidos y tipos de células especificados (especie predeterminada: humano)
+$ gget setup cellxgene # solo debe ejecutarse una vez
+$ gget cellxgene --gene ACE2 SLC5A1 --tissue lung --cell_type 'mucus secreting cell' -o example_adata.h5ad
+
+# Predice la estructura proteica de GFP a partir de su secuencia de aminoácidos
+$ gget setup alphafold # solo debe ejecutarse una vez
+$ gget alphafold MSKGEELFTGVVPILVELDGDVNGHKFSVSGEGEGDATYGKLTLKFICTTGKLPVPWPTLVTTFSYGVQCFSRYPDHMKQHDFFKSAMPEGYVQERTIFFKDDGNYKTRAEVKFEGDTLVNRIELKGIDFKEDGNILGHKLEYNYNSHNVYIMADKQKNGIKVNFKIRHNIEDGSVQLADHYQQNTPIGDGPVLLPDNHYLSTQSALSKDPNEKRDHMVLLEFVTAAGITHGMDELYK
+Python (Jupyter Lab / Google Colab):
+import gget
+gget.ref("homo_sapiens")
+gget.search(["ace2", "angiotensin converting enzyme 2"], "homo_sapiens")
+gget.info(["ENSG00000130234", "ENST00000252519"])
+gget.seq("ENSG00000130234", translate=True)
+gget.blat("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS")
+gget.blast("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS")
+gget.muscle(["MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS", "MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS"])
+gget.diamond("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS", reference="MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS")
+gget.enrichr(["ACE2", "AGT", "AGTR1", "ACE", "AGTRAP", "AGTR2", "ACE3P"], database="ontology", plot=True)
+gget.archs4("ACE2", which="tissue")
+gget.pdb("1R42", save=True)
+
+gget.setup("elm") # solo debe ejecutarse una vez
+ortho_df, regex_df = gget.elm("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS")
+
+gget.setup("cellxgene") # solo debe ejecutarse una vez
+gget.cellxgene(gene = ["ACE2", "SLC5A1"], tissue = "lung", cell_type = "mucus secreting cell")
+
+gget.setup("alphafold") # solo debe ejecutarse una vez
+gget.alphafold("MSKGEELFTGVVPILVELDGDVNGHKFSVSGEGEGDATYGKLTLKFICTTGKLPVPWPTLVTTFSYGVQCFSRYPDHMKQHDFFKSAMPEGYVQERTIFFKDDGNYKTRAEVKFEGDTLVNRIELKGIDFKEDGNILGHKLEYNYNSHNVYIMADKQKNGIKVNFKIRHNIEDGSVQLADHYQQNTPIGDGPVLLPDNHYLSTQSALSKDPNEKRDHMVLLEFVTAAGITHGMDELYK")
+Use a gget con R usando reticulate:
system("pip install gget")
+install.packages("reticulate")
+library(reticulate)
+gget <- import("gget")
+
+gget$ref("homo_sapiens")
+gget$search(list("ace2", "angiotensin converting enzyme 2"), "homo_sapiens")
+gget$info(list("ENSG00000130234", "ENST00000252519"))
+gget$seq("ENSG00000130234", translate=TRUE)
+gget$blat("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS")
+gget$blast("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS")
+gget$muscle(list("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS", "MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS"), out="out.afa")
+gget$diamond("MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS", reference="MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS")
+gget$enrichr(list("ACE2", "AGT", "AGTR1", "ACE", "AGTRAP", "AGTR2", "ACE3P"), database="ontology")
+gget$archs4("ACE2", which="tissue")
+gget$pdb("1R42", save=TRUE)
+++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Predice la estructura en 3D de cualquier proteína derivada de su secuencia de aminoácidos usando una versión simplificada del algoritmo AlphaFold2 de DeepMind, originalmente producido y publicado para AlphaFold Colab.
+Resultado: Predicción de la estructura (en formato PDB) y el errór de alineación (en formato json).
Antes de usar gget alphafold por primera vez:
conda install -qy conda==4.13.0 && conda install -qy -c conda-forge openmm=7.5.1openmm=7.7.0 para Python >= 3.10)conda update -qy conda para actualizar conda a la última versión.gget setup alphafold / gget.setup("alphafold") (ver también gget setup). Al ejecutar gget setup alphafold / gget.setup("alphafold") se descargará e instalará la última versión de AlphaFold2 alojada en el AlphaFold GitHub Repo. Puede volver a ejecutar este comando en cualquier momento para actualizar el software cuando hay una nueva versión de AlphaFold.Parámetro posicional
+sequence
+Secuencia de aminoácidos (str), o una lista de secuencias (gget alphafold automaticamente usa el algoritmo del multímero si múltiples secuencias son ingresadas), o una ruta a un archivo formato FASTA.
Parámetros optionales
+-mr --multimer_recycles
+El algoritmo de multímero se reciclara hasta que las predicciones dejen de cambiar, el limite de ciclos esta indicado aqui. Por defecto: 3
+Para obtener más exactitud, ajusta este limite a 20 (al costo de ejecuciones mas tardadas).
-o --out
+Ruta a la carpeta para guardar los resultados de la predicción (str). Por defecto: "./[fecha_tiempo]_gget_alphafold_prediction".
Banderas
+-mfm --multimer_for_monomer
+Usa el algoritmo de multímero para un monómero.
-r --relax
+Relaja el mejor modelo con el algoritmo AMBER.
-q --quiet
+Uso limitado para Terminal. Impide la información de progreso de ser exhibida durante la ejecución del programa.
+Para Python, usa verbose=False.
plot
+Solo para Python. plot=True provée una visualización interactiva de la predicción con el errór de alineación en 3D con py3Dmol y matplotlib (por defecto: True).
show_sidechains
+Solo para Python. show_sidechains=True incluye las cadenas laterales de proteínas en el esquema (por defecto: True).
# Predice la estructura de una proteína derivada de su secuencia de aminoácidos
+gget alphafold MAAHKGAEHHHKAAEHHEQAAKHHHAAAEHHEKGEHEQAAHHADTAYAHHKHAEEHAAQAAKHDAEHHAPKPH
+
+# Encuentra secuencias similares previamente depositadas en el PDB para análisis comparativo
+gget blast --database pdbaa MAAHKGAEHHHKAAEHHEQAAKHHHAAAEHHEKGEHEQAAHHADTAYAHHKHAEEHAAQAAKHDAEHHAPKPH
+
+# Busca los archivos PDB de estructuras similares resultantes de gget blast para comparar y obtener una medida de calidad del modelo predecido.
+gget pdb 3UQ3 -o 3UQ3.pdb
+gget pdb 2K42 -o 2K42.pdb
+# Python
+# Predice la estructura de una proteína derivada de su secuencia de aminoácidos
+gget.alphafold("MAAHKGAEHHHKAAEHHEQAAKHHHAAAEHHEKGEHEQAAHHADTAYAHHKHAEEHAAQAAKHDAEHHAPKPH")
+
+# Encuentra secuencias similares previamente depositadas en el PDB para análisis comparativo
+gget.blast("MAAHKGAEHHHKAAEHHEQAAKHHHAAAEHHEKGEHEQAAHHADTAYAHHKHAEEHAAQAAKHDAEHHAPKPH", database="pdbaa")
+
+# Busca los archivos PDB de estructuras similares resultantes de gget blast para comparar y obtener una medida de calidad del modelo predecido.
+gget.pdb("3UQ3", save=True)
+gget.pdb("2K42", save=True)
+→ gget alphafold produce la estructura predecida (en formato PDB) y el errór de alineación (en formato json) en una nueva carpeta ("./[fecha_tiempo]_gget_alphafold_prediction"). Este ejemplo demuestra como usar gget blast y gget pdb para correr un análisis comparativo. Los archivos PDB se pueden ver en 3D con RCSB 3D view, o usando programas como PyMOL o Blender. Para comparar múltiples archivos PDB, use RCSB alignment. Python también produce esquemas interactivos, los cuales se pueden generar de los archivos PDB y JSON, como es describido en gget alphafold FAQ Q4.
Si utiliza gget alphafold en una publicación, favor de citar los siguientes artículos:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Jumper, J., Evans, R., Pritzel, A. et al. Highly accurate protein structure prediction with AlphaFold. Nature 596, 583–589 (2021). https://doi.org/10.1038/s41586-021-03819-2
+Y, si corresponde:
+++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no es especificado de otra manera. Las banderas son designadas como cierto o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede obtener desde Terminal con la bandera-h--help.
Encuentra los genes más correlacionados a un gen de interés, o bién, encuentra los tejidos donde un gen se expresa usando la base de datos ARCHS4.
+Produce: Resultados en formato JSON (Terminal) o Dataframe/CSV (Python).
Parámetro posicional
+gene
+Nombre corto (símbolo del gen) del gen de interés, p. ej. STAT4.
+Alternativamente: usa la bandera --ensembl para ingresar un ID tipo Ensembl, p. ej. ENSG00000138378.
Parámetros optionales
+-w --which
+'correlation' (correlación; se usa por defecto) o 'tissue' (tejido).
+'correlation' produce una tabla que contiene los 100 genes más correlacionados con el gen de interés. La correlación de Pearson se calcula de todas las muestras y tejidos en ARCHS4.
+'tissue' produce un atlas de expresión tisular calculado de todas las muestras humanas o de ratón (según lo definido usando el parámetro --species (especies)) en ARCHS4.
-s --species
+'human' (humano; se usa por defecto) o 'mouse' (ratón).
+Define si se usan muestras humanas o de ratón de ARCHS4.
+(Solo aplica para el atlas de expresión tisular.)
-o --out
+Ruta al archivo en el que se guardarán los resultados, p. ej. ruta/al/directorio/resultados.csv (o .json). Por defecto: salida estándar (STDOUT).
+Para Python, use save=True para guardar los resultados en el directorio de trabajo actual.
Banderas
+-e --ensembl
+Usa esta bandera si gene se ingresa como ID tipo Ensembl.
-csv --csv
+Solo para Terminal. Produce los resultados en formato CSV.
+Para Python, usa json=True para obtener los resultados en formato JSON.
-q --quiet
+Solo para Terminal. Impide la información de progreso de ser exhibida durante la ejecución del programa.
+Para Python, usa verbose=False para impedir la información de progreso de ser exhibida durante la ejecución del programa.
gget archs4 ACE2
+# Python
+gget.archs4("ACE2")
+→ Produce los 100 genes más correlacionados con el gen ACE2:
+| gene_symbol | pearson_correlation |
|---|---|
| SLC5A1 | 0.579634 |
| CYP2C18 | 0.576577 |
| . . . | . . . |
gget archs4 -w tissue ACE2
+# Python
+gget.archs4("ACE2", which="tissue")
+→ Produce la expresión tisular de ACE2 (por defecto, se utilizan datos humanos):
+| id | min | q1 | median | q3 | max |
|---|---|---|---|---|---|
| System.Urogenital/Reproductive System.Kidney.RENAL CORTEX | 0.113644 | 8.274060 | 9.695840 | 10.51670 | 11.21970 |
| System.Digestive System.Intestine.INTESTINAL EPITHELIAL CELL | 0.113644 | 5.905560 | 9.570450 | 13.26470 | 13.83590 |
| . . . | . . . | . . . | . . . | . . . | . . . |
+Consulte este tutorial de Dave Tang, quien escribió un script R para crear esta visualización con los resultados de gget archs4 en formato JSON:
Si utiliza gget archs4 en una publicación, favor de citar los siguientes artículos:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Lachmann A, Torre D, Keenan AB, Jagodnik KM, Lee HJ, Wang L, Silverstein MC, Ma’ayan A. Massive mining of publicly available RNA-seq data from human and mouse. Nature Communications 9. Article number: 1366 (2018), doi:10.1038/s41467-018-03751-6
+Bray NL, Pimentel H, Melsted P and Pachter L, Near optimal probabilistic RNA-seq quantification, Nature Biotechnology 34, p 525--527 (2016). https://doi.org/10.1038/nbt.3519
+++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Las banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
BLAST una secuencia de nucleótidos o aminoácidos a cualquier base de datos BLAST.
+Produce: Resultados en formato JSON (Terminal) o Dataframe/CSV (Python).
Parámetro posicional
+sequence
+Secuencia de nucleótidos o aminoácidos, o una ruta a un archivo tipo FASTA o .txt.
Parámetros optionales
+-p --program
+'blastn', 'blastp', 'blastx', 'tblastn', o 'tblastx'.
+Por defecto: 'blastn' para secuencias de nucleótidos; 'blastp' para secuencias de aminoácidos.
-db --database
+'nt', 'nr', 'refseq_rna', 'refseq_protein', 'swissprot', 'pdbaa', o 'pdbnt'.
+Por defecto: 'nt' para secuencias de nucleótidos; 'nr' para secuencias de aminoácidos.
+Más información sobre los bases de datos BLAST
-l --limit
+Limita el número de resultados producidos. Por defecto: 50.
-e --expect
+Define el umbral de 'expect value'. Por defecto: 10.0.
-o --out
+Ruta al archivo en el que se guardarán los resultados, p. ej. ruta/al/directorio/resultados.csv (o .json). Por defecto: salida estándar (STDOUT).
+Para Python, usa save=True para guardar los resultados en el directorio de trabajo actual.
Banderas
+-lcf --low_comp_filt
+Activa el 'low complexity filter' (filtro de baja complejidad).
-mbo --megablast_off
+Desactiva el algoritmo MegaBLAST. Por defecto: MegaBLAST esta activado (solo aplicable para blastn).
-csv --csv
+Solo para Terminal. Produce los resultados en formato CSV.
+Para Python, usa json=True para producir los resultados en formato JSON.
-q --quiet
+Solo para Terminal. Impide la información de progreso de ser exhibida durante la ejecución del programa.
+Para Python, usa verbose=False para imipidir la informacion de progreso de ser exhibida durante la ejecución del programa.
wrap_text
+Solo para Python. wrap_text=True muestra los resultados con texto envuelto para facilitar la lectura (por defecto: False).
gget blast MKWMFKEDHSLEHRCVESAKIRAKYPDRVPVIVEKVSGSQIVDIDKRKYLVPSDITVAQFMWIIRKRIQLPSEKAIFLFVDKTVPQSR
+# Python
+gget.blast("MKWMFKEDHSLEHRCVESAKIRAKYPDRVPVIVEKVSGSQIVDIDKRKYLVPSDITVAQFMWIIRKRIQLPSEKAIFLFVDKTVPQSR")
+→ Produce los resultados BLAST de la secuencia de interés. gget blast automáticamente detecta esta secuencia como una secuencia de aminoácidos y, por lo tanto, establece el programa BLAST en blastp con la base de datos nr.
| Description | Scientific Name | Common Name | Taxid | Max Score | Total Score | Query Cover | ... |
|---|---|---|---|---|---|---|---|
| PREDICTED: gamma-aminobutyric acid receptor-as... | Colobus angolensis palliatus | NaN | 336983 | 180 | 180 | 100% | ... |
| . . . | . . . | . . . | . . . | . . . | . . . | . . . | ... |
BLAST desde un archivo .fa o .txt:
+gget blast fasta.fa
+# Python
+gget.blast("fasta.fa")
+→ Produce los resultados BLAST de la primera secuencia contenida en el archivo 'fasta.fa'.
+Si utiliza gget blast en una publicación, favor de citar los siguientes artículos:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ. Basic local alignment search tool. J Mol Biol. 1990 Oct 5;215(3):403-10. doi: 10.1016/S0022-2836(05)80360-2. PMID: 2231712.
+++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Encuentra la ubicación genómica de una secuencia de nucleótidos o aminoácidos usando BLAT.
+Produce: Resultados en formato JSON (Terminal) o Dataframe/CSV (Python).
Parámetro posicional
+sequence
+Secuencia de nucleótidos o aminoácidos, o una ruta a un archivo tipo FASTA o .txt.
Parámetros optionales
+-st --seqtype
+'DNA', 'protein', 'translated%20RNA', o 'translated%20DNA'.
+Por defecto: 'DNA' para secuencias de nucleótidos; 'protein' para secuencias de aminoácidos.
-a --assembly
+Ensamblaje del genoma. 'human' (hg38) (se usa por defecto), 'mouse' (mm39) (ratón), 'zebrafish' (taeGut2) (pinzón cebra),
+o cualquiera de los ensamblajes de especies disponibles aquí (use el nombre corto del ensamblado, p. ej. 'hg38').
-o --out
+Ruta al archivo en el que se guardarán los resultados, p. ej. ruta/al/directorio/resultados.csv (o .json). Por defecto: salida estándar (STDOUT).
+Para Python, usa save=True para guardar los resultados en el directorio de trabajo actual.
Banderas
+-csv --csv
+Solo para Terminal. Produce los resultados en formato CSV.
+Para Python, usa json=True para producir los resultados en formato JSON.
-q --quiet
+Solo para Terminal. Impide la información de progreso de ser exhibida durante la ejecución del programa.
+Para Python, usa verbose=False para impedir la información de progreso de ser exhibida durante la ejecución del programa.
gget blat -a taeGut2 MKWMFKEDHSLEHRCVESAKIRAKYPDRVPVIVEKVSGSQIVDIDKRKYLVPSDITVAQFMWIIRKRIQLPSEKAIFLFVDKTVPQSR
+# Python
+gget.blat("MKWMFKEDHSLEHRCVESAKIRAKYPDRVPVIVEKVSGSQIVDIDKRKYLVPSDITVAQFMWIIRKRIQLPSEKAIFLFVDKTVPQSR", assembly="taeGut2")
+→ Produce los resultados de BLAT para el ensamblaje taeGut2 (pinzón cebra). En este ejemplo, gget blat automáticamente detecta esta secuencia como una secuencia de aminoácidos y, por lo tanto, establece el tipo de secuencia (--seqtype) como proteína.
| genome | query_size | aligned_start | aligned_end | matches | mismatches | %_aligned | ... |
|---|---|---|---|---|---|---|---|
| taeGut2 | 88 | 12 | 88 | 77 | 0 | 87.5 | ... |
Si utiliza gget blat en una publicación, favor de citar los siguientes artículos:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Kent WJ. BLAT--the BLAST-like alignment tool. Genome Res. 2002 Apr;12(4):656-64. doi: 10.1101/gr.229202. PMID: 11932250; PMCID: PMC187518.
+++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Las banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Query data de la base de datos CZ CELLxGENE Discover usando CZ CELLxGENE Discover Census.
+Produce: Un objeto AnnData que contiene la matriz de recuentos de genes y los metadatos de resultados de single cell RNA-seq de los tejidos/genes/etcetera previamente definidos.
Antes de usar gget cellxgene por primera vez, corre gget setup cellxgene / gget.setup("cellxgene") (ver también gget setup).
Parámetros opcionales
+-s --species
+'homo_sapiens' o 'mus_musculus'. Por defecto: 'homo_sapiens'.
-g --gene
+Str o lista de genes de interés o ID(s) tipo Ensembl. Por defecto: None (ninguno).
+Atención: Utilice la bandera -e / --ensembl (Python: ensembl=True) cuando ingrese ID(s) tipo Ensembl.
+Ver https://cellxgene.cziscience.com/gene-expression para ejemplos de genes.
-cv --census_version
+Versión del CZ CELLxGENE Discover Census (str), p. ej. "2023-05-15", o "latest" (ultima) o "stable" (estable). Por defecto: "stable" (estable).
-cn --column_names
+Lista de columnas de metadatos a obtener (almacenadas en AnnData.obs).
+Por defecto: ['dataset_id', 'assay', 'suspension_type', 'sex', 'tissue_general', 'tissue', 'cell_type']
+Para más opciones, ver: https://api.cellxgene.cziscience.com/curation/ui/#/ -> 'Schemas' -> 'dataset'
-o --out
+Ruta al archivo para guardar el objeto AnnData formato .h5ad (o .csv con bandera -mo / --meta_only).
+¡Requerido cuando se usa desde Terminal!
Banderas
+-e --ensembl
+Usa esta bandera si gene se ingresa como ID tipo Ensembl.
-mo --meta_only
+Solo produce la tabla (Dataframe) con metadatos (corresponde a AnnData.obs).
-q --quiet
+Solo para Terminal. Impide la información de progreso de ser exhibida durante la ejecución del programa.
+Para Python, usa verbose=False para impedir la información de progreso de ser exhibida durante la ejecución del programa.
Parámetros opcionales correspondientes a los atributos de metadatos de CZ CELLxGENE Discover
+--tissue
+Str o lista de tejido(s), p. ej. ['lung', 'blood']. Por defecto: None.
+Ver https://cellxgene.cziscience.com/gene-expression para ejemplos de tejidos.
--cell_type
+Str o lista de tipo(s) de célula(s), p. ej. ['mucus secreting cell', 'neuroendocrine cell']. Por defecto: None.
+Ver https://cellxgene.cziscience.com/gene-expression y seleccione un tejido para ejemplos de tipos de células.
--development_stage
+Str o lista de etapa(s) de desarrollo. Por defecto: None.
--disease
+Str o lista de enfermedad(es). Por defecto: None.
--sex
+Str o lista de sexo(s), p. ej. 'female' (femenina). Por defecto: None.
--dataset_id
+Str o lista de CELLxGENE ID(s). Por defecto: None.
--tissue_general_ontology_term_id
+Str o lista de tejido(s) del tipo high-level UBERON ID. Por defecto: None.
+Tejidos y sus IDs tipo UBERON se enumeran aquí.
--tissue_general
+Str o lista de tejido(s) del tipo high-level. Por defecto: None.
+Tejidos y sus IDs de UBERON se enumeran aquí.
--tissue_ontology_term_id
+Str o lista de ID(s) de 'tissue ontology term' como están definidos en el esquema de datos del CELLxGENE. Por defecto: None.
--assay_ontology_term_id
+Str o lista de ID(s) de 'assay ontology term' como están definidos en el esquema de datos del CELLxGENE. Por defecto: None.
--assay
+Str o lista de 'assays' (métodos) como están definidos en el esquema de datos del CELLxGENE. Por defecto: None.
--cell_type_ontology_term_id
+Str o lista de ID(s) de 'celltype ontology term' como están definidos en el esquema de datos del CELLxGENE. Por defecto: None.
--development_stage_ontology_term_id
+Str o lista de ID(s) de 'development stage ontology term' como están definidos en el esquema de datos del CELLxGENE. Por defecto: None.
--disease_ontology_term_id
+Str o lista de ID(s) de 'disease ontology term' como están definidos en el esquema de datos del CELLxGENE. Por defecto: None.
--donor_id
+Str o lista de ID(s) de 'donor' (donador) como están definidos en el esquema de datos del CELLxGENE. Por defecto: None.
--self_reported_ethnicity_ontology_term_id
+Str o lista de ID(s) de 'self-reported ethnicity ontology' como están definidos en el esquema de datos del CELLxGENE. Por defecto: None.
--self_reported_ethnicity
+Str o lista de etnias autoinformadas como están definidas en el esquema de datos del CELLxGENE. Por defecto: None.
--sex_ontology_term_id
+Str o lista de ID(s) de 'sex ontology' como están definidos en el esquema de datos del CELLxGENE. Por defecto: None.
--suspension_type
+Str o lista de tipo(s) de suspensión como están definidos en el esquema de datos del CELLxGENE. Por defecto: None.
gget cellxgene --gene ACE2 ABCA1 SLC5A1 --tissue lung --cell_type 'mucus secreting cell' 'neuroendocrine cell' -o example_adata.h5ad
+# Python
+adata = gget.cellxgene(
+ gene = ["ACE2", "ABCA1", "SLC5A1"],
+ tissue = "lung",
+ cell_type = ["mucus secreting cell", "neuroendocrine cell"]
+)
+adata
+→ Produce un objeto AnnData que contiene la matriz de recuentos de scRNAseq de los genes ACE2, ABCA1 y SLC5A1 en 3322 células secretoras de mucosidad y neuroendocrinas pulmonares humanas y sus metadatos correspondientes.
+Obtiene solo los metadatos (corresponde a AnnData.obs):
+gget cellxgene --meta_only --gene ENSMUSG00000015405 --ensembl --tissue lung --species mus_musculus -o example_meta.csv
+# Python
+df = gget.cellxgene(
+ meta_only = True,
+ gene = "ENSMUSG00000015405",
+ ensembl = True,
+ tissue = "lung",
+ species = "mus_musculus"
+)
+df
+→ Produce solo los metadatos de los conjuntos de datos de ENSMUSG00000015405 (ACE2), los cuales corresponden a células pulmonares murinas.
+Ver también: https://chanzuckerberg.github.io/cellxgene-census/notebooks/api_demo/census_gget_demo.html
+Si utiliza gget cellxgene en una publicación, favor de citar los siguientes artículos:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Chanzuckerberg Initiative. (n.d.). CZ CELLxGENE Discover. Retrieved [insert date here], from https://cellxgene.cziscience.com/
+++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Las banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Busque genes, mutaciones, etc. asociados con cánceres utilizando la base de datos COSMIC (Catálogo de mutaciones somáticas en cáncer).
+Produce: JSON (línea de comandos) o marco de datos/CSV (Python) cuando download_cosmic=False. Cuando download_cosmic=True, descarga la base de datos solicitada en la carpeta especificada.
Este módulo fue escrito en parte por @AubakirovArman (consulta de información) y @josephrich98 (descarga de base de datos).
+NOTA: Se aplican tarifas de licencia para el uso comercial de COSMIC. Puede leer más sobre la concesión de licencias de datos COSMIC aquí.
+Parámetro posicional (para consultar información)
+searchterm
+Término de búsqueda, que puede ser una mutación, un nombre de gen (o ID de Ensembl), una muestra, etc.
+Ejemplos para los argumentos de searchterm y entidad:
| searchterm | entidad | |
|---|---|---|
| EGFR | mutaciones | -> Encuentra mutaciones en el gen EGFR asociadas con el cáncer |
| v600e | mutaciones | -> Encuentra genes para los cuales una mutación v600e está asociada con el cáncer |
| COSV57014428 | mutaciones | -> Encuentra mutaciones asociadas con esta ID de mutaciones COSMIC |
| EGFR | genes | -> Obtiene el número de muestras, mutaciones simples/codificantes y fusiones observadas en COSMIC para EGFR |
| prostate | cáncer | -> Obtiene el número de muestras probadas y mutaciones para el cáncer de próstata |
| prostate | sitio_tumoral | -> Obtiene el número de muestras probadas, genes, mutaciones, fusiones, etc. con 'próstata' como sitio de tejido primario |
| ICGC | estudios | -> Obtiene el código de proyecto y descripciones de todos los estudios de ICGC (Consortio Internacional del Genoma del Cáncer) |
| EGFR | pubmed | -> Encuentra publicaciones de PubMed sobre EGFR y cáncer |
| ICGC | muestras | -> Obtiene metadatos sobre todas las muestras de ICGC (Consortio Internacional del Genoma del Cáncer) |
| COSS2907494 | muestras | -> Obtiene metadatos sobre esta ID de muestra COSMIC (tipo de cáncer, tejido, # genes analizados, # mutaciones, etc.) |
NOTA: (Solo Python) Establezca en None cuando se descarguen bases de datos COSMIC con download_cosmic=True.
Parámetros opcionales (para consultar información)
+-e --entity
+'mutations' (predeterminado), 'genes', 'cáncer', 'sitio_tumoral', 'estudios', 'pubmed' o 'muestras'.
+Define el tipo de resultados a devolver.
-l --limit
+Limita el número de resultados a devolver. Predeterminado: 100.
Banderas (para descargar bases de datos COSMIC)
+-d --download_cosmic
+Conmuta al modo de descarga de base de datos.
-gm --gget_mutate
+DESACTIVA la creación de una versión modificada de la base de datos para usar con gget mutate.
+Python: gget_mutate es Verdadero por defecto. Establezca gget_mutate=False para deshabilitar.
Parámetros opcionales (para descargar bases de datos COSMIC)
+-mc --mutation_class
+'cancer' (predeterminado), 'cell_line', 'census', 'resistance', 'genome_screen', 'targeted_screen', o 'cancer_example'
+Tipo de base de datos COSMIC para descargar.
-cv --cosmic_version
+Versión de la base de datos COSMIC. Predeterminado: Ninguno -> Se establece en la última versión por defecto.
-gv --grch_version
+Versión del genoma de referencia humano GRCh en el que se basó la base de datos COSMIC (37 o 38). Predeterminado: 37
Parámetros opcionales (generales)
+-o --out
+Ruta al archivo (o carpeta cuando se descargan bases de datos con el flag download_cosmic) donde se guardarán los resultados, p. ej. 'ruta/a/resultados.json'.
+Predeterminado: None
+-> Cuando download_cosmic=False: Los resultados se devolverán a la salida estándar
+-> Cuando download_cosmic=True: La base de datos se descargará en el directorio de trabajo actual
Banderas (generales)
+-csv --csv
+Solo para Terminal. Produce los resultados en formato CSV.
+Para Python, usa json=True para producir los resultados en formato JSON.
-q --quiet
+Solo para Terminal. Impide la información de progreso de ser exhibida durante la ejecución del programa.
+Para Python, usa verbose=False para imipidir la informacion de progreso de ser exhibida durante la ejecución del programa.
gget cosmic -e genes EGFR
+# Python
+gget.cosmic("EGFR", entity="genes")
+→ Devuelve mutaciones en el gen EGFR asociadas con el cáncer en el formato:
+| Gene | Syntax | Alternate IDs | Canonical |
|---|---|---|---|
| EGFR | c.*2446A>G | EGFR c.*2446A>G, EGFR p.?, ... | y |
| EGFR | c.(2185_2283)ins(18) | EGFR c.(2185_2283)ins(18), EGFR p.?, ... | y |
| . . . | . . . | . . . | . . . |
gget cosmic --download_cosmic
+# Python
+gget.cosmic(searchterm=None, download_cosmic=True)
+→ Descargue la base de datos sobre cáncer de COSMIC de la última versión de COSMIC.
+Si utiliza gget alphafold en una publicación, favor de citar los siguientes artículos:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Tate JG, Bamford S, Jubb HC, Sondka Z, Beare DM, Bindal N, Boutselakis H, Cole CG, Creatore C, Dawson E, Fish P, Harsha B, Hathaway C, Jupe SC, Kok CY, Noble K, Ponting L, Ramshaw CC, Rye CE, Speedy HE, Stefancsik R, Thompson SL, Wang S, Ward S, Campbell PJ, Forbes SA. COSMIC: the Catalogue Of Somatic Mutations In Cancer. Nucleic Acids Res. 2019 Jan 8;47(D1):D941-D947. doi: 10.1093/nar/gky1015. PMID: 30371878; PMCID: PMC6323903.
+++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Las banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Alinee múltiples proteínas o secuencias de ADN traducidas usando DIAMOND (DIAMOND es similar a BLAST, pero este es un cálculo local).
+Produce: Resultados en formato JSON (Terminal) o Dataframe/CSV (Python).
Parámetro posicional
+query
+Secuencia(s) (str o lista) de aminoácidos, o una ruta a un archivo tipo FASTA.
Parámetro requerido
+-ref --reference
+Secuencias de aminoácidos de referencia (str o lista), o una ruta a un archivo tipo FASTA.
Parámetros optionales
+-db --diamond_db
+Ruta para guardar la base de datos DIAMOND creada a partir de reference (str).
+Por defecto: None -> El archivo de base de datos DIAMOND temporal se eliminará después de la alineación o se guardará en out si se proporciona out.
-s --sensitivity
+Sensibilidad de la alineación (str). Por defecto: "very-sensitive" (muy sensible).
+Uno de los siguientes: fast, mid-sensitive, sensitive, more-sensitive, very-sensitive, or ultra-sensitive.
-t --threads
+Número de hilos de procesamiento utilizados (int). Por defecto: 1.
-db --diamond_binary
+Ruta al binario DIAMOND (str). Por defecto: None -> Utiliza el binario DIAMOND instalado automáticamente con gget.
-o --out
+Ruta al archivo en el que se guardarán los resultados (str), p. ej. "ruta/al/directorio". Por defecto: salida estándar (STDOUT); los archivos temporales se eliminan.
Banderas
+-u --uniprot
+Use esta bandera cuando sequence es un ID de Uniprot en lugar de una secuencia de aminoácidos.
-csv --csv
+Solo para Terminal. Produce los resultados en formato CSV.
+Para Python, usa json=True para producir los resultados en formato JSON.
-q --quiet
+Solo para Terminal. Impide la información de progreso de ser exhibida durante la ejecución del programa.
+Para Python, usa verbose=False para impedir la información de progreso de ser exhibida durante la ejecución del programa.
# !!! Asegúrese de enumerar primero el argumento posicional aquí para que no se agregue como secuencia de referencia
+gget diamond GGETISAWESQME ELVISISALIVE LQVEFRANKLIN PACHTERLABRQCKS -ref GGETISAWESQMEELVISISALIVELQVEFRANKLIN PACHTERLABRQCKS
+# Python
+gget.diamond(["GGETISAWESQME", "ELVISISALIVE", "LQVEFRANKLIN", "PACHTERLABRQCKS"], reference=["GGETISAWESQMEELVISISALIVELQVEFRANKLIN", "PACHTERLABRQCKS"])
+→ Produce los resultados de la alineación en formato JSON (Terminal) o Dataframe/CSV:
+| query_accession | subject_accession | identity_percentage | query_seq_length | subject_seq_length | length | mismatches | gap_openings | query_start | query_end | subject_start | subject_end | e-value | bit_score |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Seq0 | Seq0 | 100 | 13 | 37 | 13 | 0 | 0 | 1 | 13 | 1 | 13 | 2.82e-09 | 30.8 |
| Seq2 | Seq0 | 100 | 12 | 37 | 12 | 0 | 0 | 1 | 12 | 26 | 37 | 4.35e-08 | 27.7 |
| Seq3 | Seq1 | 100 | 15 | 15 | 15 | 0 | 0 | 1 | 15 | 1 | 15 | 2.01e-11 | 36.2 |
Si utiliza gget diamond en una publicación, favor de citar los siguientes artículos:
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
+Buchfink, B., Xie, C. & Huson, D. Fast and sensitive protein alignment using DIAMOND. Nat Methods 12, 59–60 (2015). https://doi.org/10.1038/nmeth.3176
+++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Prediga localmente motivos lineales eucarióticos (ELMs) a partir de una secuencia de aminoácidos o UniProt Acc utilizando datos de la base de datos ELM.
+Produce: Resultados en formato JSON (Terminal) o Dataframe/CSV (Python). Este módulo devuelve dos tipos de resultados (ver ejemplos).
Los datos de ELM se pueden descargar y distribuir para uso no comercial de acuerdo con el acuerdo de licencia de software de ELM.
+Si utiliza gget elm en una publicación, favor de citar:
Antes de usar gget elm por primera vez, ejecute gget setup elm / gget.setup("elm") una vez (consulte también gget setup).
Parámetro posicional
+sequence
+Secuencia de aminoácidos o Uniprot Acc (str).
+Al proporcionar una Uniprot Acc, use la bandera --uniprot (Python: uniprot=True).
Parámetros optionales
+-s sensitivity
+Sensibilidad de la alineación DIAMOND (str). Por defecto: "very-sensitive" (muy sensible).
+Uno de los siguientes: fast, mid-sensitive, sensitive, more-sensitive, very-sensitive, or ultra-sensitive.
-t threads
+Número de hilos de procesamiento utilizados en la alineación de secuencias con DIAMOND (int). Por defecto: 1.
-bin diamond_binary
+Ruta al binario DIAMOND (str). Por defecto: None -> Utiliza el binario DIAMOND instalado automáticamente con gget.
-o --out
+Ruta al archivo en el que se guardarán los resultados (str), p. ej. "ruta/al/directorio". Por defecto: salida estándar (STDOUT); los archivos temporales se eliminan.
Banderas
+-u --uniprot
+Use esta bandera cuando sequence es una Uniprot Acc en lugar de una secuencia de aminoácidos.
-e --expand
+Amplíe la información devuelta en el marco de datos de expresiones regulares para incluir los nombres de proteínas, los organismos y las referencias en las que se validó originalmente el motivo.
-csv --csv
+Solo para Terminal. Produce los resultados en formato CSV.
+Para Python, usa json=True para producir los resultados en formato JSON.
-q --quiet
+Solo para Terminal. Impide la información de progreso de ser exhibida durante la ejecución del programa.
+Para Python, usa verbose=False para impedir la información de progreso de ser exhibida durante la ejecución del programa.
Encuentre ELM en una secuencia de aminoácidos:
+gget setup elm # Descarga/actualiza la base de datos ELM local
+gget elm -o gget_elm_results LIAQSIGQASFV
+# Python
+gget.setup(“elm”) # Descarga/actualiza la base de datos ELM local
+ortholog_df, regex_df = gget.elm("LIAQSIGQASFV")
+Encuentre ELM que proporcionen a una UniProt Acc:
+gget setup elm # Descarga/actualiza la base de datos ELM local
+gget elm -o gget_elm_results --uniprot Q02410 -e
+# Python
+gget.setup(“elm”) # Descarga/actualiza la base de datos ELM local
+ortholog_df, regex_df = gget.elm("Q02410", uniprot=True, expand=True)
+→ Produce dos resultados con información extensa sobre ELMs asociados con proteínas ortólogas y motivos encontrados en la secuencia de entrada directamente en función de sus expresiones regex:
+ortholog_df:
+| Ortholog_UniProt_Acc | ProteinName | class_accession | ELMIdentifier | FunctionalSiteName | Description | Organism | … |
|---|---|---|---|---|---|---|---|
| Q02410 | APBA1_HUMAN | ELME000357 | LIG_CaMK_CASK_1 | CASK CaMK domain binding ligand motif | Motif that mediates binding to the calmodulin-dependent protein kinase (CaMK) domain of the peripheral plasma membrane protein CASK/Lin2. | Homo sapiens | … |
| Q02410 | APBA1_HUMAN | ELME000091 | LIG_PDZ_Class_2 | PDZ domain ligands | The C-terminal class 2 PDZ-binding motif is classically represented by a pattern such as | Homo sapiens | … |
regex_df:
+| Instance_accession | ELMIdentifier | FunctionalSiteName | ELMType | Description | Instances (Matched Sequence) | Organism | … |
|---|---|---|---|---|---|---|---|
| ELME000321 | CLV_C14_Caspase3-7 | Caspase cleavage motif | CLV | Caspase-3 and Caspase-7 cleavage site. | ERSDG | Mus musculus | … |
| ELME000102 | CLV_NRD_NRD_1 | NRD cleavage site | CLV | N-Arg dibasic convertase (NRD/Nardilysin) cleavage site. | RRA | Rattus norvegicus | … |
| ELME000100 | CLV_PCSK_PC1ET2_1 | PCSK cleavage site | CLV | NEC1/NEC2 cleavage site. | KRD | Mus musculus | … |
| ELME000146 | CLV_PCSK_SKI1_1 | PCSK cleavage site | CLV | Subtilisin/kexin isozyme-1 (SKI1) cleavage site. | RLLTA | Homo sapiens | … |
| ELME000231 | DEG_APCC_DBOX_1 | APCC-binding Destruction motifs | DEG | An RxxL-based motif that binds to the Cdh1 and Cdc20 components of APC/C thereby targeting the protein for destruction in a cell cycle dependent manner | SRVKLNIVR | Saccharomyces cerevisiae S288c | … |
| … | … | … | … | … | … | … | … |
(Los motivos que aparecen en muchas especies diferentes pueden parecer repetidos, pero todas las filas deben ser únicas.)
+++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Las banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Realice un análisis de enriquecimiento de una lista de genes utilizando Enrichr.
+Produce: Resultados en formato JSON (Terminal) o Dataframe/CSV (Python).
Parámetro posicional
+genes
+Lista de nombres cortos (símbolos) de los genes de interés para realizar el análisis de enriquecimiento, p. PHF14 RBM3 MSL1 PHF21A.
+Alternativamente: use la bandera --ensembl para ingresar IDs tipo Ensembl, p. ENSG00000106443 ENSG00000102317 ENSG00000188895.
Otros parámetros requeridos
+-db --database
+Base de datos que será utilizada como referencia para el análisis de enriquecimiento.
+Admite cualquier base de datos enumerada aquí o uno de los siguientes accesos directos:
+'pathway' (KEGG_2021_Human)
+'transcription' (ChEA_2016)
+'ontology' (GO_Biological_Process_2021)
+'diseases_drugs' (GWAS_Catalog_2019)
+'celltypes' (PanglaoDB_Augmented_2021)
+'kinase_interactions' (KEA_2015)
Parámetros opcionales
+-bkg_l --background_list
+Lista de nombres cortos (símbolos) de genes de 'background' (de fondo/control), p. NSUN3 POLRMT NLRX1.
+Alternativamente: usa la bandera --ensembl_background para ingresar IDs tipo Ensembl.
-o --out
+Ruta al archivo en el que se guardarán los resultados, p. ruta/al/directorio/resultados.csv (o .json). Por defecto: salida estándar (STDOUT).
+Para Python, usa save=True para guardar los resultados en el directorio de trabajo actual.
-ko --kegg_out
+Ruta al archivo png en el que se guardará la imágen de la vía de señalización celular KEGG, p. ej. ruta/al/directorio/KEGG.png. (Por defecto: None)
-kr --kegg_rank
+Rango de la ruta KEGG que se va a trazar. (Por defecto: 1)
figsize
+Solo para Python. (ancho, alto) de la visualización en pulgadas. (Por defecto: (10,10))
ax
+Solo para Python. Ingresa un objeto de ejes matplotlib para personalizar la visualización.(Por defecto: None)
Banderas
+-e --ensembl
+Usa esta bandera si genes se ingresa como una lista de IDs tipo Ensembl.
-e_b --ensembl_background
+Usa esta bandera si background_list se ingresa como una lista de IDs tipo Ensembl.
-bkg --background
+Use un conjunto de 20,625 genes 'background'
+listados aquí.
-csv --csv
+Solo para Terminal. Produce los resultados en formato CSV.
+Para Python, usa json=True produce los resultados en formato JSON.
-q --quiet
+Solo para Terminal. Impide la información de progreso de ser exhibida durante la ejecución del programa.
+Para Python, usa verbose=False para imipidir la información de progreso de ser exhibida durante la ejecución del programa.
plot
+Solo para Python. plot=True provée la visualización de los primeros 15 resultados (por defecto: False).
gget enrichr -db ontology ACE2 AGT AGTR1
+# Python
+gget.enrichr(["ACE2", "AGT", "AGTR1"], database="ontology", plot=True)
+→ Produce vías/funciones celulares relacionadas con los genes ACE2, AGT y AGTR1 de la base de datos GO Biological Process 2021. En Python, plot=True provee la visualización de resultados:

Use gget enrichr con una lista de genes 'background':
# Aquí, primero ingresamos los genes de interés (parámetro posicional 'genes'), para que no se agreguen a la lista de genes 'background' detrás del parámetro '-bkgr_l'
+gget enrichr \
+ PHF14 RBM3 MSL1 PHF21A ARL10 INSR JADE2 P2RX7 LINC00662 CCDC101 PPM1B KANSL1L CRYZL1 ANAPC16 TMCC1 CDH8 RBM11 CNPY2 HSPA1L CUL2 PLBD2 LARP7 TECPR2 ZNF302 CUX1 MOB2 CYTH2 SEC22C EIF4E3 ROBO2 ADAMTS9-AS2 CXXC1 LINC01314 ATF7 ATP5F1 \
+ -db ChEA_2022 \
+ -bkg_l NSUN3 POLRMT NLRX1 SFXN5 ZC3H12C SLC25A39 ARSG DEFB29 PCMTD2 ACAA1A LRRC1 2810432D09RIK SEPHS2 SAC3D1 TMLHE LOC623451 TSR2 PLEKHA7 GYS2 ARHGEF12 HIBCH LYRM2 ZBTB44 ENTPD5 RAB11FIP2 LIPT1 INTU ANXA13 KLF12 SAT2 GAL3ST2 VAMP8 FKBPL AQP11 TRAP1 PMPCB TM7SF3 RBM39 BRI3 KDR ZFP748 NAP1L1 DHRS1 LRRC56 WDR20A STXBP2 KLF1 UFC1 CCDC16 9230114K14RIK RWDD3 2610528K11RIK ACO1 CABLES1 LOC100047214 YARS2 LYPLA1 KALRN GYK ZFP787 ZFP655 RABEPK ZFP650 4732466D17RIK EXOSC4 WDR42A GPHN 2610528J11RIK 1110003E01RIK MDH1 1200014M14RIK AW209491 MUT 1700123L14RIK 2610036D13RIK PHF14 RBM3 MSL1 PHF21A ARL10 INSR JADE2 P2RX7 LINC00662 CCDC101 PPM1B KANSL1L CRYZL1 ANAPC16 TMCC1 CDH8 RBM11 CNPY2 HSPA1L CUL2 PLBD2 LARP7 TECPR2 ZNF302 CUX1 MOB2 CYTH2 SEC22C EIF4E3 ROBO2 ADAMTS9-AS2 CXXC1 LINC01314 ATF7 ATP5F1COX15 TMEM30A NSMCE4A TM2D2 RHBDD3 ATXN2 NFS1 3110001I20RIK BC038156 C330002I19RIK ZFYVE20 POLI TOMM70A LOC100047782 2410012H22RIK RILP A230062G08RIK PTTG1IP RAB1 AFAP1L1 LYRM5 2310026E23RIK SLC7A6OS MAT2B 4932438A13RIK LRRC8A SMO NUPL2
+# Python
+gget.enrichr(
+ genes = [
+ "PHF14", "RBM3", "MSL1", "PHF21A", "ARL10", "INSR", "JADE2", "P2RX7",
+ "LINC00662", "CCDC101", "PPM1B", "KANSL1L", "CRYZL1", "ANAPC16", "TMCC1",
+ "CDH8", "RBM11", "CNPY2", "HSPA1L", "CUL2", "PLBD2", "LARP7", "TECPR2",
+ "ZNF302", "CUX1", "MOB2", "CYTH2", "SEC22C", "EIF4E3", "ROBO2",
+ "ADAMTS9-AS2", "CXXC1", "LINC01314", "ATF7", "ATP5F1"
+ ],
+ database = "ChEA_2022",
+ background_list = [
+ "NSUN3","POLRMT","NLRX1","SFXN5","ZC3H12C","SLC25A39","ARSG",
+ "DEFB29","PCMTD2","ACAA1A","LRRC1","2810432D09RIK","SEPHS2",
+ "SAC3D1","TMLHE","LOC623451","TSR2","PLEKHA7","GYS2","ARHGEF12",
+ "HIBCH","LYRM2","ZBTB44","ENTPD5","RAB11FIP2","LIPT1",
+ "INTU","ANXA13","KLF12","SAT2","GAL3ST2","VAMP8","FKBPL",
+ "AQP11","TRAP1","PMPCB","TM7SF3","RBM39","BRI3","KDR","ZFP748",
+ "NAP1L1","DHRS1","LRRC56","WDR20A","STXBP2","KLF1","UFC1",
+ "CCDC16","9230114K14RIK","RWDD3","2610528K11RIK","ACO1",
+ "CABLES1", "LOC100047214","YARS2","LYPLA1","KALRN","GYK",
+ "ZFP787","ZFP655","RABEPK","ZFP650","4732466D17RIK","EXOSC4",
+ "WDR42A","GPHN","2610528J11RIK","1110003E01RIK","MDH1","1200014M14RIK",
+ "AW209491","MUT","1700123L14RIK","2610036D13RIK",
+ "PHF14", "RBM3", "MSL1", "PHF21A", "ARL10", "INSR", "JADE2",
+ "P2RX7", "LINC00662", "CCDC101", "PPM1B", "KANSL1L", "CRYZL1",
+ "ANAPC16", "TMCC1","CDH8", "RBM11", "CNPY2", "HSPA1L", "CUL2",
+ "PLBD2", "LARP7", "TECPR2", "ZNF302", "CUX1", "MOB2", "CYTH2",
+ "SEC22C", "EIF4E3", "ROBO2", "ADAMTS9-AS2", "CXXC1", "LINC01314", "ATF7",
+ "ATP5F1""COX15","TMEM30A","NSMCE4A","TM2D2","RHBDD3","ATXN2","NFS1",
+ "3110001I20RIK","BC038156","C330002I19RIK","ZFYVE20","POLI","TOMM70A",
+ "LOC100047782","2410012H22RIK","RILP","A230062G08RIK",
+ "PTTG1IP","RAB1","AFAP1L1", "LYRM5","2310026E23RIK",
+ "SLC7A6OS","MAT2B","4932438A13RIK","LRRC8A","SMO","NUPL2"
+ ],
+ plot=True
+)
+→ Provée factores de transcripción relacionados a los genes de interés y controlados con la lista de genes background de la base de datos ChEA 2022. En Python, plot=True permite la visualización de resultados:

Genere una imagen de la vía de señalización de células KEGG con los genes del análisis de enriquecimiento resaltados:
+Esta función está disponible gracias a un PR de Noriaki Sato.
gget enrichr -db pathway --kegg_out kegg.png --kegg_rank 1 ZBP1 IRF3 RIPK1
+# Python
+gget.enrichr(["ZBP1", "IRF3", "RIPK1"], database="pathway", kegg_out="kegg.png", kegg_rank=1)
+→ Además de los resultados estándar gget enrichr, el argumento kegg_out guarda una imagen con los genes del análisis de enriquecimiento resaltados guardado como kegg.png:
El siguiente ejemplo fue enviado por Dylan Lawless a través de un PR (con ajustes de Laura Luebbert):
+Use gget enrichr en R y cree unq visualización similar usando ggplot.
+TENGA EN CUENTA el cambio de ejes en comparación con la visualización en Python.
system("pip install gget")
+install.packages("reticulate")
+library(reticulate)
+gget <- import("gget")
+
+# Perform enrichment analysis on a list of genes
+df <- gget$enrichr(list("ACE2", "AGT", "AGTR1"), database = "ontology")
+
+# Count number of overlapping genes
+df$overlapping_genes_count <- lapply(df$overlapping_genes, length) |> as.numeric()
+
+# Only keep the top 15 results
+df <- df[1:15, ]
+
+# Plot
+library(ggplot2)
+
+df |>
+ ggplot() +
+ geom_bar(aes(
+ x = -log10(adj_p_val),
+ y = reorder(path_name, -adj_p_val)
+ ),
+ stat = "identity",
+ fill = "lightgrey",
+ width = 0.5,
+ color = "black") +
+ geom_text(
+ aes(
+ y = path_name,
+ x = (-log10(adj_p_val)),
+ label = overlapping_genes_count
+ ),
+ nudge_x = 0.75,
+ show.legend = NA,
+ color = "red"
+ ) +
+ geom_text(
+ aes(
+ y = Inf,
+ x = Inf,
+ hjust = 1,
+ vjust = 1,
+ label = "# of overlapping genes"
+ ),
+ show.legend = NA,
+ color = "red"
+ ) +
+ geom_vline(linetype = "dotted", linewidth = 1, xintercept = -log10(0.05)) +
+ ylab("Pathway name") +
+ xlab("-log10(adjusted P value)")
+++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Genera texto en lenguaje natural basado en mensaje de entrada. gget gpt use la API 'openai.ChatCompletion.create' de OpenAI.
+Este módulo, incluido su código, documentación y pruebas unitarias, fue escrito en parte por Chat-GTP3 de OpenAI.
TENGA EN CUENTA:
+Las llamadas a la API de OpenAI solo son 'gratuitas' durante los primeros tres meses después de generar su cuenta de OpenAI (OpenAI proporciona un crédito de $5 que vence).
+Puede definir un límite de facturación mensual estricto (por ejemplo, $1) aquí.
+Vea sus precios y preguntas frecuentes aquí.
+Obtenga su clave API de OpenAI aquí.
Regresa: El texto generado (str).
+Antes de usar gget gpt por primera vez, corre gget setup gpt / gget.setup("gpt") (ver también gget setup).
Parámetros posicionales
+prompt
+Mensaje de entrada basado en el cual generar texto (str).
api_key
+Su clave API de OpenAI (str) (obtenga su clave API).
Parámetros optionales
+-m --model
+El nombre del algoritmo GPT que se usará para generar el texto (str). Por defecto: "gpt-3.5-turbo".
+See https://platform.openai.com/docs/models/gpt-4 for more information on the available models.
-temp --temperature
+Valor entre 0 y 2 que controla el nivel de aleatoriedad y creatividad en el texto generado (float).
+Los valores más altos resultan en un texto más creativo y variado. Por defecto: 1.
-tp --top_p
+Controla la diversidad del texto generado como alternativa al muestreo con --temperature (float).
+Los valores más altos resultan en un texto más diverso e inesperado. Por defecto: 1.
+Tenga en cuenta que OpenAI recomienda modificar --top_p o el parámetro --temperature, pero no ambas.
-s --stop
+Una secuencia de tokens para marcar el final del texto generado (str). Por defecto: None.
-mt --max_tokens
+Controla la longitud máxima del texto generado, en tokens (int). Por defecto: 200.
-pp --presence_penalty
+Número entre -2.0 y 2.0. Los valores más altos aumentan la probabilidad de que el modelo hable sobre temas nuevos (float). Por defecto: 0.
-fp --frequency_penalty
+Número entre -2.0 y 2.0. Los valores más altos reducen la probabilidad de que el modelo repita la misma línea palabra por palabra (float). Por defecto: 0.
-lb --logit_bias
+Un diccionario que especifica un sesgo hacia ciertos tokens en el texto generado (dict). Por defecto: None.
-o --out
+Ruta al archivo en el que se guardarán los resultados, p. ej. ruta/al/directorio/resultados.txt. Por defecto: salida estándar (STDOUT).
gget gpt "Cómo estás hoy GPT?" su_clave_api
+# Python
+print(gget.gpt("Cómo estás hoy GPT?", "su_clave_api"))
+ +
+++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Obtenga información detallada sobre genes y transcripciones de Ensembl, UniProt y NCBI utilizando sus IDs del tipo Ensembl.
+Regresa: Resultados en formato JSON (Terminal) o Dataframe/CSV (Python).
Parámetro posicional
+ens_ids
+Uno o más ID del tipo Ensembl.
Parámetros optionales
+-o --out
+Ruta al archivo en el que se guardarán los resultados, p. ej. ruta/al/directorio/resultados.csv (o .json). Por defecto: salida estándar (STDOUT).
+Para Python, usa save=True para guardar los resultados en el directorio de trabajo actual.
Banderas
+-n --ncbi
+DESACTIVA los resultados de NCBI.
+Para Python: ncbi=False evita la incluida de datos de NCBI (por defecto: True).
-u --uniprot
+DESACTIVA los resultados de UniProt.
+Para Python: uniprot=False evita la incluida de datos de UniProt (por defecto: True).
-pdb --pdb
+INCLUYE PDB IDs en los resultados (podría aumentar el tiempo de ejecución).
+Para Python: pdb=True incluye IDs de PDB en los resultados (por defecto: False).
-csv --csv
+Solo para la Terminal. Regresa los resultados en formato CSV.
+Para Python, usa json=True para regresar los resultados en formato JSON.
-q --quiet
+Solo para la Terminal. Impide la informacion de progreso de ser exhibida durante la corrida.
+Para Python, usa verbose=False para imipidir la informacion de progreso de ser exhibida durante la corrida.
wrap_text
+Solo para Python. wrap_text=True muestra los resultados con texto envuelto para facilitar la lectura (por defecto: False).
gget info ENSG00000034713 ENSG00000104853 ENSG00000170296
+# Python
+gget.info(["ENSG00000034713", "ENSG00000104853", "ENSG00000170296"])
+→ Regresa información detallada sobre cada ID de Ensembl ingresada:
+| uniprot_id | ncbi_gene_id | primary_gene_name | synonyms | protein_names | ensembl_description | uniprot_description | ncbi_description | biotype | canonical_transcript | ... | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ENSG00000034713 | P60520 | 11345 | GABARAPL2 | [ATG8, ATG8C, FLC3A, GABARAPL2, GATE-16, GATE16, GEF-2, GEF2] | Gamma-aminobutyric acid receptor-associated protein like 2 (GABA(A) receptor-associated protein-like 2)... | GABA type A receptor associated protein like 2 [Source:HGNC Symbol;Acc:HGNC:13291] | FUNCTION: Ubiquitin-like modifier involved in intra- Golgi traffic (By similarity). Modulates intra-Golgi transport through coupling between NSF activity and ... | Enables ubiquitin protein ligase binding activity. Involved in negative regulation of proteasomal protein catabolic process and protein... | protein_coding | ENST00000037243.7 | ... |
| . . . | . . . | . . . | . . . | . . . | . . . | . . . | . . . | . . . | . . . | . . . | ... |
++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Alinea múltiples secuencias de nucleótidos o aminoácidos usando el algoritmo Muscle5.
+Regresa: Salida estándar (STDOUT) en formato ClustalW o archivo de tipo 'aligned FASTA' (.afa).
Parámetro posicional
+fasta
+Lista de secuencias o ruta al archivo FASTA o .txt que contiene las secuencias de nucleótidos o aminoácidos que se van a alinear.
Parámetros optionales
+-o --out
+Ruta al archivo en el que se guardarán los resultados, p. ej. ruta/al/directorio/resultados.afa. Por defecto: salida estándar (STDOUT).
+Para Python, usa save=True para guardar los resultados en el directorio de trabajo actual.
Banderas
+-s5 --super5
+Alinea las secuencies usando el algoritmo Super5 en lugar del algoritmo Parallel Perturbed Probcons (PPP) para disminuir el tiempo y la memoria usada durante la corrida.
+Use para ingresos grandes (unos cientos secuencias).
-q --quiet
+Solo para la Terminal. Impide la informacion de progreso de ser exhibida durante la corrida.
+Para Python, usa verbose=False para imipidir la informacion de progreso de ser exhibida durante la corrida.
gget muscle MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS
+# Python
+gget.muscle(["MSSSSWLLLSLVAVTAAQSTIEEQAKTFLDKFNHEAEDLFYQSSLAS", "MSSSSWLLLSLVEVTAAQSTIEQQAKTFLDKFHEAEDLFYQSLLAS"])
+gget muscle fasta.fa
+# Python
+gget.muscle("fasta.fa")
+→ Regresa las secuencias alineadas con coloración ClustalW. (Para devolver un archivo FASTA alineado (.afa), use el argumento --out (o save=True en Python).) En este ejemplo, el archivo 'fasta.fa' incluye varias secuencias para alineación (por ejemplo, isoformas devueltas desde gget seq).

También puede ver archivos FASTA alineados devueltos por gget.muscle usando programas como alv:
# Python
+!pip install biopython
+!pip install alv
+from Bio import AlignIO
+import alv
+
+gget.muscle("fasta.fa", out="fasta_aligned.afa")
+msa = AlignIO.read("fasta_aligned.afa", "fasta")
+alv.view(msa)
+++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Recibe secuencias de nucleótidos y mutaciones (en anotación de mutación estándar) y devuelve versiones mutadas de las secuencias según las mutaciones proporcionadas.
+Formato de devolución: Guarda las secuencias mutadas en formato FASTA (o devuelve una lista que contiene las secuencias mutadas si out=None).
Este módulo fue coescrito por Joseph Rich.
+Parámetro posicional
+sequences
+Ruta al archivo FASTA que contiene las secuencias a mutar, por ejemplo, 'path/to/seqs.fa'.
+Los identificadores de secuencia que siguen al carácter '>' deben corresponder a los identificadores en la columna seq_ID de mutations.
+NOTA: Solo se utilizará la cadena que sigue al '>' hasta el primer espacio o punto como identificador de secuencia. -> Se ignorarán los números de versión de los IDs de Ensembl.
Ejemplo de formato del archivo FASTA:
+>seq1 (o ENSG00000106443)
+ACTGCGATAGACT
+>seq2
+AGATCGCTAG
+Alternativamente: Proporcione secuencia(s) como una cadena o lista, por ejemplo, 'AGCTAGCT'.
+Otros parámetros requeridos
+-m --mutations
+Ruta al archivo csv o tsv (por ejemplo, 'path/to/mutations.csv') o marco de datos (objeto DataFrame) que contiene información sobre las mutaciones en el siguiente formato (la columna 'notas' no es necesaria):
| mutation | mut_ID | seq_ID | notas |
|---|---|---|---|
| c.2C>T | mut1 | seq1 | -> Aplicar la mutación 1 a la secuencia 1 |
| c.9_13inv | mut2 | seq2 | -> Aplicar la mutación 2 a la secuencia 2 |
| c.9_13inv | mut2 | seq4 | -> Aplicar la mutación 2 a la secuencia 4 |
| c.9_13delinsAAT | mut3 | seq4 | -> Aplicar la mutación 3 a la secuencia 4 |
| ... | ... | ... |
'mutation' = Columna que contiene las mutaciones a realizar, escritas en anotación de mutación estándar
+'mut_ID' = Columna que contiene el identificador de cada mutación
+'seq_ID' = Columna que contiene los identificadores de las secuencias a mutar (deben corresponder a la cadena que sigue al carácter '>' en el archivo FASTA de 'sequences'; NO incluir espacios ni puntos)
Alternativamente: Mutación(es) de entrada como una cadena o lista, por ejemplo, 'c.2C>T'.
+Si se proporciona una lista, el número de mutaciones debe ser igual al número de secuencias de entrada.
+Para uso desde el terminal (bash): Encierre las anotaciones de mutación individuales entre comillas para evitar errores.
Parámetros opcionales
+-k --k
+Longitud de las secuencias que flanquean la mutación. Por defecto: 30.
+Si k > longitud total de la secuencia, se mantendrá toda la secuencia.
-mc --mut_column
+Nombre de la columna que contiene las mutaciones a realizar en mutations. Por defecto: 'mutation'.
-mic --mut_id_column
+Nombre de la columna que contiene los IDs de cada mutación en mutations. Por defecto: 'mut_ID'.
-sic --seq_id_column
+Nombre de la columna que contiene los IDs de las secuencias a mutar en mutations. Por defecto: 'seq_ID'.
-o --out
+Ruta al archivo FASTA de salida que contiene las secuencias mutadas, por ejemplo, 'path/to/output_fasta.fa'.
+Por defecto: None -> devuelve una lista de las secuencias mutadas a la salida estándar.
+Los identificadores (después del '>') de las secuencias mutadas en el FASTA de salida serán '>[seq_ID]_[mut_ID]'.
Flags
+-q --quiet
+Solo para Terminal. Impide la información de progreso de ser exhibida durante la ejecución del programa.
+Para Python, usa verbose=False para imipidir la información de progreso de ser exhibida durante la ejecución del programa.
gget mutate ATCGCTAAGCT -m 'c.4G>T'
+# Python
+gget.mutate("ATCGCTAAGCT", "c.4G>T")
+→ Devuelve ATCTCTAAGCT.
+Lista de secuencias con una mutación para cada secuencia proporcionada en una lista:
+gget mutate ATCGCTAAGCT TAGCTA -m 'c.4G>T' 'c.1_3inv' -o mut_fasta.fa
+# Python
+gget.mutate(["ATCGCTAAGCT", "TAGCTA"], ["c.4G>T", "c.1_3inv"], out="mut_fasta.fa")
+→ Guarda el archivo 'mut_fasta.fa' que contiene:
+>seq1_mut1
+ATCTCTAAGCT
+>seq2_mut2
+GATCTA
+Una mutación aplicada a varias secuencias con k ajustado:
+gget mutate ATCGCTAAGCT TAGCTA -m 'c.1_3inv' -k 3
+# Python
+gget.mutate(["ATCGCTAAGCT", "TAGCTA"], "c.1_3inv", k=3)
+→ Devuelve ['CTAGCT', 'GATCTA'].
+++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Obtenga la estructura o los metadatos de una proteína usando data de RCSB Protein Data Bank (PDB).
+Regresa: El archivo 'pdb' se regresa en formato PDB. Todos los demás datos se regresan en formato JSON.
Parámetro posicional
+pdb_id
+ID del tipo PDB, p. ej. '7S7U'.
Parámetros optionales
+-r --resource
+Define el tipo de información a regresar. Uno de los siguientes:
+'pdb': Regresa la estructura de la proteína en formato PDB (regresa por defecto).
+'entry': Regresa información sobre las estructuras PDB en el nivel superior de la organización de datos PDB jerárquicos.
+'pubmed': Regresa anotaciones de PubMed (datos integrados de PubMed) para la cita principal de un ID PDB.
+'assembly': Regresa información sobre estructuras PDB en el nivel de estructura cuaternaria.
+'branched_entity': Regresa la descripción de la entidad ramificada (defina el ID de la entidad como identifier).
+'nonpolymer_entity': Regresa datos de entidades no poliméricas (defina el ID de la entidad como identifier).
+'polymer_entity': Regresa datos de entidades poliméricas (defina el ID de la entidad como identifier).
+'uniprot': Regresa anotaciones UniProt para una entidad macromolecular (defina el ID de la entidad como identifier).
+'branched_entity_instance': Regresa la descripción de instancia de entidad ramificada (defina el ID de cadena como identifier).
+'polymer_entity_instance': Regresa datos de instancia de entidad polimérica (también conocida como cadena) (defina el ID de cadena como identifier).
+'nonpolymer_entity_instance': Regresa datos de instancia de entidad no polimérica (defina el ID de cadena como identifier).
-i --identifier
+Este parámetro se puede utilizar para definir el ID de ensamblaje, entidad o cadena (po defecto: None). Los IDs de ensamblaje/entidad son números (p. ej., 1) y los IDs de cadena son letras (p. ej., 'A').
-o --out
+Ruta al archivo en el que se guardarán los resultados, p. ej. ruta/al/directorio/7S7U.pdb (o 7S7U_entry.json). Por defecto: salida estándar (STDOUT).
+Para Python, usa save=True para guardar los resultados en el directorio de trabajo actual.
gget pdb 7S7U -o 7S7U.pdb
+# Python
+gget.pdb("7S7U", save=True)
+→ Guarda la estructura de 7S7U en formato PDB como '7S7U.pdb' en el directorio de trabajo actual.
+Encuentre estructuras cristalinas de PDB para un análisis comparativo de la estructura de proteínas:
+# Encuentre IDs de PDB asociados con un ID de Ensembl
+gget info ENSG00000130234
+
+# Alternativamente: como que muchas entradas en el PDB no tienen ID de Ensembl vinculados,
+# es probable que encuentre más entradas de PDB BLASTing la secuencia contra el PDB:
+
+# Obtenga la secuencia de aminoácidos
+gget seq --translate ENSG00000130234 -o gget_seq_results.fa
+
+# BLAST la secuencia de aminoácidos para encontrar estructuras similares en el PDB
+gget blast --database pdbaa gget_seq_results.fa
+
+# Obtenga archivos PDB de los IDs de PDB regresados por gget blast para un análisis comparativo
+gget pdb 7DQA -o 7DQA.pdb
+gget pdb 7CT5 -o 7CT5.pdb
+# Encuentre IDs de PDB asociados con un ID de Ensembl
+gget.info("ENSG00000130234")
+
+# Alternativamente: como que muchas entradas en el PDB no tienen ID de Ensembl vinculados,
+# es probable que encuentre más entradas de PDB BLASTing la secuencia contra el PDB:
+
+# Obtenga la secuencia de aminoácidos
+gget.seq("ENSG00000130234", translate=True, save=True)
+
+# BLAST la secuencia de aminoácidos para encontrar estructuras similares en el PDB
+gget.blast("gget_seq_results.fa", database="pdbaa")
+
+# Obtenga archivos PDB de los IDs de PDB regresados por gget blast para un análisis comparativo
+gget.pdb("7DQA", save=True)
+gget.pdb("7CT5", save=True)
+→ Este caso de uso ejemplifica cómo encontrar archivos PDB para un análisis comparativo de la estructura de las proteínas asociado con IDs de Ensembl o secuencias de aminoácidos. Los archivos PDB obtenidos también se pueden comparar con las estructuras predichas generadas por gget alphafold. Los archivos PDB se pueden ver de forma interactiva en 3D aquí, o usando programas como PyMOL o Blender. Múltiple archivos PDB se pueden visualizar para comparación aquí.
++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Obtenga enlaces FTP y sus respectivos metadatos (o use la bandera ftp para regresar solo los enlaces) para referenciar genomas y anotaciones de Ensembl.
+Regresa: Resultados en formato JSON.
Parámetro posicional
+species
+La especie por la cual que se buscará los FTP en el formato género_especies, p. ej. homo_sapiens.
+Nota: No se requiere cuando se llama a la bandera --list_species.
+Accesos directos: 'human', 'mouse', 'human_grch37' (accede al ensamblaje del genoma GRCh37)
Parámetros optionales
+-w --which
+Define qué resultados devolver. Por defecto: 'all' -> Regresa todos los resultados disponibles.
+Las entradas posibles son uno solo o una combinación de las siguientes (como lista separada por comas):
+'gtf' - Regresa la anotación (GTF).
+'cdna' - Regresa el transcriptoma (cDNA).
+'dna' - Regresa el genoma (DNA).
+'cds' - Regresa las secuencias codificantes correspondientes a los genes Ensembl. (No contiene UTR ni secuencia intrónica).
+'cdrna' - Regresa secuencias de transcripción correspondientes a genes de ARN no codificantes (ncRNA).
+'pep' - Regresa las traducciones de proteínas de los genes Ensembl.
-r --release
+Define el número de versión de Ensembl desde el que se obtienen los archivos, p. ej. 104. Default: latest Ensembl release.
-o --out
+Ruta al archivo en el que se guardarán los resultados, p. ej. ruta/al/directorio/resultados.json. Por defecto: salida estándar (STDOUT).
+Para Python, usa save=True para guardar los resultados en el directorio de trabajo actual.
Banderas
+-l --list_species
+Enumera todas las especies disponibles. (Para Python: combina con species=None.)
-ftp --ftp
+Regresa solo los enlaces FTP solicitados.
-d --download
+Solo para Terminal. Descarga los FTP solicitados al directorio actual (requiere curl para ser instalado).
-q --quiet
+Solo para la Terminal. Impide la informacion de progreso de ser exhibida durante la corrida.
+Para Python, usa verbose=False para imipidir la informacion de progreso de ser exhibida durante la corrida.
Use gget ref en combinación con kallisto | bustools para construir un índice de referencia:
kb ref -i INDEX -g T2G -f1 FASTA $(gget ref --ftp -w dna,gtf homo_sapiens)
+→ kb ref crea un índice de referencia utilizando los últimos archivos de ADN y GTF de especies Homo sapiens que le ha pasado gget ref.
Enumere todos los genomas disponibles de la versión 103 de Ensembl:
+gget ref --list_species -r 103
+# Python
+gget.ref(species=None, list_species=True, release=103)
+→ Regresa una lista con todos los genomas disponibles (gget ref verifica si GTF y FASTA están disponibles) de la versión 103 de Ensembl.
+(Si no se especifica ninguna versión, gget ref siempre devolverá información de la última versión de Ensembl).
Obtenga la referencia del genoma para una especie específica:
+gget ref -w gtf,dna homo_sapiens
+# Python
+gget.ref("homo_sapiens", which=["gtf", "dna"])
+→ Regresa un JSON con los últimos FTP humanos GTF y FASTA, y sus respectivos metadatos, en el formato:
+{
+ "homo_sapiens": {
+ "annotation_gtf": {
+ "ftp": "http://ftp.ensembl.org/pub/release-106/gtf/homo_sapiens/Homo_sapiens.GRCh38.106.gtf.gz",
+ "ensembl_release": 106,
+ "release_date": "28-Feb-2022",
+ "release_time": "23:27",
+ "bytes": "51379459"
+ },
+ "genome_dna": {
+ "ftp": "http://ftp.ensembl.org/pub/release-106/fasta/homo_sapiens/dna/Homo_sapiens.GRCh38.dna.primary_assembly.fa.gz",
+ "ensembl_release": 106,
+ "release_date": "21-Feb-2022",
+ "release_time": "09:35",
+ "bytes": "881211416"
+ }
+ }
+}
+++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Obtenga genes y transcripciones de Ensembl usando términos de búsqueda de forma libre.
+Los resultados se comparan según las secciones "nombre del gen" y "descripción" en la base de datos de Ensembl. gget versión >= 0.27.9 también incluye resultados que coinciden con la sección "sinónimo" de Ensembl.
+Regresa: Resultados en formato JSON (Terminal) o Dataframe/CSV (Python).
Parámetro posicional
+searchwords
+Una o más palabras de búsqueda de forma libre, p. ej. gaba nmda. (Nota: la búsqueda no distingue entre mayúsculas y minúsculas).
Otros parámetros requeridos
+-s --species
+Especies o base de datos a buscar.
+Una especie se puede pasar en el formato 'género_especie', p. ej. 'homo_sapiens' o 'arabidopsis_thaliana'.
+Para pasar una base de datos específica, pase el nombre de la base de datos CORE, p. ej. 'mus_musculus_dba2j_core_105_1'.
Todas las bases de datos disponibles para cada versión de Ensembl se pueden encontrar aquí:
+Vertebrados: http://ftp.ensembl.org/pub/current/mysql/
+Invertebrados: http://ftp.ensemblgenomes.org/pub/current/ + selecciona reino animal + selecciona mysql/
Accesos directos: 'human', 'mouse'
+Parámetros optionales
+-r --release
+Define el número de versión de Ensembl desde el que se obtienen los archivos, p. ej. 104. Por defecto: None -> se usa la última versión de Ensembl.
Nota: No se aplica a las especies invertebrados (en su lugar, puede pasar una base de datos de una especies específica (incluyen un número de versión) al argumento species). Para especies de invertebrados, Ensembl solo almacena bases de datos de 10 versiones anteriores a la versión actual.
Este argumento se sobrescribe si se pasa una base de datos específica (que incluye un número de publicación) al argumento species.
-t --id_type
+'gene' (esto se use por defecto) o 'transcript'
+Regesa genes o transcripciones, respectivamente.
-ao --andor
+'or' (esto se use por defecto) o 'and'
+'or' ('o'): Regresa todos los genes que INCLUYEN AL MENOS UNA de las palabras de búsqueda en su nombre/descripción.
+'and' ('y'): Regresa solo los genes que INCLUYEN TODAS las palabras de búsqueda en su nombre/descripción.
-l --limit
+Limita el número de resultados de búsqueda, p. ej. 10. Por defecto: None.
-o --out
+Ruta al archivo en el que se guardarán los resultados, p. ej. ruta/al/directorio/resultados.csv (o .json). Por defecto: salida estándar (STDOUT).
+Para Python, usa save=True para guardar los resultados en el directorio de trabajo actual.
Banderas
+-csv --csv
+Solo para la Terminal. Regresa los resultados en formato CSV.
+Para Python, usa json=True para regresar los resultados en formato JSON.
-q --quiet
+Solo para la Terminal. Impide la informacion de progreso de ser exhibida durante la corrida.
+Para Python, usa verbose=False para imipidir la informacion de progreso de ser exhibida durante la corrida.
wrap_text
+Solo para Python. wrap_text=True muestra los resultados con texto envuelto para facilitar la lectura (por defecto: False).
gget search -s human gaba gamma-aminobutyric
+# Python
+gget.search(["gaba", "gamma-aminobutyric"], "homo_sapiens")
+→ Regresa todos los genes que contienen al menos una de las palabras de búsqueda en su nombre o descripción de Ensembl/referencia externa:
+| ensembl_id | gene_name | ensembl_description | ext_ref_description | biotype | url |
|---|---|---|---|---|---|
| ENSG00000034713 | GABARAPL2 | GABA type A receptor associated protein like 2 [Source:HGNC Symbol;Acc:HGNC:13291] | GABA type A receptor associated protein like 2 | protein_coding | https://uswest.ensembl.org/homo_sapiens/Gene/Summary?g=ENSG00000034713 |
| . . . | . . . | . . . | . . . | . . . | . . . |
++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Función para instalar/descargar dependencias de terceros para un módulo de gget.
+Parámetro posicional
+module
+Módulo gget para el que se deben instalar las dependencias.
gget setup alphafold
+# Python
+gget.setup("alphafold")
+→ Instala todas las dependencias de terceros (modificadas) y descarga los parámetros del algoritmo (~4 GB) necesarios para ejecutar gget alphafold.
++Parámetros de Python són iguales a los parámetros largos (
+--parámetro) de Terminal, si no especificado de otra manera. Banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
Obtenga la(s) secuencia(s) nucleótidos o aminoácidos de un gen (y todas sus isoformas) con su ID de Ensembl.
+Regresa: Archivo de tipo FASTA.
Parámetro posicional
+ens_ids
+One or more Ensembl IDs.
Parámetros optionales
+-o --out
+Ruta al archivo en el que se guardarán los resultados, p. ruta/al/directorio/resultados.fa. Por defecto: salida estándar (STDOUT).
+Para Python, usa save=True para guardar los resultados en el directorio de trabajo actual.
Banderas
+-t --translate
+Regresa secuencias de aminoácidos (en lugar de nucleótidos).
+Las secuencias de nucleótidos se obtienen de Ensembl.
+Las secuencias de aminoácidos se obtienen de UniProt.
-iso --isoforms
+Regresa las secuencias de todas las transcripciones conocidas.
+(Solo para IDs de genes).
-q --quiet
+Solo para la Terminal. Impide la informacion de progreso de ser exhibida durante la corrida.
+Para Python, usa verbose=False para imipidir la informacion de progreso de ser exhibida durante la corrida.
gget seq ENSG00000034713 ENSG00000104853 ENSG00000170296
+# Python
+gget.seq(["ENSG00000034713", "ENSG00000104853", "ENSG00000170296"])
+→ Regresa las secuencias de nucleótidos de ENSG00000034713, ENSG00000104853, y ENSG00000170296 en formato FASTA.
+gget seq -t -iso ENSG00000034713
+# Python
+gget.seq("ENSG00000034713", translate=True, isoforms=True)
+→ Regresa las secuencias de aminoácidos de todas las transcripciones conocidas de ENSG00000034713 en formato FASTA.
+¡Gracias por invertir su tiempo en contribuir con nuestro proyecto! Cualquier contribución que hagas se verá reflejada en el repositorio de GitHub de gget. ✨
+Lea nuestro Código de conducta para mantener nuestra comunidad accesible y respetable.
+En esta guía, obtendrá una descripción general del flujo de trabajo de contribución desde la creación de un GitHub Issue (asunto) o la creación de un GitHub Pull Request (PR) hasta la revisión y fusión de un PR.
+Si detecta un problema con gget o tiene una idea para una nueva función, comproba si ya existe un Issue para este problema/sugerencia. Si no existe un Issue relacionado, puede abrir un nuevo Issue utilizando el formulario correspondiente.
+Explore nuestros Issues existentes para encontrar uno que le interese. Puede restringir la búsqueda utilizando "labels" como filtros. Si encuentra un Issue en el que desea trabajar, puede abrir un PR con una solución.
+Usando GitHub Desktop:
+Usando la Terminal:
+Confirme sus cambios una vez que esté satisfecho con ellos.
+pip install -r dev-requirements.txtcoverage run -m pytest -ra -v tests && coverage report --omit=main.py,tests*Si tiene alguna pregunta, no dude en iniciar una discusión o crear un Issue como se describe anteriormente.
+Cuando haya terminado con los cambios, cree un Pull Request, también conocido como "PR".
+‼️ Realice todos los PRs contra la rama dev del repositorio gget
Una vez que envíe su PR, un miembro del equipo gget revisará su propuesta. Podemos hacer preguntas o solicitar información adicional.
+¡Felicidades! 🎉 El equipo de gget te lo agradece. ✨
+Una vez que su PR se fusione, sus contribuciones serán visibles públicamente en el repositorio de gget.
+Nosotros, como miembros, contribuyentes y administradores nos comprometemos a hacer de la participación en nuestra comunidad sea una experiencia libre de acoso para todo el mundo, independientemente de la edad, dimensión corporal, discapacidad visible o invisible, etnicidad, características sexuales, identidad y expresión de género, nivel de experiencia, educación, nivel socio-económico, nacionalidad, apariencia personal, raza, casta, color, religión, o identidad u orientación sexual.
+Nos comprometemos a actuar e interactuar de maneras que contribuyan a una comunidad abierta, acogedora, diversa, inclusiva y sana.
+Ejemplos de comportamientos que contribuyen a crear un ambiente positivo para nuestra comunidad:
+Ejemplos de comportamiento inaceptable:
+Los administradores de la comunidad son responsables de aclarar y hacer cumplir nuestros estándares de comportamiento aceptable y tomarán acciones apropiadas y correctivas de forma justa en respuesta a cualquier comportamiento que consideren inapropiado, amenazante, ofensivo o dañino.
+Los administradores de la comunidad tendrán el derecho y la responsabilidad de eliminar, editar o rechazar comentarios, commits, código, ediciones de páginas de wiki, issues y otras contribuciones que no se alineen con este Código de Conducta, y comunicarán las razones para sus decisiones de moderación cuando sea apropiado.
+Este código de conducta aplica tanto a espacios del proyecto como a espacios públicos donde un individuo esté en representación del proyecto o comunidad. Ejemplos de esto incluyen el uso de la cuenta oficial de correo electrónico, publicaciones a través de las redes sociales oficiales, o presentaciones con personas designadas en eventos en línea o no.
+Instancias de comportamiento abusivo, acosador o inaceptable de otro modo podrán ser reportadas a los administradores de la comunidad responsables del cumplimiento a través de lpachter@caltech.edu. Todas las quejas serán evaluadas e investigadas de una manera puntual y justa.
+Todos los administradores de la comunidad están obligados a respetar la privacidad y la seguridad de quienes reporten incidentes.
+Los administradores de la comunidad seguirán estas Guías de Impacto en la Comunidad para determinar las consecuencias de cualquier acción que juzguen como un incumplimiento de este Código de Conducta:
+Impacto en la Comunidad: El uso de lenguaje inapropiado u otro comportamiento considerado no profesional o no acogedor en la comunidad.
+Consecuencia: Un aviso escrito y privado por parte de los administradores de la comunidad, proporcionando claridad alrededor de la naturaleza de este incumplimiento y una explicación de por qué el comportamiento es inaceptable. Una disculpa pública podría ser solicitada.
+Impacto en la Comunidad: Un incumplimiento causado por un único incidente o por una cadena de acciones.
+Consecuencia: Un aviso con consecuencias por comportamiento prolongado. No se interactúa con las personas involucradas, incluyendo interacción no solicitada con quienes se encuentran aplicando el Código de Conducta, por un periodo especificado de tiempo. Esto incluye evitar las interacciones en espacios de la comunidad, así como a través de canales externos como las redes sociales. Incumplir estos términos puede conducir a una expulsión temporal o permanente.
+Impacto en la Comunidad: Una serie de incumplimientos de los estándares de la comunidad, incluyendo comportamiento inapropiado continuo.
+Consecuencia: Una expulsión temporal de cualquier forma de interacción o comunicación pública con la comunidad durante un intervalo de tiempo especificado. No se permite interactuar de manera pública o privada con las personas involucradas, incluyendo interacciones no solicitadas con quienes se encuentran aplicando el Código de Conducta, durante este periodo. Incumplir estos términos puede conducir a una expulsión permanente.
+Impacto en la Comunidad: Demostrar un patrón sistemático de incumplimientos de los estándares de la comunidad, incluyendo conductas inapropiadas prolongadas en el tiempo, acoso de individuos, o agresiones o menosprecio a grupos de individuos.
+Consecuencia: Una expulsión permanente de cualquier tipo de interacción pública con la comunidad del proyecto.
+Este Código de Conducta es una adaptación del Contributor Covenant, versión 2.1, +disponible en https://www.contributor-covenant.org/es/version/2/1/code_of_conduct.html
+Las Guías de Impacto en la Comunidad están inspiradas en la escalera de aplicación del código de conducta de Mozilla.
+Para respuestas a las preguntas frecuentes de este código de conducta, consulta las FAQ en +https://www.contributor-covenant.org/faq. Hay traducciones disponibles en https://www.contributor-covenant.org/translationshttps://www.contributor-covenant.org/translations
+ +Si utiliza gget en una publicación, favor de citar:
+Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
Si utiliza gget alphafold, favor de citar también:
Y si aplica:
+Si utiliza gget archs4, favor de citar también:
Lachmann A, Torre D, Keenan AB, Jagodnik KM, Lee HJ, Wang L, Silverstein MC, Ma’ayan A. Massive mining of publicly available RNA-seq data from human and mouse. Nature Communications 9. Article number: 1366 (2018), doi:10.1038/s41467-018-03751-6
+Bray NL, Pimentel H, Melsted P and Pachter L, Near optimal probabilistic RNA-seq quantification, Nature Biotechnology 34, p 525--527 (2016). https://doi.org/10.1038/nbt.3519
+Si utiliza gget bgee, favor de citar también:
Si utiliza gget blast, favor de citar también:
Si utiliza gget blat, favor de citar también:
Si utiliza gget cbio, favor de citar también:
Cerami E, Gao J, Dogrusoz U, Gross BE, Sumer SO, Aksoy BA, Jacobsen A, Byrne CJ, Heuer ML, Larsson E, Antipin Y, Reva B, Goldberg AP, Sander C, Schultz N. The cBio cancer genomics portal: an open platform for exploring multidimensional cancer genomics data. Cancer Discov. 2012 May;2(5):401-4. doi: 10.1158/2159-8290.CD-12-0095. Erratum in: Cancer Discov. 2012 Oct;2(10):960. PMID: 22588877; PMCID: PMC3956037.
+Gao J, Aksoy BA, Dogrusoz U, Dresdner G, Gross B, Sumer SO, Sun Y, Jacobsen A, Sinha R, Larsson E, Cerami E, Sander C, Schultz N. Integrative analysis of complex cancer genomics and clinical profiles using the cBioPortal. Sci Signal. 2013 Apr 2;6(269):pl1. doi: 10.1126/scisignal.2004088. PMID: 23550210; PMCID: PMC4160307.
+de Bruijn I, Kundra R, Mastrogiacomo B, Tran TN, Sikina L, Mazor T, Li X, Ochoa A, Zhao G, Lai B, Abeshouse A, Baiceanu D, Ciftci E, Dogrusoz U, Dufilie A, Erkoc Z, Garcia Lara E, Fu Z, Gross B, Haynes C, Heath A, Higgins D, Jagannathan P, Kalletla K, Kumari P, Lindsay J, Lisman A, Leenknegt B, Lukasse P, Madela D, Madupuri R, van Nierop P, Plantalech O, Quach J, Resnick AC, Rodenburg SYA, Satravada BA, Schaeffer F, Sheridan R, Singh J, Sirohi R, Sumer SO, van Hagen S, Wang A, Wilson M, Zhang H, Zhu K, Rusk N, Brown S, Lavery JA, Panageas KS, Rudolph JE, LeNoue-Newton ML, Warner JL, Guo X, Hunter-Zinck H, Yu TV, Pilai S, Nichols C, Gardos SM, Philip J; AACR Project GENIE BPC Core Team, AACR Project GENIE Consortium; Kehl KL, Riely GJ, Schrag D, Lee J, Fiandalo MV, Sweeney SM, Pugh TJ, Sander C, Cerami E, Gao J, Schultz N. Analysis and Visualization of Longitudinal Genomic and Clinical Data from the AACR Project GENIE Biopharma Collaborative in cBioPortal. Cancer Res. 2023 Dec 1;83(23):3861-3867. doi: 10.1158/0008-5472.CAN-23-0816. PMID: 37668528; PMCID: PMC10690089.
+Please also cite the source of the data if you are using a publicly available dataset.
+Si utiliza gget cellxgene, favor de citar también:
Si utiliza gget cosmic, favor de citar también:
Si utiliza gget diamond, favor de citar también:
Si utiliza gget elm, favor de citar también:
Laura Luebbert, Chi Hoang, Manjeet Kumar, Lior Pachter, Fast and scalable querying of eukaryotic linear motifs with gget elm, Bioinformatics, 2024, btae095, https://doi.org/10.1093/bioinformatics/btae095
+Manjeet Kumar, Sushama Michael, Jesús Alvarado-Valverde, Bálint Mészáros, Hugo Sámano‐Sánchez, András Zeke, Laszlo Dobson, Tamas Lazar, Mihkel Örd, Anurag Nagpal, Nazanin Farahi, Melanie Käser, Ramya Kraleti, Norman E Davey, Rita Pancsa, Lucía B Chemes, Toby J Gibson, The Eukaryotic Linear Motif resource: 2022 release, Nucleic Acids Research, Volume 50, Issue D1, 7 January 2022, Pages D497–D508, https://doi.org/10.1093/nar/gkab975
+Si utiliza gget enrichr, favor de citar también:
Chen EY, Tan CM, Kou Y, Duan Q, Wang Z, Meirelles GV, Clark NR, Ma'ayan A. Enrichr: interactive and collaborative HTML5 gene list enrichment analysis tool. BMC Bioinformatics. 2013; 128(14). https://doi.org/10.1186/1471-2105-14-128
+Kuleshov MV, Jones MR, Rouillard AD, Fernandez NF, Duan Q, Wang Z, Koplev S, Jenkins SL, Jagodnik KM, Lachmann A, McDermott MG, Monteiro CD, Gundersen GW, Ma'ayan A. Enrichr: a comprehensive gene set enrichment analysis web server 2016 update. Nucleic Acids Research. 2016; gkw377. doi: 10.1093/nar/gkw377
+Xie Z, Bailey A, Kuleshov MV, Clarke DJB., Evangelista JE, Jenkins SL, Lachmann A, Wojciechowicz ML, Kropiwnicki E, Jagodnik KM, Jeon M, & Ma’ayan A. Gene set knowledge discovery with Enrichr. Current Protocols, 1, e90. 2021. doi: 10.1002/cpz1.90.
+Si trabaja con conjuntos de datos no humanos/ratón, cite también:
+Si utiliza gget info, favor de citar también:
Martin FJ, Amode MR, Aneja A, Austine-Orimoloye O, Azov AG, Barnes I, Becker A, Bennett R, Berry A, Bhai J, Bhurji SK, Bignell A, Boddu S, Branco Lins PR, Brooks L, Ramaraju SB, Charkhchi M, Cockburn A, Da Rin Fiorretto L, Davidson C, Dodiya K, Donaldson S, El Houdaigui B, El Naboulsi T, Fatima R, Giron CG, Genez T, Ghattaoraya GS, Martinez JG, Guijarro C, Hardy M, Hollis Z, Hourlier T, Hunt T, Kay M, Kaykala V, Le T, Lemos D, Marques-Coelho D, Marugán JC, Merino GA, Mirabueno LP, Mushtaq A, Hossain SN, Ogeh DN, Sakthivel MP, Parker A, Perry M, Piližota I, Prosovetskaia I, Pérez-Silva JG, Salam AIA, Saraiva-Agostinho N, Schuilenburg H, Sheppard D, Sinha S, Sipos B, Stark W, Steed E, Sukumaran R, Sumathipala D, Suner MM, Surapaneni L, Sutinen K, Szpak M, Tricomi FF, Urbina-Gómez D, Veidenberg A, Walsh TA, Walts B, Wass E, Willhoft N, Allen J, Alvarez-Jarreta J, Chakiachvili M, Flint B, Giorgetti S, Haggerty L, Ilsley GR, Loveland JE, Moore B, Mudge JM, Tate J, Thybert D, Trevanion SJ, Winterbottom A, Frankish A, Hunt SE, Ruffier M, Cunningham F, Dyer S, Finn RD, Howe KL, Harrison PW, Yates AD, Flicek P. Ensembl 2023. Nucleic Acids Res. 2023 Jan 6;51(D1):D933-D941. doi: 10.1093/nar/gkac958. PMID: 36318249; PMCID: PMC9825606.
+Sayers EW, Beck J, Bolton EE, Brister JR, Chan J, Comeau DC, Connor R, DiCuccio M, Farrell CM, Feldgarden M, Fine AM, Funk K, Hatcher E, Hoeppner M, Kane M, Kannan S, Katz KS, Kelly C, Klimke W, Kim S, Kimchi A, Landrum M, Lathrop S, Lu Z, Malheiro A, Marchler-Bauer A, Murphy TD, Phan L, Prasad AB, Pujar S, Sawyer A, Schmieder E, Schneider VA, Schoch CL, Sharma S, Thibaud-Nissen F, Trawick BW, Venkatapathi T, Wang J, Pruitt KD, Sherry ST. Database resources of the National Center for Biotechnology Information. Nucleic Acids Res. 2024 Jan 5;52(D1):D33-D43. doi: 10.1093/nar/gkad1044. PMID: 37994677; PMCID: PMC10767890.
+The UniProt Consortium , UniProt: the Universal Protein Knowledgebase in 2023, Nucleic Acids Research, Volume 51, Issue D1, 6 January 2023, Pages D523–D531, https://doi.org/10.1093/nar/gkac1052
+Si utiliza gget muscle, favor de citar también:
Si utiliza gget opentargets, favor de citar también:
Si utiliza gget pdb, favor de citar también:
Si utiliza gget ref o gget search, favor de citar también:
Si utiliza gget seq, favor de citar también:
Martin FJ, Amode MR, Aneja A, Austine-Orimoloye O, Azov AG, Barnes I, Becker A, Bennett R, Berry A, Bhai J, Bhurji SK, Bignell A, Boddu S, Branco Lins PR, Brooks L, Ramaraju SB, Charkhchi M, Cockburn A, Da Rin Fiorretto L, Davidson C, Dodiya K, Donaldson S, El Houdaigui B, El Naboulsi T, Fatima R, Giron CG, Genez T, Ghattaoraya GS, Martinez JG, Guijarro C, Hardy M, Hollis Z, Hourlier T, Hunt T, Kay M, Kaykala V, Le T, Lemos D, Marques-Coelho D, Marugán JC, Merino GA, Mirabueno LP, Mushtaq A, Hossain SN, Ogeh DN, Sakthivel MP, Parker A, Perry M, Piližota I, Prosovetskaia I, Pérez-Silva JG, Salam AIA, Saraiva-Agostinho N, Schuilenburg H, Sheppard D, Sinha S, Sipos B, Stark W, Steed E, Sukumaran R, Sumathipala D, Suner MM, Surapaneni L, Sutinen K, Szpak M, Tricomi FF, Urbina-Gómez D, Veidenberg A, Walsh TA, Walts B, Wass E, Willhoft N, Allen J, Alvarez-Jarreta J, Chakiachvili M, Flint B, Giorgetti S, Haggerty L, Ilsley GR, Loveland JE, Moore B, Mudge JM, Tate J, Thybert D, Trevanion SJ, Winterbottom A, Frankish A, Hunt SE, Ruffier M, Cunningham F, Dyer S, Finn RD, Howe KL, Harrison PW, Yates AD, Flicek P. Ensembl 2023. Nucleic Acids Res. 2023 Jan 6;51(D1):D933-D941. doi: 10.1093/nar/gkac958. PMID: 36318249; PMCID: PMC9825606.
+The UniProt Consortium , UniProt: the Universal Protein Knowledgebase in 2023, Nucleic Acids Research, Volume 51, Issue D1, 6 January 2023, Pages D523–D531, https://doi.org/10.1093/nar/gkac1052
+gget es tan preciso como la base de datos/servidores/APIs que utiliza. La exactitud o fiabilidad de los datos no es garantizada por ningún motivo. Los proveedores por ningún motivo seran responsables de (incluyendo, sin limite alguno) la calidad, ejecución, o comerciabilidad para cualquier propósito particular surgiendo del uso o la incapacidad de usar los datos.